2. 上海交通大学 农业与生物学院,上海 200240;
3. 上海市疾病预防控制中心,上海 200336;
4. 上海科玛嘉微生物技术中心,上海 200434
2. College of Agriculture and Biology, Shanghai Jiaotong University, Shanghai 200240, China;
3. Shanghai Municipal Center for Disease Control and Prevention, Shanghai 200336, China;
4. Shanghai Municipal Ke-Ma-Jia Technology Center for Microbiology, Shanghai 200434, China
沙门菌Salmonella是世界范围内重要的人畜共患病原菌,其在自然界分布广泛,种类繁多,目前已发现超过2 500个血清型[1].2006—2010年上海地区腹泻病人中分离到的山夫登堡沙门菌Salmonella Senftenberg位列所分离肠道沙门菌的第3位[2].上海疾病控制与预防中心按照全球沙门菌监测方案进行连续性腹泻病例监测,其数据显示2006年的山夫登堡病例符合沙门菌性相对散发和集中暴发的分子流行特征[3].本研究于2008—2012年在上海地区各农贸市场的动物源性食品中分离到15株山夫登堡沙门菌,并对其进行了药物敏感性分析和脉冲场凝胶电泳技术(Pulse field gel electrophoresis,PFGE)分子分型的相关性研究,以对其流行态势进行合理评估,为预防和控制该病危害提出合理化数据支持和理论依据.
1 材料与方法 1.1 材料 1.1.1 样品来源15株山夫登堡沙门菌于2008—2012年先后分离自上海市部分农贸市场内动物源食品中;药敏试验质控菌株大肠埃希菌Escherichia coli ATCC25922、ATCC35218,PFGE分型用标准菌株H9812,由上海交通大学农业与生物学院食品微生物实验室保存.
1.1.2 主要试剂预增菌、增菌及分离纯化沙门菌所用培养基均购自上海科玛嘉科技有限公司;药敏试验抗生素为Sigma公司产品;Xba I限制性内切酶为TaKaRa公司产品;蛋白酶K(Proteinase K)为Merck公司产品;PFGE专用琼脂糖Semkem Gold为Cambrex Bio Science Rockland公司产品;沙门分型诊断血清(SSI丹麦);细菌微量生化反应管(杭州微生物试剂有限公司).
1.2 方法 1.2.1 分离鉴定参考许学斌等[4]的文献方法对上海部分农贸市场的动物源性食品进行沙门菌分离、菌株初筛鉴定;系统生化反应符合沙门菌,再根据Kauffmann-White法测定其血清分型,血清抗原式符合[3,19:g,s,t:-]者鉴定为山夫登堡沙门菌.
1.2.2 药敏试验选用常见的16种抗生素,包括氨苄西林、阿莫西林/克拉维酸(质量比2:1)、头孢曲松、头孢噻呋、氯霉素、萘啶酸、环丙沙星、氧氟沙星、左氧氟沙星、阿米卡星、庆大霉素、卡那霉素、链霉素、四环素、磺胺异恶唑、甲氧苄啶/磺胺甲恶唑(质量比1:19).质控菌株为大肠埃希菌ATCC25922、ATCC35218.按照美国临床实验室标准化委员会(Clinical and Laboratory Standards Institute,CLSI)推荐的琼脂稀释法进行药敏性测定,按照CLSI的规范进行测定结果判读[5],链霉素的耐药折点参考文献[6].
1.2.3 PFGE分子分型方法参考美国CDC的PulseNet网络实验室推荐的沙门菌PFGE分型标准方法操作[7].将标准菌株H9812与试验菌分别用PFGE专用琼脂糖Semkem Gold固定、裂解、洗涤后,包埋DNA在37 ℃水浴中,使用50 U的Xba I至少酶切2 h.得到的酶切后限制性DNA片段在0.01 kg/L的Semkem Gold琼脂糖凝胶中使用Chef Mapper脉冲场凝胶电泳分型,电泳缓冲液为0.5×TBE,电泳时间为19 h,电泳温度为14 ℃,起始与最终转换时间分别设置为2.16、63.8 s.凝胶使用Gelrad染色及纯水脱色后成像,PFGE成像后的DNA图谱用Bionumerics5.1软件进行聚类分析,导出亲缘关系树状图谱.
2 结果与分析 2.1 分离鉴定结果根据GB4789-2010标准的生化反应表,其中14株菌株硫化氢(H2S)阳性、靛基质阴性、pH7.2尿素阴性、氰化钾阴性、赖氨酸脱羧酶阳性,为沙门菌典型反应.另外1株(SH09SF118)H2S呈阴性,补做ONPG试验呈阴性,为不典型沙门菌.经血清学确认后,分离到山夫登堡沙门菌15株.其中,5株来自禽肉制品,5株来自猪肉制品或其加工品,4株来自水产品,1株来自牛肉制品.
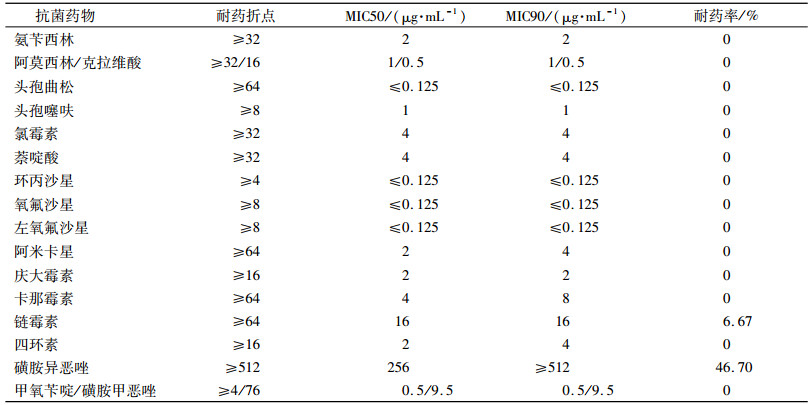
2.2 药敏试验结果药敏试验结果(表 1)显示:15株山夫登堡沙门菌对磺胺异恶唑耐药率最高,为46.7%,除1株对链霉素耐药,所有菌株对其他抗生素均不耐药.将16种抗生素的MIC50及MIC90进行比较,发现磺胺异恶唑的MIC50及MIC90最高,分别为256、≥512 μg/mL;另外,尽管只发现1株对链霉素耐药的菌株,但是其MIC50及MIC90分别高达16 μg/mL;头孢曲松以及环丙沙星、氧氟沙星、左氧氟沙星的MIC50及MIC90都≤0.125 μg/mL.
|
|
表 1 15株山夫登堡沙门菌药敏试验结果 Table 1 Antimicrobial susceptibility of 15 strains of Salmonella Senftenberg |
PFGE聚类分析的结果见图 1. 15株沙门菌可分为10个PFGE型别,即X1~X10;X4、X7分别包括2株禽肉制品来源的菌株,X5、X9分别包括2株猪肉制品源的菌株.X3包括1株水产品来源以及1株猪肉蛋饺来源的菌株.按相似度≥85%划分,X3、X4、X5又可划分到同一簇.

|
图 1 15株山夫登堡沙门菌PFGE基因型聚类结果 Figure 1 PFGE genotype cluster analysis of 15 strains of Salmonella Senftenberg |
食物传播是引起人感染沙门菌的主要途径.周晓红等[8]对我国杭州地区食品、公共场所从业人员健康体检中山夫登堡沙门菌监测结果表明,食品相关行业的人员极有可能成为该种细菌的隐性传染源.Dong等[9]的研究显示在牛肉加工厂分离到的山夫登堡高居所有沙门菌血清型的第2位.这就要求我们进一步做好食品来源中山夫登堡沙门菌的耐药及分子分型的监测.
本研究共分离到15株山夫登堡沙门菌,其中有5株分离自禽肉制品,5株分离自猪肉制品及其加工品,有研究表明这是我国沙门菌分离率最高的两种零售肉类型[10];1株分离自牛肉源,还有4株分离自水产品,其中1株为硫化氢阴性沙门菌,而2006年上海市山夫登堡的多点暴发病例中12株属于硫化氢阴性[11],二者可能存在一定的流行相关性.
抗菌药物耐药性分析表明,这15株山夫登堡沙门菌对磺胺异恶唑的耐药性最高,达到46.7%,这与我国上海地区分离到的人源沙门菌对该抗生素的耐药率(47.9%)相似[2].15株细菌对其他抗生素十分敏感,除1株细菌对链霉素耐药,对另外14种抗生素均无耐药性,表明相对于鼠伤寒、肠炎沙门菌等常见血清型,上海地区山夫登堡沙门菌的整体耐药情况不高.然而随着我国沙门菌整体耐药情况的逐年严重,同时鉴于山夫登堡沙门菌在近年造成过暴发流行,监测以及评估这一血清型的流行态势及耐药趋势对于防控这一疾病具有重要的意义.
在食源性致病菌监测项目中,食品中污染细菌的分型与溯源一直是问题的核心.PFGE以其分辨力强,重复性好的特点,被称为细菌分子生物学分型技术的金标准.本研究将15株山夫登堡沙门菌分为10个PFGE型,其中X4、X7型来自禽肉制品,X5、X9来自猪肉制品,这4个PFGE型分别包括2株相似度为100%的菌株,表明在各自PFGE型中的菌株污染来源一致.X3包含的2株菌株,1株分离自2009年的水产品,1株分离自2010年的猪肉蛋饺,表明这一PFGE型的沙门菌有可能已经在环境中流行,并造成不同来源食品的交叉污染.已有国外的研究证明,含有山夫登堡沙门菌的水产品被海水污染后,形成了生态循环能力,从而成为持续的污染源[12].另外,X3、X4、X5这3种不同来源菌株的PFGE型相似度高达88.2%,按照Tenover等[13]用相似度进行同源性分析的解释,认为相似性≥85%的菌株是高度同源,可能来自同一克隆株,即呈流行相关性,提示上海地区存在优势PFGE型并且这一PFGE型及其变型可能是造成上海地区山夫登堡沙门菌病散发和集中暴发的根源.
| [1] |
FRESNO M, BARRETO M, GUTIERREZ S, et al. Serotype-associated polymorphisms in a partial rpoB gene sequence of Salmonella enterica[J]. Can J Microbiol, 2014, 60(3): 177-181. DOI:10.1139/cjm-2013-0872 (  0) 0) |
| [2] |
ZHANG J, JIN H, HU J, et al. Serovars and antimicrobial resistance of non-typhoidal Salmonella from human patients in Shanghai, China, 2006-2010[J]. Epidemiol Infect, 2014, 142(4): 826-832. DOI:10.1017/S0950268813001659 (  0) 0) |
| [3] |
许学斌, 顾宝柯, 金汇明, 等. 上海市沙门菌血清型流行特征[J]. 中国人兽共患病学报, 2009, 25(2): 156-158. DOI:10.3969/j.issn.1002-2694.2009.02.017 (  0) 0) |
| [4] |
许学斌, 顾宝柯, 陈敏, 等. 沙门菌检测方法的优化[J]. 检验医学, 2007, 22(6): 677-680. DOI:10.3969/j.issn.1673-8640.2007.06.017 (  0) 0) |
| [5] |
CLSI. Performance standards for antimicrobial susceptibility testing, 20th informational supplement (M100-S20)[S]. Wayne: Clinical and Laboratory Standards Institute, 2010.
(  0) 0) |
| [6] |
CHEN S, ZHAO S, WHITE D G, et al. Characterization of multiple-antimicrobial-resistant Salmonella serovars isolated from retail meats[J]. Appl Environ Microbiol, 2004, 70(1): 1-7. (  0) 0) |
| [7] |
RIBOT E M, FAIR M A, GAUTOM R, et al. Standardization of pulsed-field gel electrophoresis protocols for the subtyping of Escherichia coli O157:H7, Salmonella, and Shigella for PulseNet[J]. Foodborne Pathog Dis, 2006, 3(1): 59-67. DOI:10.1089/fpd.2006.3.59 (  0) 0) |
| [8] |
周晓红, 商晓春, 帅慧群, 等. 89株山夫登堡沙门菌脉冲场凝胶电泳分子分型及耐药性分析研究[J]. 疾病监测, 2012, 27(10): 764-767. DOI:10.3784/j.issn.1003-9961.2012.10.004 (  0) 0) |
| [9] |
DONG P, ZHU L, MAO Y, et al. Prevalence and profile of Salmonella from samples along the production line in Chinese beef processing plants[J]. Food Control, 2014, 38: 54-60. DOI:10.1016/j.foodcont.2013.09.066 (  0) 0) |
| [10] |
YANG B, QU D, ZHANG X, et al. Prevalence and characterization of Salmonella serovars in retail meats of marketplace in Shaanxi, China[J]. Int J Food Microbiol, 2010, 141(1/2): 63-72. (  0) 0) |
| [11] |
黄峥, 许学斌, 金汇明, 等. 一类罕见硫化氢阴性山夫登堡沙门菌腹泻株的分子溯源[J]. 上海预防医学, 2010, 22(2): 62-65. DOI:10.3969/j.issn.1004-9231.2010.02.002 (  0) 0) |
| [12] |
MARTINEZ-URTAZA J, LIEBANA E. Investigation of clonal distribution and persistence of Salmonella Senftenberg in the marine environment and identification of potential sources of contamination[J]. FEMS Microbiol Ecol, 2005, 52(2): 255-263. DOI:10.1016/j.femsec.2004.11.018 (  0) 0) |
| [13] |
TENOVER F C, ARBEIT R D, GOERING R V, et al. Interpreting chromosomal DNA restriction patterns produced by pulsed-field gel electrophoresis:Criteria for bacterial strain typing[J]. J Clin Microbiol, 1995, 33(9): 2233-2239. (  0) 0) |
 2015, Vol. 36
2015, Vol. 36