在畜禽养殖业中, 抗生素作为禽畜疾病预防及治疗药物、生长促进剂、饲料添加剂等被广泛应用.抗生素的大量使用是导致细菌耐药性产生的重要原因之一[1], 同时也刺激了耐药基因的传播, 耐药基因已经成为环境中的重要污染源.世界卫生组织已将抗生素耐药基因作为21世纪威胁人类健康最重大的挑战之一, 并宣布将在全球范围内对控制耐药基因做战略部署.耐药基因作为一类新型的环境污染物, 与其他污染物不同之处是它具有遗传性, 而且可以通过物种间遗传物质的交换无限制地传播.耐药基因一旦进入环境中, 会持久性地存在, 很难控制和消除, 将对人类的公共卫生和食品、饮用水的安全构成威胁, 损害人体的健康.研究表明, 即使携带抗生素耐药基因的微生物菌株死亡后, 携带抗生素耐药基因的裸露DNA也可以在脱氧核糖核酸酶(DNAse)的保护下在环境中长期存在, 直到环境中这些裸露的DNA分子最终转入到其他生物体细胞内, 即使选择压力消失, 这些产生的耐药基因也不会随之消失[2].
植物附生菌是一类附着在植物表面、以植物分泌物为营养的微生物类群.内生菌是指在其生活史的某一个阶段或整个阶段生活于健康植物各种组织和器官内部或细胞间隙并且正常情况下不引起病害的微生物, 包括细菌、真菌、放线菌等[3].有关蔬菜细菌的报道大多集中于细菌的生物多样性及其在植物中的分布, 以及它们的生物学作用等方面, 国外对环境中耐药基因的研究又大多集中于医院和水环境(包括地表水、地下水、沉积物和污水处理厂等).因此对蔬菜中耐药基因的污染水平和种类进行调查是非常有必要的.本试验就华南地区蔬菜中细菌的耐药基因携带情况进行调查, 以期为保障蔬菜等植物性食品的质量安全和人畜健康提供科学依据.
1 材料与方法 1.1 材料 1.1.1 样品受试样品是2012-2013年从华南地区10个不同猪场采集的地瓜叶、生菜、白菜和葱等蔬菜.样品编号为A ~ J, 详见表 1.
|
|
表 1 华南地区采集的蔬菜样品 Table 1 Vegetable samples selected from Southern China |
液氮购自佛山气体公司; NA琼脂、NB肉汤购自青岛海博生物技术有限公司; 次氯酸钠、无水乙醇、PBS为北京普博欣公司产品; rTaq DNA聚合酶、DL2000 DNA Marker、dNTP Mixture、DNA提取试剂盒Bacterial DNA Kit为宝生物工程(大连)有限公司产品; 琼脂糖为西班牙Biowest公司产品.
1.2 方法 1.2.1 样品的采集猪场的菜地采样按照“S”形法布点, 采样点不少于5个, 同一猪场菜地采集的样品混合成1份样品.按试验所用量的3倍采集蔬菜的茎、叶, 分别装入塑料袋, 粘贴标签, 扎紧袋口.采集好的样品放入冷藏箱中(4 ℃), 立即送往实验室进行分析检验.
1.2.2 细菌的提取(前处理)将蔬菜样品用双蒸水冲洗干净.先用体积分数为1%的次氯酸钠和体积分数为0.02%吐温-20的混合溶液浸泡1 min, 再用体积分数为75%无水乙醇浸泡1 min, 最后用PBS溶液浸泡3次, 每次1 min.用无菌水充分淋洗, 无菌滤纸吸干.在超净工作台内用无菌手术刀将茎切成0.1 ~0.5 cm的小段, 将叶切成0.5 cm × 0.5 cm的小块.附生细菌的提取:取10 g切好的茎、叶, 加入20 mL含质量浓度为0.008 5 kg·L-1的NaCl溶液和体积分数为0.01%吐温-20的混合溶液, 于4 ℃条件下浸泡1 h, 将浸泡液用无菌水梯度稀释至10-2、10-3、10-4, 取200 μL稀释液均匀涂布于NA平板上, 每个梯度3个重复, 静置30 min后, 倒置于25 ℃恒温培养箱中培养5 d.内生细菌的提取:取10 g切好的茎、叶, 加入20 mL含质量浓度为0.008 5 kg· L-1的NaCl溶液和体积分数为0.01%吐温-20的混合溶液, 于4 ℃条件下浸泡1 h, 用体积分数为75%无水乙醇浸泡1 min, 再用体积分数为5%的次氯酸钠溶液浸泡5 min, 用无菌水充分淋洗, 无菌滤纸吸干.最后加适量液氮到陶瓷研钵中迅速、充分研磨.取100 μL研磨液加入NB培养液中, 于37 ℃恒温振荡器培养24 h, 然后将培养液用无菌水梯度稀释至10-2、10-3、10-4, 取200 μL稀释液均匀涂布于NA平板上, 每个梯度3个重复, 正置30 min后, 倒置于25 ℃恒温培养箱中培养5 d.
1.2.3 基因的提取选取长有100 ~ 200个单菌落的梯度稀释的NA平板, 用10 mL灭菌双蒸水将单菌落冲洗下来, 按照E.Z.N.A.Bacterial DNA Kit试剂盒的说明书提取细菌DNA.
1.2.4 引物的设计与合成根据参考文献报道的相应基因的引物序列, 由北京华大生物工程有限公司合成引物.将引物用灭菌双蒸水进行100倍稀释, 存放至4 ℃冰箱, 引物使用时置于冰上.所用引物见表 2.
|
|
表 2 试验所用的引物及其条件 Table 2 PCR primers and conditions used in this study |
PCR产物送北京六合华大基因测序.Blast程序在GenBankTM中对目的基因序列进行相似性检索分析.
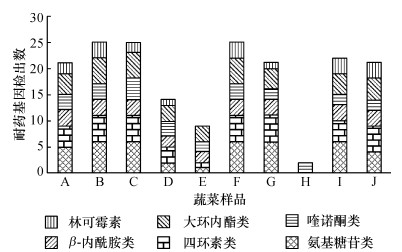
2 结果与分析 2.1 蔬菜样品附生细菌中耐药基因的检测结果从10个蔬菜样品中提取10份附生细菌样品, 对其进行6类27种耐药基因检测.猪场环境附生细菌耐药基因的携带情况见图 1, 按耐药基因的类别计算, A、B、C、D、F、G、I和J样品中均检测出了上述6类耐药基因, 检出率100% (6/6).样品E中检测到了5类耐药基因, 检出率83.33% (5/6), 而样品H中仅检测到了1类耐药基因, 检出率16.67% (1/6).从耐药基因的种类数目计算, A ~ J样品中耐药基因检出率分别是77.78% (21/27)、92.59% (25/27)、92.59% (25/27)、51.85% (14/27)、33.33% (9/27)、92.59% (25/27)、77.78% (21/27)、7.41% (2/27)、81.48% (22/27)和77.78% (21/27).其中, B、C、F样品的耐药基因检出率最高, 均为92.59%.从耐药基因分布范围来看(图 1), 喹诺酮类耐药基因在10个猪场的蔬菜样品中均有分布, 检出率100% (10/10), 四环素类、大环内酯类、氨基糖苷类和β-内酰胺类耐药基因的检出率均为90% (9/10), 林可霉素耐药基因的检出率为80% (8/10).由此看出, 猪场环境蔬菜附生细菌中此6类耐药基因的分布范围是非常广泛的, 其中喹诺酮类耐药基因的分布最为广泛.
|
图 1 蔬菜附生细菌的耐药基因检出数 Figure 1 The number of resistance genes in epiphytic bacteria isolated from vegetables |
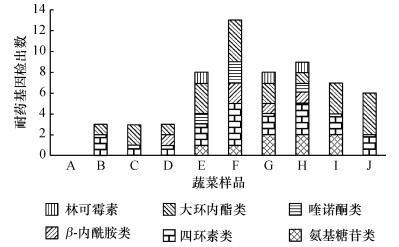
从10个蔬菜样品中提取10份内生细菌样品, 对其进行6类27种耐药基因的检测.猪场环境蔬菜内生细菌耐药基因的携带情况见图 2.按耐药基因的类别计算, H样品检测出了上述6类耐药基因, 检出率100% (6/6), E、F、G样品分别检测到5类耐药基因, 检出率83.33%(5/6), D和I样品分别检测到了3类耐药基因, 检出率50.00% (3/6), B、C、J样品检测到了2类耐药基因, 检出率33.33% (2/6), A样品未检出任何种类耐药基因.按耐药基因的种类数目计算, A ~ J样品中耐药基因的检出率分别是0(0/27)、11.11% (3/27)、11.11% (3/27)、11.11% (3/27)、29.63% (8/27)、48.15% (13/27)、29.63% (8/27)、33.33%(9/27)、25.93% (7/27)和22.22% (6/27).其中, F样品中耐药基因的检出率最高(48.15%), 高于耐药基因类别检出率高的H样品(33.33%).其次是H、E、G、I、J样品, 检出率较低的是B、C、D样品, 但均高于A样品的检出率(0.00%).对比蔬菜样品附生细菌中的耐药基因检出情况, 可以看出, 蔬菜样品中附生细菌(图 1)和内生细菌(图 2)所含的耐药基因的种类是有差别的, 并且附生细菌中的耐药基因比内生细菌的丰富.从耐药基因分布范围来看(图 2), 四环素类和大环内酯类耐药基因在9个猪场的蔬菜内生细菌样品中均有分布, 检出率均为90% (9/10), 氨基糖苷类、β-内酰胺类、林可霉素和喹诺酮类耐药基因的检出率分别为50% (5/10)、40% (4/10)、30% (3/10)和30% (3/10).由此看出, 四环素类和大环内酯类耐药基因的分布范围是非常广泛的.对比附生细菌中耐药基因的分布情况, 喹诺酮类耐药基因在附生细菌中的检出率为100% (10/10), 而在内生细菌中检出率为30% (3/10), 可见附生细菌中喹诺酮类耐药基因的分布范围比内生细菌的广泛.四环素类和大环内酯类耐药基因在附生细菌和内生细菌的检出率均为90% (9/10), 其分布范围是非常广泛的.
|
图 2 蔬菜内生细菌的耐药基因检出数 Figure 2 The number of resistance genes in endophytic bacteria isolated from vegetables |
本研究中, 对从华南地区10个不同猪场采集的地瓜叶、生菜、白菜和葱等蔬菜中提取的附生和内生细菌进行耐药基因携带情况的调查发现, A样品的附生细菌中检测出了6类耐药基因, 检出率100% (6/6), 而A样品的内生细菌中没有检出任何耐药基因, 检测率0, H样品的附生细菌中只检测到1类耐药基因, 检出率是16.67% (1/6), 而H样品的内生细菌中检测到了6类耐药基因, 检出率100% (6/6), 这可能与蔬菜对抗生素的吸收富集能力、其生产环境污染状况等因素及附生细菌、内生细菌所处的环境不同有关, 内生细菌在植物体内具有稳定的生存空间, 不易受环境条件的影响, 也可能是由于某些细菌的内生性很强, 用一般条件根本无法分离到.Kumar等[13]研究了玉米、洋葱与卷心菜从施用粪肥的土壤中吸收金霉素与泰乐菌素的情况, 3种作物均可从土壤中吸收金霉素而不吸收泰乐菌素, 说明蔬菜对抗生素的吸收是有选择性的.在本研究中, 附生细菌中喹诺酮类耐药基因在10个猪场的蔬菜样品中均有分布, 检出率100% (10/10), 其分布范围是最广的, 这可能与喹诺酮类药物具有抗菌谱广、抗菌活性强、体内分布广泛等优点, 而且可以大量地人工合成, 在结构上也具有较好的可塑性, 在人类和畜牧业广泛应用有关.朱恒乾等[14]对广州分离得到的宠物源(主要是宠物猫、狗)分离株进行质粒介导喹诺酮类耐药PMQR基因流行性调查, 检测到qnrA、qnrB、qnrD、qnrS和aac(6’) -Ib-cr基因.Hata等[15]在福氏志贺菌Shigella flexneri 2b临床分离株中发现了qnrS基因.Jacoby等[16]在肺炎克雷伯菌Klebsiella pneumoniae中发现了qnrB基因.岳磊等[17]从广东地区兽医临床动物源肠杆菌中检测到qnrA、qnrB、qnrS基因.Rodriguez-Martinez等[18]从美国和欧洲人体中以临床分离株检测到了qnrA基因.Wang等[19]从上海人体中以临床分离株检测到了qnrA基因.Wu等[20]从中国台湾人体中以临床分离株检测到qnrA、qnrB、qnrS基因.不同来源的细菌都分离到喹诺酮类耐药基因, 说明喹诺酮类耐药基因的分布非常广泛.
在本研究中, 蔬菜附生细菌和内生细菌中四环素类耐药基因的检出率较高, 说明这些细菌很可能存在严重的耐药问题.这可能是由于长期施用含四环素抗生素的粪便作为肥料或者蔬菜吸收了土壤中的抗生素造成的.猪场是四环素等抗生素使用较为频繁的地方.吴楠等[21]提取了北京一规模化养猪场周边土壤的微生物DNA, 利用普通PCR检测到5种四环素抗性基因(tetB/P、tetM、tetO、tetT、tetW)的存在.邹世春等[22]采用PCR方法研究了养殖场土壤细菌DNA中9种四环素抗性基因的存在和污染水平.结果表明, 所研究的9种四环素抗性基因中除tetD之外, 其他8种抗性基因(tetA、tetB、tetC、tetG、tetL、tetO、tetQ和tetX)均可检出.Chee-Sanford等[23]采用PCR-DGGE和序列分析技术在养猪场附近的粪池中检测到8种编码核糖体保护蛋白(RJPP)的四环素耐药基因, 分别是tetO、tetQ、tetW、tetM、tetB/P、tetS、tetT和otrA, 在其他许多养殖场如猪场、牛场等的周围环境中也常被检测到耐药基因tetW、tetT、tetM、tetO, 说明这几种四环素耐药基因在养殖场周围环境中是广泛存在的.本试验蔬菜样品B、C、F、G和J的附生细菌中分别检测到5种四环素类耐药基因.Schmitt等[24]通过PCR方法也证明了土壤中四环素耐药基因的多样性, 这与本试验的结果是一致的.由于不同种类的抗生素对应存在着不同的耐药基因, 而每种抗生素的耐药基因又有很多种, 加上客观条件(如环境条件)、检测方法等因素的影响, 很难将各种耐药基因进行详细统计.虽然本试验只基于10个猪场蔬菜样品的部分耐药基因进行研究, 但从一定程度上证实了耐药基因的多样性以及在动物养殖系统与周围环境之间传播的可能性.另外, 由于耐药基因可以整合到一些可移动元件(如质粒、转座子、整合子等)上, 并且能够在致病菌和非致病菌之间, 甚至在革兰阳性菌和革兰阴性菌之间相互传播.通过这种基因横向转移, 耐药基因很可能通过食物链危及动物和人体健康.
本试验调查了华南地区10个不同猪场蔬菜样品中附生细菌和内生细菌的耐药基因携带情况.通过PCR结合测序的方法检测氨基糖苷类、四环素类、β-内酰胺类、喹诺酮类、大环内酯类和林可霉素共计6类27种耐药基因, 结果发现, 内生细菌中四环素类耐药基因的检出率特别高, 附生细菌携带的耐药基因不论种类还是数目都比内生细菌的广泛和丰富.提示猪场蔬菜中附生细菌耐药基因携带情况已经非常严重, 应给予足够的关注和重视.
| [1] |
WITTE W. Medical consequences of antibiotic use in agriculture[J]. Science, 1998, 279(5353): 996-997. DOI:10.1126/science.279.5353.996 (  0) 0) |
| [2] |
MANSON J M, SMITH J M, COOK G M. Persistence of vancomycin-resistant enterococci in New Zealand broilers after discontinuation of avoparcin use[J]. Appl Environ Microbiol, 2004, 70(10): 5764-5768. DOI:10.1128/AEM.70.10.5764-5768.2004 (  0) 0) |
| [3] |
ANDREWS J H, HARRIS R F. The ecology and biogeography of microorganisms on plant surfaces[J]. Annu Rev Phytopathol, 2000, 38(1): 145-180. DOI:10.1146/annurev.phyto.38.1.145 (  0) 0) |
| [4] |
CATTOIR V, POIREL L, NORDMANN P. Plasmid-mediated quinolone resistance determinant QnrB4 identified in France in an Enterobacter cloacae clinical isolate coexpressing a QnrS1 determinant[J]. Antimicrob Agents Chemother, 2007, 51(7): 2652-2653. DOI:10.1128/AAC.01616-06 (  0) 0) |
| [5] |
KIM H B, PARK C H, KIM C J, et al. Prevalence of plasmid-mediated quinolone resistance determinants over a 9-year period[J]. Antimicrob Agents Chemother, 2009, 53(2): 639-645. DOI:10.1128/AAC.01051-08 (  0) 0) |
| [6] |
NG L K, MARTIN I, ALFA M, et al. Multiplex PCR for the detection of tetracycline resistant genes[J]. Mol Cell Probes, 2001, 15(4): 209-215. DOI:10.1006/mcpr.2001.0363 (  0) 0) |
| [7] |
AMINOV R I, GARRIGUES-JEANJEAN N, MACKIE R I. Molecular ecology of tetracycline resistance: Development and validation of primers for detection of tetracycline resistance genes encoding ribosomal protection proteins[J]. Appl Environ Microbiol, 2001, 67(1): 22-32. DOI:10.1128/AEM.67.1.22-32.2001 (  0) 0) |
| [8] |
LINA G, QUAGLIA A, REVERDY M E, et al. Distribution of genes encoding resistance to macrolides, lincosamides, and streptogramins among Staphylococci[J]. Antimicrob Agents Chemother, 1999, 43(5): 1062-1066. (  0) 0) |
| [9] |
DE GRAEF E M, DECOSTERE A, DE LEENER E, et al. Prevalence and mechanism of resistance against macrolides, lincosamides, and streptogramins among Enterococcus faecium isolates from food-producing animals and hospital patients in Belgium[J]. Microb Drug Resist, 2007, 13(2): 135-141. DOI:10.1089/mdr.2007.718 (  0) 0) |
| [10] |
SINGH K V, WEINSTOCK G M, MURRAY B E. An Enterococcus faecalis ABC homologue (Lsa) is required for the resistance of this species to clindamycin and quinupristin-dalfopristin[J]. Antimicrob Agents Chemother, 2002, 46(6): 1845-1850. DOI:10.1128/AAC.46.6.1845-1850.2002 (  0) 0) |
| [11] |
SUTCLIFFE J, GREBE T, TAIT-KAMRADT A, et al. Detection of erythromycin-resistant determinants by PCR[J]. Antimicrob Agents Chemother, 1996, 40(11): 2562-2566. (  0) 0) |
| [12] |
KEHRENBERG C, MEUNIER D, TARGANT H, et al. Plasmid-mediated florfenicol resistance in Pasteurella trehalosi[J]. J Antimicrob Chemother, 2006, 58(1): 13-17. DOI:10.1093/jac/dkl174 (  0) 0) |
| [13] |
KUMAR K, GUPTA S C, BAIDOO S K, et al. Antibiotic uptake by plants from soil fertilized with animal manure[J]. J Environ Qual, 2005, 34(6): 2082-2085. DOI:10.2134/jeq2005.0026 (  0) 0) |
| [14] |
朱恒乾, 廖晓萍, 陈朝喜, 等. 宠物源大肠杆菌质粒介导喹诺酮类耐药基因流行性检测[J]. 中国农业科学, 2010, 43(16): 3447-3454. DOI:10.3864/j.issn.0578-1752.2010.16.022 (  0) 0) |
| [15] |
HATA M, SUZUKI M, MATSUMOTO M, et al. Cloning of a novel gene for quinolone resistance from a transferable plasmid in Shigella flexneri 2b[J]. Antimicrob Agents Chemother, 2005, 49(2): 801-803. DOI:10.1128/AAC.49.2.801-803.2005 (  0) 0) |
| [16] |
JACOBY G A, WALSH K E, MILLS D M, et al. qnrB, another plasmid-mediated gene for quinolone resistance[J]. Antimicrob Agents Chemother, 2006, 50(4): 1178-1182. DOI:10.1128/AAC.50.4.1178-1182.2006 (  0) 0) |
| [17] |
岳磊, 蒋红霞, 刘健华, 等. 鸡源肠杆菌质粒介导喹诺酮类耐药基因检测[J]. 中国农业科学, 2009, 42(8): 2966-2971. DOI:10.3864/j.issn.0578-1752.2009.08.040 (  0) 0) |
| [18] |
RODRIGUEZ-MARTINEZ J M, PASCUAL A, GARCIA I, et al. Detection of the plasmid-mediated quinolone resistance determinant qnr among clinical isolates of Klebsiella pneumoniae producing AmpC-type beta-lactamase[J]. J Antimicrob Chemother, 2003, 52(4): 703-706. DOI:10.1093/jac/dkg388 (  0) 0) |
| [19] |
WANG M, SAHM D F, JACOBY G A, et al. Emerging plasmid-mediated quinolone resistance associated with the qnr gene in Klebsiella pneumoniae clinical isolates in the United States[J]. Antimicrob Agents Chemother, 2004, 48(4): 1295-1299. DOI:10.1128/AAC.48.4.1295-1299.2004 (  0) 0) |
| [20] |
WU J J, KO W C, TSAI S H, et al. Prevalence of plasmid-mediated quinolone resistance determinants QnrA, QnrB, and QnrS among clinical isolates of Enterobacter cloacae in a Taiwanese hospital[J]. Antimicrob Agents Chemother, 2007, 51(4): 1223-1227. DOI:10.1128/AAC.01195-06 (  0) 0) |
| [21] |
吴楠, 乔敏, 朱永官. 猪场土壤中5种四环素抗性基因的检测和定量[J]. 生态毒理学报, 2009, 4(5): 705-710. (  0) 0) |
| [22] |
邹世春, 朱春敬, 贺竹梅, 等. 北江河水中抗生素抗性基因污染初步研究[J]. 生态毒理学报, 2009, 4(5): 655-660. (  0) 0) |
| [23] |
CHEE-SANFORD J C, AMINOV R I, KRAPAC I J, et al. Occurrence and diversity of tetracycline resistance genes in lagoons and groundwater underlying two swine production facilities[J]. Appl Environ Microbiol, 2001, 67(4): 1494-1502. DOI:10.1128/AEM.67.4.1494-1502.2001 (  0) 0) |
| [24] |
SCHMITT H, STOOB K, HAMSCHER G, et al. Tetracyclines and tetracycline resistance in agricultural soils: Microcosm and field studies[J]. Microb Ecol, 2006, 51(3): 267-276. DOI:10.1007/s00248-006-9035-y (  0) 0) |
 2015, Vol. 36
2015, Vol. 36

 ,
,