2. 华南农业大学 动物科学学院, 广东 广州 510642;
3. 广东大华农动物保健品股份有限公司, 广东 新兴 527400
2. College of Animal Science, South China Agricultural University, Guangzhou 510642, China;
3. Guangdong Dahuanong Animal Health Products Co., Ltd., Xinxing 527400, China
鸭坦布苏病毒(Duck Tembusu virus, DTMUV)属于黄病毒科黄病毒属恩他耶病毒群, 其基因组结构与其他黄病毒相似, 是一种约有11 kb单股正链的RNA病毒.基因组仅有1个大的阅读框架(ORF), 两端是与病毒复制有关的5′端和3′端非编码区[1-2].基因组编码1个大的蛋白前体, 后在宿主信号肽酶和病毒丝氨酸蛋白酶的共同催化作用下裂解为3个结构蛋白和7个非结构蛋白, 在基因组中的顺序为: 5′- C-prM-E-NS1-NS2A-NS2B-NS3-NS4A-NS4B-NS5-3′, 其中E蛋白是黄病毒主要的结构蛋白, 在病毒吸附、与宿主细胞膜融合以及病毒组装过程中具有重要的作用.同时, E蛋白也是黄病毒主要的病毒抗原, 含有多种抗原表位, 可通过诱发中和抗体产生保护性免疫应答[3].NS5是最大的非结构蛋白, 含有910个氨基酸, 具有RNA依赖性RNA聚合酶、甲基转移酶、鸟苷酸转移酶等多种酶活性[4].Forwood等[5]发现登革病毒NS5茎环区的37个氨基酸(369 ~405位氨基酸)含有核定位信号(Nuclear localization signal, NLS), 在氨基端的327 ~343氨基酸含有核输出序列(NES), 在病毒基因组的复制过程中发挥关键作用.了解病毒基因结构特点是分析病毒遗传进化、生长繁殖、毒力、致病机理的分子基础, 有助于进一步的研究鸭黄病毒病工作的开展.因此本文测定并分析了2010-2013年分离于珠三角的6株鸭源坦布苏病毒全基因结构.
1 材料与方法 1.1 试验材料 1.1.1 病毒与鸭胚鸭黄病毒为广东自然分离毒株, 鸭胚由云城温氏公司孵化场提供.
1.1.2 菌株与载体大肠埃希菌Escherichia coli DH5a由广东大华农动物保健品股份有限公司生药研发中心保存, pGEM-T-easy vector system为Promega公司产品.
1.1.3 主要试剂RNA/DNA抽提试剂盒、DNA Marker DL2000、M-MLV反转录酶、(10 ×)PCR Buffer (含Mg2+)、dNTP、PrimeSTAR Max高保真DNA聚合酶、rTaq聚合酶均为TaKaRa公司产品; 凝胶回收试剂盒、小质粒抽提试剂盒为Omega公司产品; 氨苄青霉素(Amp)为Invirtogen公司产品; LB培养基购自广州环凯生物技术公司.
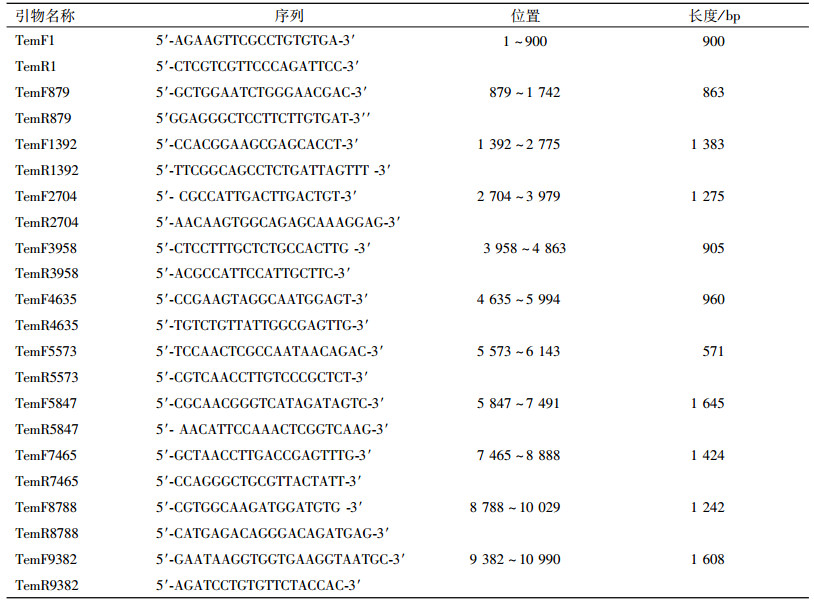
1.2 试验方法 1.2.1 鸭坦布苏病毒全基因序列的引物设计与合成参照GenBank收藏的坦布苏病毒JS804株(登录号: NC_015843)序列, 应用Primer premier5.0软件设计引物, 见表 1, 由广州英潍捷基贸易有限公司合成.
|
|
表 1 鸭坦布苏病毒病全基因组的PCR扩增引物 Table 1 PCR primers for amplification of the full-length genome of duck Tembusu virus |
参照TaKaRa产品RNA/DNA抽提试剂盒说明书步骤抽提病毒RNA.利用M-MLV反转录酶(TaKaRa公司)和随机引物将RNA反转录成cDNA:取模板2 μL、Ran- dom Primer 1 μL和RNase free ddH2O至6 μL, 70 ℃作用10 min后冰上急冷2 min; 离心数秒后, 将上述模板变性溶液6 μL与2 μL 5 × M-MLV Buffer、0.5 μL dNTP-Mixture、0.25 μL RNase Inhibitor、0.25 μL RNase M-MLV、1 μL RNase free ddH2O混匀, 合成第1链cDNA, 反应条件: 30 ℃ 10 min, 42 ℃ 1 h, 70 ℃ 15 min, 结束反应.取转录产物, 分别用上下游引物进行PCR扩增, 反应体系为: 2 × PrimeSTAR Max高保真DNA聚合酶预混剂25 μL、上下游引物各1 μL、cDNA模板2 μL, 灭菌ddH2O至50 μL, PCR反应条件: 95 ℃预变性5 min; 98 ℃变性10 s, 55 ℃退火10 s, 72 ℃延伸时间根据片段大小而定(10 s·kb-1), 35个循环; 72 ℃10 min, 获得目的片段.
1.2.3 目的基因的克隆与测序PCR扩增产物加A碱基后, 经0.01 g·mL-1琼脂糖凝胶电泳后, 切下目的条带, 用DNA凝胶回收试剂盒回收目的片段; 取目的片段连接pGEM-T-easy vector载体, 重组质粒转化DH5a感受态细胞; 转化后均匀涂布于含100 μg·mL-1的Amp的LB琼脂板上, 过夜, 挑取可疑阳性菌落, 摇菌, 抽提质粒PCR鉴定为阳性后送英潍捷基公司测序.
1.2.4 目的基因的序列结果与分析测序结果通过国际互联网提交NCBI Blast Server进行联机检索, 以及与GenBank中注册的其他黄病毒的相应核苷酸序列进行比较分析.应用DNAstar(Version 5.07)分析软件, 将所获得的4株鸭坦布苏病毒基因片段的序列分别进行整理和拼接, 形成完整的病毒基因序列.联合GDHZ2012.1、GDHZ2012.2病毒序列, DNAstar的MegAlign中的clustal W method算法比较不同地区的鸭坦布苏病毒各蛋白的核苷酸和氨基酸序列相似性, 用Mega 6.0软件的NJ算法, 绘制出系统发育进化树.
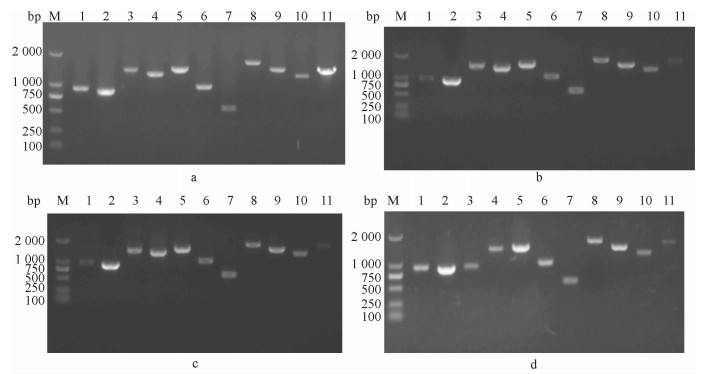
2 结果 2.1 全基因组各片段的扩增使用设计的引物经PCR扩增目的基因, 电泳后获得了与预期大小相符的条带(图 1).
|
图 1 GDNS2010.1(a)、GDZQ2012(b)、GDNS2010.2(c)、GDPY2013(d)全基因序列片段的PCR扩增 Figure 1 PCR amplification of the complete genome of GDNS2010.1(a), GDZQ2012(b), GDNS2010.2(c), GDPY2013(d) M: DNA marker DL2000; 1 ~ 11片段大小分别为900、863、1 383、1 275、905、960、571、1 645、1 424、1 242、1 608 bp. |
测序结果经SeqMan软件去载体、拼接后, 得到了病毒的全长核苷酸序列, 其中GDNS2010.1、GDNS2010.2两毒株在103 395 ~ 103 411 bp处缺失10个碱基; 编码区位于95 ~ 10 372 bp, 共编码3 462个氨基酸; 将拼接好的序列与GenBank上收藏的16个相关病毒序列(包含10个TMUV和6个黄病毒属成员病毒), 见表 2前16条序列.经MegAlign软件比对(图 2):分离株与其他TMUV毒株相似性都在98%以上, 其中GDHZ2012.1与GDPY2013两毒株与参考毒株JS804相似性最高, 分别为99.4%、99.3%;进化树分析显示, GDPY2013株与JS804处在同一节点; 在6毒株之间, GDZQ2012与其他毒株差异最大, 分离于同一地方的GDNS2010.1、GDNS2010.2和GDHZ2012.1、GDHZ2012.2的核苷酸相似性很高, 处于进化树上的同一节点, 可能由同一毒株进化得到.与黄病毒属成员相比较发现:相似性最高的是Sitiawan病毒, 相似性为87.5% ~ 88.1%, 其次为Ntaya-IPDIA株和以色列的火鸡脑脊髓炎病毒, 相似度都为72%以上, 而西尼罗河病毒、日本乙型脑炎病毒、黄热病毒等相比较, 相似性皆低于63%.

|
图 2 鸭坦布苏病毒株进化树(邻接法) Figure 2 Phylogenetic tree of DTMUV strains(by NJ method) |
|
|
表 2 参考毒株及序列登录号 Table 2 Reference strains in GenBank and their accession numbers |
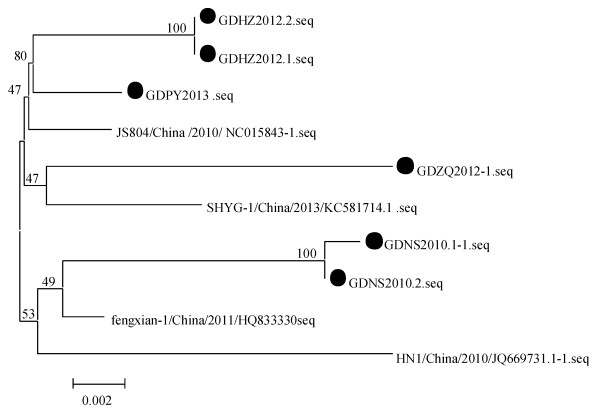
将病毒的主要结构蛋白E蛋白与其他TMUV (参考株见表 2后4条序列)比较发现, 核苷酸序列相似性为97.5% ~ 99.3%, 推导氨基酸序列相似性在97%以上(图 3).由图 3可见, 根据较保守的病毒E基因序列分析建树发现:珠三角分离的6株毒与国内其他地区分离的毒株在遗传关系上JS804与GDHZ2012.1、GDHZ2012.2、GDPY2013, GDZQ2012与SHYg-1, GDNS2010.1、GDNS2010.2与Fengxian-1分别有共同节点.这提示DTUMV病毒可能在近3年来并未形成较大差异的地方毒株.GDNS2010.1、GDNS2010.2、GDZQ2012、GDHZ2012.1、GDHZ2012.2、GDPY2013各毒株与参考毒株JS804相比较碱基变化较多, 但多为无义突变.6株毒株间推导氨基酸变化情况见表 3: GDHZ2012.1、GDHZ2012.2第128位氨基酸由极性带正电荷的Arg变为非极性不带电荷的Met; GDHZ2012.2第289位由极性带正电荷的Lys变为极性带负电荷的Glu; 在第458位由极性不带电荷的Ser变为非极性Pro; GDPY2013株在313位由极性带正电荷的Arg变为非极性不带电荷的Met.这些改变蛋白极性或带电荷数的位点很可能就是潜在的毒力位点.另外, 囊膜蛋白第154位存在一个潜在的糖基化位点(Asn154-Try-Ser), 这一位点在大多数黄病毒中保守[6].
|
图 3 鸭坦布苏病毒E基因遗传进化树分析(邻接法) Figure 3 Phylogenetic tree of envelope gene of DTMUV strains(by NJ method) |
|
|
表 3 E基因推导氨基酸突变表 Table 3 The mutations in the deduced amino acid sequences of envelope gene |
2010年鸭坦布苏病毒病的突然爆发与迅速蔓延给我国水禽养殖带来了巨大的经济损失.目前认为鸭坦布苏病毒只感染鸭、鹅、家雀, 但同时鸭黄病毒属于黄病毒科黄病毒属的成员; 而黄病毒属中大量成员威胁人类健康, 如日本乙型脑炎、登革热、黄热病、蜱传染性脑炎等[7-8].Tang等[9]对132份来自鸭产业工作人员的血清检测发现其抗体阳性率达71.9%, 63份口腔拭子检测病毒RNA阳性率达47.7%, 这是否意味着鸭坦布苏病毒同其他的黄病毒属成员一样具有公共卫生意义还需进一步确认.虽鸭坦布苏病毒是一种国内新发疾病, 但近两年报道病毒的全基因序列已有几十条, Yu等[3]对2010-2012年分离到的6株坦布苏病毒的E蛋白结构、抗原性和进化特性的研究发现, 坦布苏病毒分I、Ⅱ2个基因型, Ⅱ为主要的基因型, 他们还发现病毒E蛋白氨基酸突变位点有13处, 而中和试验病毒的中和特性却没有表现出差异.本试验将2010-2013年分离测序获得的6株病毒核苷酸序列与GenBank收藏的10株坦布苏病毒相比较, 发现6毒株的核苷酸相似性与近4年国内报道的坦布苏病毒的核苷酸序列相似性都很高, 在97%以上, 这提示在4年间里坦布苏病毒并未发生大的变异, 感染宿主范围并没有扩大; 国内也未出现如2010年的大范围流行, 仅出现地方性流行[10].这可能一方面与感染宿主逐渐产生特异性抗体, 形成了免疫保护有关; 另一方面可能与目前国内缺乏有效防治此病疫苗与药物的情况有关, 在这种情况下病毒面临的选择压力小, 没能使病毒发生大的新的变异.但这不能否认病毒在各个地方呈现地方特色, 如在前期试验中, 笔者发现这6毒株中2012年分离于惠州的2株病毒就存在毒力差异, 测定发现GDHZ2012.1的EID50比GDHZ2012.2的EID50高出2个稀释度, 第5代毒颅内接种7日龄小鸭时前者能引起明显的临床症状, 甚至死亡, 后者发病较缓, 且临床症状较轻[11].巧合的是在E蛋白基因序列里GDHZ2012.2比GDHZ2012.1多2个突变位点, 尤其是GDHZ2012.2第289位由极性带正电荷的Lys变为极性带负电荷的Glu, 这一位点使得蛋白的带电荷数发生改变.已有研究表明, 第289位氨基酸位于E蛋白结构域Ⅱ中, 结构域Ⅱ中有糖基化位点和具有血清学及生物活性的抗原表位[12-14].
在序列拼接完成后, 发现GDNS2010.1、GDNS2010.2毒株在103 395 ~ 103 411 bp处缺失10个碱基.有趣的是这2毒株的EID50都不高, 第5代毒的EID50分别为10-3.13、10-2.8, 这段缺失序列处在病毒的3′UTR, 这段缺失的序列是否是病毒复制能力低于其他4株毒的原因, 可通过感染性克隆技术作进一步研究确认, 文献报道的是黄病毒的3′UTR影响着病毒的复制、增殖, 调控病毒翻译效率[15-16].
鸭坦布苏病毒病作为一种国内新发的传染性疾病, 目前对于其流行病学特点、致病机理等生物学特征尚不是很清楚, 临床上缺乏有效的检测手段与防治疫苗、药物; 也不清楚是否威胁人类健康.本文测定分析了2010-2013年间分离于珠三角地区的6株鸭源坦布苏病毒, 为下一步的疫苗筛选、分子诊断、反向操作奠定了基础.
| [1] |
BAI X, LU R, LIU C, et al. Molecular characterization of a duck Tembusu virus from China[J]. Virus Genes, 2013, 47(3): 478-482. DOI:10.1007/s11262-013-0966-3 (  0) 0) |
| [2] |
MUKHOPADHYAY S, KUHN R J, ROSSMANN M G. A structural perspective of the flavivirus life cycle[J]. Nat Rev Microbiol, 2005, 3(1): 13-22. DOI:10.1038/nrmicro1067 (  0) 0) |
| [3] |
YU K, SHENG Z Z, HUANG B, et al. Structural, anti- genic, and evolutionary characterizations of the envelope protein of newly emerging duck Tembusu virus[J]. PLoS One, 2013, 8(8): e71319. DOI:10.1371/journal.pone.0071319 (  0) 0) |
| [4] |
LU G, GONG P. Crystal structure of the full-length Japa- nese encephalitis virus NS5 reveals a conserved methyl- transferase-polymerase interface[J]. PLoS Pathog, 2013, 9(8): e1003549. DOI:10.1371/journal.ppat.1003549 (  0) 0) |
| [5] |
FORWOOD J K, BROOKS A, BRIGGS L J, et al. The 37-amino-acid interdomain of dengue virus NS5 protein contains a functional NLS and inhibitory CK2 site[J]. Biochem Biophys Res Commun, 1999, 257(3): 731-737. DOI:10.1006/bbrc.1999.0370 (  0) 0) |
| [6] |
ZHANG Y, CHEN P, CAO R, et al. Mutation of putative N-linked glycosylation sites in Japanese encephalitis virus premembrane and envelope proteins enhances humoral im- munity in BALB/C mice after DNA vaccination[J]. Vir- ol J, 2011, 8: 138.
(  0) 0) |
| [7] |
TANG Y, DIAO Y, YU C, et al. Characterization of a Tembusu virus isolated from naturally infected house spar- rows (Passer domesticus) in Northern China[J]. Trans- bound Emerg Dis, 2013, 60(2): 152-158. DOI:10.1111/tbed.2013.60.issue-2 (  0) 0) |
| [8] |
ZHU W, CHEN J, WEI C, et al. Complete genome se- quence of duck Tembusu virus, isolated from Muscovy ducks in southern China[J]. J Virol, 2012, 86(23): 13119. DOI:10.1128/JVI.02361-12 (  0) 0) |
| [9] |
TANG Y, GAO X, DIAO Y, et al. Tembusu virus in hu- man, China[J]. Transbound Emerg Dis, 2013, 60(3): 193-196. DOI:10.1111/tbed.2013.60.issue-3 (  0) 0) |
| [10] |
刘志刚, 孙青松, 姚蓉, 等. 鸭坦布苏病毒研究进展[J]. 中国动物传染病学报, 2013, 21(1): 81-86. DOI:10.3969/j.issn.1674-6422.2013.01.015 (  0) 0) |
| [11] |
李庆阳, 陈芳艳, 刘平, 等. 鸭坦布苏病毒TaqMan荧光定量RT-PCR检测方法的建立[J]. 动物医学进展, 2012, 33(7): 18-22. DOI:10.3969/j.issn.1007-5038.2012.07.004 (  0) 0) |
| [12] |
GRITSUN T S, HOLMES E C, GOULD E A. Analysis of flavivirus envelope proteins reveals variable domains that reflect their antigenicity and may determine their pathogen- esis[J]. Virus Res, 1995, 35(3): 307-321. DOI:10.1016/0168-1702(94)00090-Y (  0) 0) |
| [13] |
KIMURA-KURODA J, YASUI K. Antigenic comparison of envelope protein E between Japanese encephalitis virus and some other flaviviruses using monoclonal antibodies[J]. J Gen Virol, 1986, 67(Pt 12): 2663-2672. (  0) 0) |
| [14] |
张羽, 曹瑞斌, 顾金燕, 等. 猪源乙型脑炎病毒NJ2008株E138位突变病毒的构建与生物学特性初步研究[C]∥佚名. 中国畜牧兽医学会兽医公共卫生学分会第二次学术研讨会论文集. 北京: 中国畜牧兽医学会, 2014: 312-315.
(  0) 0) |
| [15] |
ALVAREZ D E, EZCURRA A L D L, FUCITO S, et al. Role of RNA structures present at the 3' UTR of dengue virus on translation, RNA synthesis, and viral replication[J]. Virology, 2005, 339(2): 200-212. DOI:10.1016/j.virol.2005.06.009 (  0) 0) |
| [16] |
PROUTSKI V, GOULD E A, HOLMES E C. Secondary structure of the 3' untranslated region of flaviviruses: Simi- larities and differences[J]. Nucleic Acids Res, 1997, 25(6): 1194-1202. DOI:10.1093/nar/25.6.1194 (  0) 0) |
 2015, Vol. 36
2015, Vol. 36