猪流行性腹泻(Porcine epidemic diarrhea,PED)是由猪流行性腹泻病毒(Porcine epidemic diarrhea vi-rus,PEDV)引起的急性、接触性胃肠道疾病,感染仔猪临床表现为呕吐、腹泻,最终因严重脱水而死亡; 而成年猪感染只表现轻微的临床症状,如精神抑郁、食欲下降,一段时间后恢复正常[1].PEDV虽首次在欧洲报道[2],但近年在亚洲和北美的流行对养猪业造成的经济损失远大于欧洲.亚洲如泰国[3]、韩国[4]、日本[5]、越南与中国[6]等国家,因近年PEDV的流行,对养猪业影响尤为严重.2013年5月此病毒在美国[7]首次发现,并在之后迅速传播至美各个州和周边的加拿大与墨西哥,目前仍无有效的控制措施.虽然采取疫苗接种方式来控制此病流行,但疫苗不能对整个猪群提供完全的保护效果,据报道接种过疫苗的猪场仍然有此病的流行[8-9].PEDV属于套式病毒目冠状病毒科冠状病毒属,与猪传染性胃肠炎病毒同属,生物学特性及致病性都相似[10].PEDV基因组大小约28 kb,由7个ORFs组成.ORF1a和ORF1b占病毒基因组2 /3,编码病毒RNA复制酶; ORF3为辅助性基因,编码一多态性的辅助性蛋白; 其余基因分别编码病毒4个结构蛋白:纤突糖(S)蛋白,小囊膜(E)蛋白,膜(M)蛋白和核(N)蛋白[1].S蛋白由S1和S2两部分组成,S1介导病毒与宿主细胞的吸附,S2诱导构象改变使病毒易于进入宿主细胞[1].中国于1979年发现PEDV,一直呈散发状态.自2012年10月呈大规模流行趋势,对哺乳仔猪的危害最为严重,感染后死亡率高达100%,对养猪业造成重大经济损失[8-9].此病毒在发现10年之后,Hofmann等[11]在Vero细胞中添加胰酶的方式终于成功培养.本试验参考其他分离病毒的方法[12-15],将猪流行性腹泻病毒疑似病料处理后,接种Vero细胞以分离病毒.通过RT-PCR特异性片段的检测、典型病变的观察和间接免疫荧光(IFA)试验,鉴定所分离到的毒株为PEDV,并对 S 基因进行遗传进化分析.PEDV在细胞上的成功分离及 S 基因的遗传进化分析,为以后生物学、疫苗开发等研究提供了基础.
1 材料与方法 1.1 主要试剂与仪器血清(BI公司); DEME细胞培养液、胰蛋白酶(GIBCO公司); RNA抽提试剂盒、M-MLV反转录酶、RNA抑制酶、dNTP、T载体、Taq DNA复制酶、胶回收试剂盒(TaKaRa公司); PEDV M蛋白单克隆抗体(Median Diagnostic公司); 荧光倒置显微镜(Leica公司); Vero细胞由华南农业大学兽医学院保存.
1.2 病料采集与处理样品来自广东某猪场疑似猪流行性腹泻感染的病例,采集的粪便样品于-80 ℃条件保存备用.采样猪场感染仔猪临床表现为水样腹泻,呕吐; 死亡仔猪小肠充满黄色液体,肠壁变薄呈透明状.
采集的样品与预冷PBS液1:5稀释,反复冻融3次,最后于4 ℃以5 000 r·min-1离心15 min,收集上清液,-80 ℃条件保存备用.
1.3 RT-PCR检测参考NCBI的CV777基因序列,使用软件Primer Premier 5.0设计1对检测PEDV的特异性引物.上游引物序列为: 5'-TCGCTTTCAGCATCCTTAT-3',下游序列为: 5'-CTTGGCGACTGTGACGAAA-3',由金唯智公司合成.依据RNA抽提试剂盒操作手册抽提病毒RNA,最后以40 μL ddH2O洗脱,取10 μL作为模板进行反转录.反转录体系为: 10 μL RNA模板,2 μL随机引物,70 ℃变性10 min; 随后分别加入1 μL RNA抑制剂,1 μL M-MLV反转录酶,2 μL dNTP,4 μL 5 × Reverse Transcriptase M-MLV Buffer,反转录1.5 h; 最后72 ℃ 15 min以终止反应.25 μL PCR体系为: 12.5 μL Taq DNA复制酶,0.5 μL上(下)游引物(10 pmol),1.5 μL cDNA模板,10 μL ddH2O;反应条件为95 ℃预变性5 min; 95 ℃变性10 s,55 ℃退火15 s,72 ℃延伸30 s(30个循环); 72 ℃延伸10 min.反应样品在0.01 g·mL-1琼脂糖凝胶进行检测.当结果出现与预期大小相似条带时,进行切胶回收,连接T载体并进行大肠埃希菌转化、克隆及抽提质粒,具体步骤参照试剂盒说明书.抽提的质粒由金唯智公司测序.
1.4 病毒的分离经RT-PCR检测为PEDV阳性的样品,进行病毒的分离与培养.以12 000 r·min-1离心样品液15 min,将上清液以0.22 μm针式滤器过滤除菌.取长满70%~80%的Vero单层细胞(25 cm2细胞培养瓶),加入1 mL的过滤上清液孵育1.5 h,孵育液中加入胰酶至终质量浓度为8 × 10-6 g·mL-1; 吸除病毒孵育液,加入含4 × 10-6g·mL-1的胰酶不含血清的维持液,放置于37 ℃、体积分数为5%的CO2细胞培养箱中培养,逐日观察病变情况,盲传3代.当细胞产生特征性病变时,接毒液减少至100 μL,并在病变85%左右收集病毒细胞培养液.收集细胞培养液时反复冻融3次,4 ℃ 4 500 r·min-1离心15 min除去细胞碎片,取上清液-80 ℃条件保存备用.
1.5 病毒的鉴定 1.5.1 细胞培养物RT-PCR检测将细胞培养物在-80 ℃冻融3次.4 ℃ 4 500 r·min-1离心15 min去除细胞碎片.上清液保存在-80 ℃条件下,取其中一部分抽提RNA进行RT-PCR检测,方法如“1.3”.
1.5.2 细胞病变的观察病毒在盲传几代之后,能在显微镜下看到明显的细胞病变(CPE).用倒置显微镜观察并记录下PEDV在Vero细胞上的CPE形成情况.
1.5.3 间接免疫荧光检测M蛋白将胰酶消化的细胞接种6孔培养板,当Vero细胞铺满至70% ~ 80%时弃掉原培养液,用孵育液(含8 × 10-6 g·mL-1胰酶的DMEM培养液)清洗2次.每孔接种病毒液10 μL,加入990 μL孵育液于37 ℃、体积分数为5% CO2的培养箱中孵育1.5 h,留1孔作为对照,直接加入1 mL孵育液.孵育完成之后弃掉孵育液,每孔再加入2 mL维持液(含有4 × 10-6g·mL-1的胰酶、无血清的DMEM培养液).在37 ℃、体积分数为5% CO2的培养箱中培养24 h,进行间接免疫荧光试验,检测PEDV的M蛋白.然后进行DAPI染色,观察细胞核的变化,主要步骤为:用预冷PBS清洗3次,以体积分数为4 %的多聚甲醛常温固定20 min; 清洗后孵育抗PEDV M蛋白的鼠源单克隆抗体,4 ℃过夜; PBS清洗一抗后孵育羊抗鼠FITC标记二抗,避光作用1 h; PBS清洗3次后在倒置荧光显微镜下观察.如需核染,用DAPI染液作用5 min,PBS清洗3次后再观察。
1.6 病毒基因组核苷酸序列分析病毒全基因组测序完成并上传至GenBank数据库[16].利用DNAStar7.1基因分析软件对分离株的 S 基因核苷酸序列与国内外各参考毒株相同区域核苷酸序列进行相似性分析,并使用MEGA6.0绘制分离株与各参考毒株的 S 基因序列的系统进化图谱.以下参考株序列均来自GenBank数据库,参考毒株分别为: HB-2012-1(JX435302),BJ-2012-1(JX435299),BJ- 2012-2(JX825706),SH(JQ771751),SD-M(JX560761),JS2008 (KC109141),CH/S (JN547228),AJ1102 (JX188454),LC (JX489155),VN/KCHY-3110113 (KJ960180),VN/JFP1013-1(KJ960178),VN/VAP113- 1(KJ960179),USA/Texas128 /2014 (KJ645697),USAIowa107-2013 (KJ645696),USA-Minnesota100-2013 (KJ645691),USA-Colorado-2013 (KF272920),USAKansas125-2014(KJ645701),YS(JQ771751),attenuated DR13 (JQ023162),SM98 (GU937797),virulent _ DR13(JQ023161),CV777(JN599150).
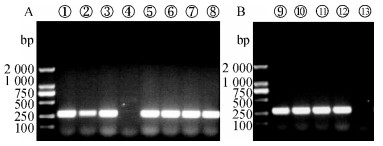
2 结果 2.1 粪便和细胞培养物的RT-PCR检测结果对采集的粪便样品进行RT-PCR检测,结果出现1条约285 bp的DNA片段,其大小与预计片段相似(图 1A).同时,将细胞培养物进行RT-PCR检测,扩增出大小相似的DNA片段(图 1B).分别将2次测序的结果在NCBI上进行BLAST,发现与PEDV序列相似性最高.由此可初步断定分离的毒株为PEDV.
|
图 1 采集粪便样品和细胞培养物RT-PCR鉴定结果 Figure 1 Identification results of RT-PCR of fecal samples and cell culture supernatant A:采集粪便样品,B:细胞培养物; ①~⑧为采集粪样的8个样品,除样品④检测为阴性外,其余为阳性; ⑨~⑫分别是培养2、4、10、20代的细胞培养物检测结果,⑬为阴性对照. |
病毒传至第5代时能观察到典型病变,传至第20代时能在Vero上增殖稳定.随着传代代次的增加,出现CPE的时间变短.稳定增殖后,细胞在18 h可观察到明显的病变,24 h病变接近50%,36 h时细胞几乎完全病变,并有成片细胞脱落(图 2).与刚开始出现病变的低代次PEDV相比,完全病变时间缩短近一半.开始病变时几个细胞边界分辨不清,与周围的正常细胞形态差异明显(图 2B); 随时间推移只有少量细胞形态完整,大量细胞皱缩融合(图 2C),最终成片脱落(图 2D).DAPI染色结果显示,感染病毒的Vero细胞确有核聚集(图 3B),而对照组(图 3D)没有,细胞核均一分布,与Hofmann等[11]描述的病变结果一致.由此可进一步断定分离的毒株是PEDV.

|
图 2 感染病毒Vero细胞不同时间的细胞病变(400 ×) Figure 2 Cytopathic effect in infected-Vero cells at different time point (400 ×) A为正常细胞对照组; B、C、D分别是接毒后18、24、36 h时细胞发生病变的情况. |
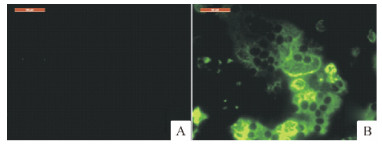
使用抗PEDV M蛋白的单克隆抗体对传12代的PEDV细胞培养毒进行间接免疫荧光鉴定,接种病毒24 h进行一抗孵育,结果显示感染的Vero细胞有明显的荧光信号,而对照组没有荧光显现(图 3、4).此结果表明分离的毒株即是PEDV.

|
图 3 间接免疫荧光(IFA)检测M蛋白与DAPI核染结果 Figure 3 The results of detecting M protein by indirect fluorescent antibody (IFA) assay and nuclear staining by DAPI A、B分别为感染病毒细胞的IFA及DAPI染色结果; C、D分别为对照组细胞的IFA及DAPI染色结果; 标尺: 50 μm. |
|
图 4 间接免疫荧光检测PEDV M蛋白结果 Figure 4 The results of detecting M protein of PEDV by indirec fluorescence antibody assay A为阴性对照,未检测到任何荧光信号; B为感染细胞组,可检测到明显的绿色荧光信号,并可看到多个细胞融合; 标尺: 50 μm. |
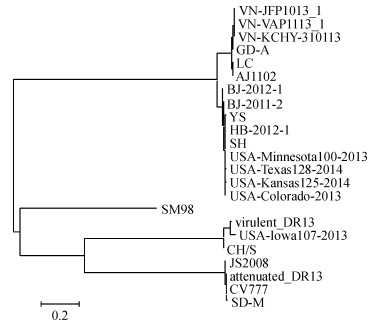
分离株GD-A与不同PEDV毒株间 S 基因相似性比较发现(图 5),与毒株LC和AJ1102相似性最高,都为99.2%;与毒株SM98和CH /S相似性最低,分别为92.5%与93.2%.与经典毒株CV777相比较,相似性较低,为93.3%.与越南分离株VN KCHY-310113和VN-JFP1013_1相似性较高,分别为98.7%和98.5%;与美国分离株相似性变化较大,如与毒株USA-Iowa107-2013和USA-Minnesota100-2013的相似性分别为94.7%和98.0%.各参考毒株的S基因系统进化树显示,GD-A与2个国内分离株LC、AJ1102和3个越南分离株位于同一个进化分支上,与近年国内的分离株及大部分美国分离株位于同一个大的分支上; 但与韩国株SM98、virulent_DR13,国内早期分离株CH /S、JS2008,经典毒株CV777等位于不同的分支上.从该分析结果来看,所分离的毒株与疫苗株如attenuated_DR13和CV777在不同分支,提示该分离株不是疫苗株.
|
图 5 GD-A与不同猪流行性腹泻病毒参考株间 S 基因遗传进化树关系 Figure 5 Phylogenetic tree based on S gene for GD-A and other reference strains of porcine epidemic diarrhea virus from GenBank database |
PEDV在欧洲首次发现时,产生与猪传染性胃肠炎相似的临床症状[17].此病在欧洲流行时,对母猪和仔猪致病力相似,但整体死亡率表现不高.与近年在亚洲和北美洲的情况有很大的差异性.从国内外报道PEDV的结果可知,感染母猪表现轻微临床症状,食欲下降,精神抑郁,最终恢复; 而感染仔猪主要表现呕吐腹泻,肠道解剖发现肿大透明,内有未消化的乳凝块,最终严重脱水而死,死亡率较高可达100%[3, 8-9, 18].此外,此病在猪场反复流行,传播速度快,而对该病毒的生物学特性了解很少,对于如何预防和控制该病毒的流行,仍是一个很大的挑战.
S 基因编码的纤突蛋白位于病毒粒子的表面,能诱导宿主产生特异性中和抗体.与经典毒株CV777相比,分离株GD-A的 S 基因在487~492位点有6个碱基缺失; 在167位点有1个碱基插入,在428 ~ 430位点有3个位点插入,在175~185位点连续插入11个碱基,另外其他位点存在多个点突变[16]. GD-A与其他参考毒株相比较,与3个越南毒株、近年国内分离株及大部分美国分离株具有相同的碱基缺失、插入与突变情况,遗传进化分析也同属于一个大分支,提示这些病毒可能有共同的祖先; 而与CV777基因变化相似的韩国毒株attenuated-DR13、SM98,美国分离株USA-Iowa107-2013及国内早期分离株JS2008、CH /S在遗传进化分析属于另一分支,这提示近年的流行毒株与疫苗株(CV777)关系较远.同时,分离株不同位置出现的碱基突变、缺失与插入对病毒的生物学特性影响有待进一步的研究.
从前人对近年PEDV的分子流行病学研究结果发现,亚洲各国PEDV流行毒株与欧洲及疫苗株之间关系较远,而这些国家之间分离的流行毒株关系相对较近[3, 19-20].同时,近期分离的毒株与几年前分离的毒株差异较大[21-22].这说明PEDV基因组在环境压力下一直处于变异的状态.这可能是导致近年PEDV的大规模爆发,而不能以疫苗接种等措施控制此病流行的主要原因.目前,对此病没有有效的治疗措施,商品化的疫苗在当前不能为猪群提供有效的保护,所以在发生疾病的猪场中隔离感染、发病猪只,做好生物安全措施是目前最好的方法[23].
3.2 PEDV在细胞的培养PEDV具有在体外难以培养的特性.到目前为止,Vero细胞是分离PEDV最敏感的细胞系,连续传代后能在此细胞系上稳定地生长与增殖,但在培养时需要培养液中添加适量的胰酶[11, 15, 23].本试验采用8 × 10-6g·mL-1的孵育液和4 × 10-6 g·mL-1的维持液在Vero细胞中分离病毒,在前代次并未观察到病变,自第5代后产生典型的细胞病变,连续传代至20代后,病毒可在细胞上稳定增殖.该试验结果与其他人的研究结果[11]相似.但也有报道称在初次接种细胞时即可产生明显的病变[23],只是每次产生病变的时间差异很大,这说明毒株间生物学特性的差异影响其在Vero细胞增殖的适应过程.本试验分离PEDV病毒的成功,为后续的病毒生物学特性,疫苗的研发都提供了试验与理论基础.
| [1] |
SONG D, PARK B. Porcine epidemic diarrhoea virus:A comprehensive review of molecular epidemiology, diagnosis and vaccines[J]. Virus Genes, 2012, 44(2): 167-175. DOI:10.1007/s11262-012-0713-1 (  0) 0) |
| [2] |
WOOD E N. An apparently new syndrome of porcine epidemic diarrhea[J]. Vet Rec, 1977, 100(12): 243-244. DOI:10.1136/vr.100.12.243 (  0) 0) |
| [3] |
PURANAVEJA S, POOLPERM P, LERTWATCHA-RASARAKUL P, et al. Chinese-like strain of porcine epidemic diarrhea virus, Thailand[J]. Emerg Infect Dis, 2009, 15(7): 1112-1115. (  0) 0) |
| [4] |
CHAE C C, KIM O O, CHOI C C, et al. Prevalence of porcine epidemic diarrhea virus and transmissible gastroenteritis virus infection in Korean pigs[J]. Vet Rec, 2000, 147(21): 606-608. DOI:10.1136/vr.147.21.606 (  0) 0) |
| [5] |
SUEYOSHI M, TSUDA T, YAMAZAKI K, et al. An immunohistochemical investigation of porcine epidemic diarrhea[J]. J Comp Pathol, 1995, 113(1): 59-67. (  0) 0) |
| [6] |
CHEN J F, WANG C B, SHI H Y, et al. Complete genome sequence of a Chinese virulent porcine epidemic diarrhea virus strain[J]. J Virol, 2011, 85(21): 11538-11539. DOI:10.1128/JVI.06024-11 (  0) 0) |
| [7] |
WANG L, BYRUM B, ZHANG Y. New variant of porcine epidemic diarrhea virus, United States, 2014[J]. EmergInfect Dis, 2014, 20(5): 917-919. (  0) 0) |
| [8] |
SUN R Q, CAI R J, CHEN Y Q, et al. Outbreak of porcine epidemic diarrhea in suckling piglets, China[J]. Emerg Infect Dis, 2012, 18(1): 161-163. DOI:10.3201/eid1801.111259 (  0) 0) |
| [9] |
LI W T, LI H, LIU Y B, et al. New variants of porcine epidemic diarrhea virus, China, 2011[J]. Emerg Infect Dis, 2012, 18(8): 1350-1353. DOI:10.3201/eid1803.120002 (  0) 0) |
| [10] |
殷震, 刘景华. 动物病毒学[M]. 2版. 北京: 科学出版社, 1996: 688-690.
(  0) 0) |
| [11] |
HOFMANN M, WYLER R. Propagation of the virus of porcine epidemic diarrhea in cell culture[J]. J Clin Microbiol, 1988, 26(11): 2235-2239. (  0) 0) |
| [12] |
张志, 李岚, 王赛赛, 等. 猪流行性腹泻新毒株的分离与鉴定[J]. 中国兽医学报, 2012, 32(10): 1423-1428. (  0) 0) |
| [13] |
庄金秋, 王金良, 梅建国, 等. 猪流行性腹泻病毒SDbz株的分离与鉴定[J]. 动物医学进展, 2013, 33(5): 650-653. (  0) 0) |
| [14] |
张桂红. 猪流行性腹泻病毒地方株LJB/03株分离鉴定[D]. 哈尔滨: 东北农业大学, 2008.
(  0) 0) |
| [15] |
付梦瑾, 朱玲, 吴云飞, 等. 猪流行性腹泻病毒的分离鉴定及增值规律[J]. 中国兽医科学, 2013, 43(11): 1133-1139. (  0) 0) |
| [16] |
FAN H Y, ZHANG J, YE Y, et al. Complete genome sequence of a novel procine epidemic diarrhea virus in south China[J]. J Virol, 2012, 86(18): 10248-10249. DOI:10.1128/JVI.01589-12 (  0) 0) |
| [17] |
PENSAERT M B, DE BOUCK P. A new coronavirus-like particle associated with diarrhea in swine[J]. Arch Virol, 1994, 58(3): 243-247. (  0) 0) |
| [18] |
JUNG K, WANG Q H, KELLY A, et al. Pathology of US porcine epidemic diarrhea virus strain PC21A in gnotobiotic pigs[J]. Emerg Infect Dis, 2014, 20(4): 661-665. DOI:10.3201/eid2004.130960 (  0) 0) |
| [19] |
FAN J H, LI Y J. Cloning and sequence analysis of the M gene of porcine epidemic diarrhea virus LJB/03[J]. Virus Genes, 2005, 30(1): 69-73. DOI:10.1007/s11262-004-4583-z (  0) 0) |
| [20] |
KMLG T J, SEO J E, KHN D H, et al. Cloning and sequence analysis of the Korean strain of spike gene of porcine epidemic diarrhea virus and expression of its neutralizing epitope in plants[J]. Protein Expr Purif, 2009, 39(7): 602-607. (  0) 0) |
| [21] |
PARK S J, KIM H K, SONG D S, et al. Molecular characterization and phylogenetic analysis of porcine epidemic diarrhea virus field isolates in Korea[J]. Arch Virol, 2011, 156(4): 577-585. DOI:10.1007/s00705-010-0892-9 (  0) 0) |
| [22] |
PARK S J, MOON H J, YANG J S, et al. Sequence anal ysis of the partial spike glycoprotein gene of pocine epidemic diarrhea viruses isolated in Korea[J]. Virus Genes, 2007, 35(2): 321-332. DOI:10.1007/s11262-007-0096-x (  0) 0) |
| [23] |
CHEN Q, LI G W, STASKO J, et al. Isolation and characterization of porcine epidemic diarrhea viruses associated with the 2013 disease outbreak in US swine[J]. J Clin Microbiol, 2014, 52(1): 234-243. DOI:10.1128/JCM.02820-13 (  0) 0) |
 2015, Vol. 36
2015, Vol. 36


