2. 延边大学 动物科学学院,吉林 延吉 133002
2. College of Animal Science, Yanbian University, Yanji 133002, China
动物转基因技术在基因功能的阐明、农业动物的遗传育种、医学模型的建立等方面有着广阔的应用前景,寻求一种简便、有效和低成本的转基因方法具有重要意义.精子可以作为外源基因转移载体来制备转基因动物.相对于其他动物转基因方法而言,它的突出优点是利用精子的自然属性克服了人为机械操作给胚胎带来的损伤,提高了转基因效率,而且操作简单、成本低廉,因而受到关注.精子介导基因转移技术的方法最早由Brackett等[1]证实.2001年,Pfeifer等[2]将慢病毒载体用于转基因动物制备的研究.2002年Lois等[3]报道了慢病毒载体不仅能整合到宿主基因组中,还能有效感染处于静息期的非分裂细胞,外源基因在动物体内能有效表达,且可稳定传代.慢病毒载体法在基因转导效率、目的基因表达率、可操作性等方面有显著的优势.结合两者的优势,本课题组建立了以精子与慢病毒共孵育来制备转基因动物的新方法.Zhang等[4]通过慢病毒颗粒与洗去精清的猪精子共孵育,成功制备出了SST基因表达下调的转基因猪的模型.蔡伟光等[5]优化了慢病毒与精子共孵育条件,成功制备得到转基因猪,即本试验中的G0代.本研究在已获得的G0代转基因猪基础上,通过扩群繁殖,检测转基因的整合及遗传效率,为精子与慢病毒共孵育法制备转基因动物的方法提供试验依据,为建立高效、简单、经济的转基因猪的制备方法奠定基础.
1 材料与方法 1.1 试验材料 1.1.1 试验动物本试验所用的转基因猪为长白猪,由广东温氏食品集团广东华农温氏畜牧股份有限公司森村实验猪场饲养管理.
1.1.2 试验耗材与试剂限制性内切酶Xba I、BamH I,DNA提取试剂盒、质粒提取试剂盒、DNA凝胶回收试剂盒、无内毒素中提取试剂盒均为美国OMEGA公司产品.DIG-High Prime DNA labeling and Detection Starter kitⅡ试剂盒、尼龙膜为美国Roche公司产品;PCR扩增引物由上海生工生物技术有限公司合成.
1.2 试验方法 1.2.1 转基因猪的杂交方案华南农业大学动物生理生化实验室前期通过精子与慢病毒共孵育法制备获得了转基因猪G0代,1头公猪和1头母猪,分别为G041♂和G018♀.在此基础上扩繁,G041♂分别和G018♀、阴性1♀、阴性2♀和阴性3♀(分别为G0亲代41♂ × 18♀、41♂ ×阴性1♀、41♂ ×阴性2♀和41♂ ×阴性3♀)进行杂交配种,获得G1代.之后选取G0亲代41♂ ×阴性2♀的杂交G1代45♂和47♂、41♂ ×阴性3♀的杂交G1代18♀以及野生阴性4♀为G1亲本进行杂交配种(分别为G1亲代45♂ × 18♀和47♂ ×阴性4♀).配种后得到转基因子代猪G1、G2代.
1.2.2 转基因猪子代中阳性个体的筛选采取转基因猪子代个体的耳组织,提取DNA.依据慢病毒载体pshRNA-copGFP Lentivector上的GFP基因(绿色荧光蛋白基因)序列(共649 bp),利用软件Primer 5设计引物649,上游引物:5′ -CTACGGCTTCTAC-CACTTCG -3′,下游引物:5′ -GCAGCGTATCCACAT-AGCGT -3′.PCR模板为耳组织DNA,阳性个体由引物Primer 649 PCR法扩增筛选.反应条件为:94 ℃预变性5 min;94 ℃变性30 s,58 ℃退火30 s,72 ℃延伸30 s,35个循环;72 ℃延伸10 min,4 ℃条件下保存.取5 μL PCR产物,0.008 g·mL-1琼脂糖凝胶,电泳30 min,Bio-rad凝胶成像系统检测电泳结果.
1.2.3 转基因阳性猪各组织的PCR及RT-PCR检测采集12头转基因猪(分别为2头G0代转基因猪18♀和41♂,3头G1代转基因猪G118♀、G140♀、G137♂以及7头G2代转基因猪G21♂、G24♂、G 25♂、G26♀、G27♀、G28♂、G29♀)的心、肝、脾、肺、肾、胃、肠、胰、肌肉、卵巢/睾丸、垂体、下丘脑、耳共13种组织样品,分别提取DNA;同时,用TRIZOL法提取胃、垂体、下丘脑组织的RNA,用DNAase消化样品中的DNA后,用随机引物OLigo dT(18)将RNA反转录成cDNA.
应用引物设计软件Primer 5,针对慢病毒载体pshRNA-copGFP Lentivector上的GFP基因设计引物649和引物108.采用20 μL体系,对反转录得到的cDNA进行巢式PCR检测.第1轮PCR反应预期扩增出来的片段为649 bp.第2轮PCR反应预期扩增出来的片段为108 bp.PCR反应条件如“1.2.2”所述.引物649序列同“1.2.2”.引物108序列的上游引物:5′ -CTACGGCTTCTACCACTTCG-3′,下游引物:5′ -CGTCCTCGTACTTCTCGATG -3′.
2 结果与分析 2.1 G1、G2代猪中转基因阳性个体的筛选用引物649对59头所得转基因后代进行PCR检测,结果显示,在G0亲代41♂ × 18♀所产的11头仔猪中4头为阳性,阳性率为36.36%(4/11).G0亲代41♂ ×阴性1♀所产的13头仔猪中3头为阳性,阳性率为21.43%(3/14).G0亲代41♂ ×阴性2♀所产的18头仔猪中5头为阳性,阳性率为27.78%(5/18).G0亲代41♂ ×阴性3♀所产的16头仔猪中3头为阳性,阳性率为18.75%(3/16).G1代的平均阳性率为25.42%(15/59).G1亲代45♂ × 18♀所产的15头仔猪中7头为阳性,阳性率为46.67%(7/15).G1、G2代PCR检测结果分别见图 1.
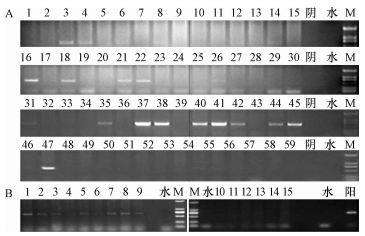
|
图 1 转基因猪子代个体的PCR检测电泳图 Figure 1 PCR electrophoresis of offspring of G0 transgenic pigs 阴:阴性对照为野生型个体;阳:阳性对照;水:空白对照;M:DNA marker DL2000(条带从上到下依次是2 000、1 000、750、500、250、100 bp);A:转基因猪G1代阳性个体的PCR检测结果,1 ~ 59:G1代转基因猪;B:转基因猪G2代阳性个体的PCR检测结果,1 ~ 15:G2代转基因猪. |
对12头经PCR检测为阳性的猪进行采样,其中G0代2头,G1代3头,G2代7头,每头猪分别取心、肝、脾、肺、肾、胃、肠、胰、卵巢/睾丸、肌肉、垂体、下丘脑和耳组织,提取各组织DNA,用引物649进行PCR检测(图 2).结果表明通过慢病毒与精子共孵育法制备得到的转基因猪,外源基因存在于各种组织中,并遗传给后代.
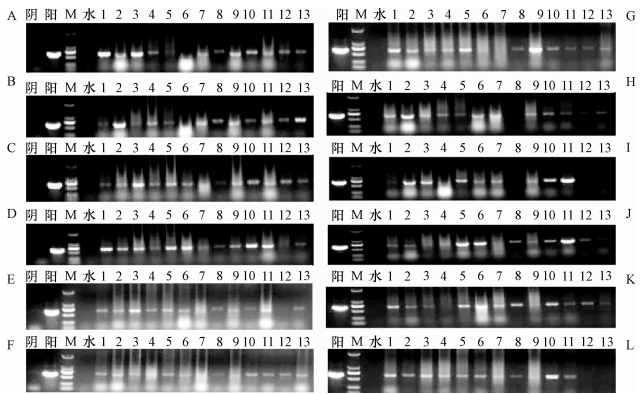
|
图 2 猪不同组织中外源DNA基因的PCR检测 Figure 2 PCR detection of foreign gene existance in pig tissues 阴:阴性对照;水:空白对照;阳:阳性对照;M:DNA marker DL 2000(条带从上到下依次是2 000、1 000、750、500、250、100 bp);1:心;2:肝;3:脾;4:肺;5:肾;6:胃;7:肠;8:胰;9:卵巢/睾丸;10:肌肉;11:垂体;12:下丘脑;13:耳朵;A:G0 18♀;B:G0 41♂;C:G1 40♀;D:G1 18♀;E:G1 37♂;F:G21♂;G:G24♂;H:G25♂;I:G26♀;J:G27♀;K:G28♂;L:G29♀. |
对上述12头阳性转基因猪的胃、垂体、下丘脑组织提取RNA,转录成cDNA后,先用引物649扩增样品(图 3A),再用引物108扩增PCR产物(图 3B).结果显示,GFP基因在胃中表达的有G018♀、G041♂、G137♂、G21♂、G24♂、G25♂、G26♀、G28♂、G29♀共9头转基因猪,在垂体中表达的有G018♀、G041♂、G118♀、G137♂、G21♂、G24♂、G25♂、G26♀、G27♀共9头转基因猪,能在下丘脑中表达的有G118♀、G137♂、G24♂、G25♂、G26♀、G29♀共6头转基因猪.在检测的组织样品中,66.67%(24/36)的组织均有外源基因表达.PCR结果中除目的条带108 bp存在外,还存在一些非特异性条带,原因可能是由于在第2轮PCR过程中,体系中存在引物649与引物108共4条引物,从而扩增出不同大小的片段.结果表明,外源基因不仅整合进入猪基因组,且能转录表达.
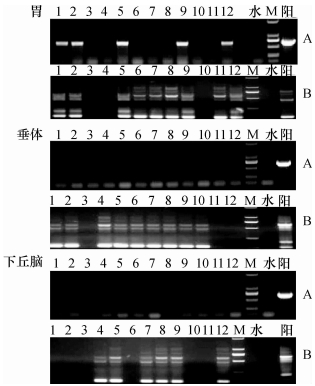
|
图 3 转基因阳性个体中胃、垂体和下丘脑组织的RT-PCR检测 Figure 3 RT-PCR detection of foreign gene expression in stomach, pituitary and hypothalamus of transgeinc pigs 1:G018♀;2:G041♂;3:G140♀;4:G118♀;5:G137♂;6:G21♂;7:G24♂;8:G25♂;9:G26♀;10:G27♀;11:G28♂;12:G29♀;水:空白对照;M:DNA marker DL 2000(条带从上到下依次是2 000、1 000、750、500、250、100 bp);阳:阳性对照. |
为了检测精子与慢病毒孵育制备的转基因猪中外源基因的遗传效率,本试验对所得到的转基因猪子代进行了分子检测.结果显示,G1代和G2代的PCR阳性率分别为25.42%(15/59)、46.67%(7/15).在转基因猪及子代中不同组织,如心、肝、脾、肺、肾、胃、肠、胰、卵巢/睾丸、肌肉、垂体、下丘脑和耳中均存在外源转基因.表明外源基因整合到了猪的基因组中,且可以遗传给下一代.Aigner等[6]在研究转基因小鼠外源基因的遗传稳定性时发现,在转基因小鼠的遗传过程中,酪氨酸激酶基因的整合位点是高度稳定的,但在不同世代、不同个体中,基因的拷贝数存在差异.通常情况下,外源基因在受体基因组中的整合位点是随机的,但一些研究结果表明整合位点在染色体上也存在一定规律.Nakanishi等[7]利用FISH技术来精确定位142个品系的转基因小鼠整合位点.统计分析结果显示,外源基因在小鼠基因组上的整合位点,位于2号和11号染色体上的概率明显比较高,且在染色体中呈集中分布,而不是均匀分布.外源基因在受体基因组中可能会有部分序列缺失,或发生重排,或某些碱基突变导致某些酶切位点丢失,或被受体基因组DNA片段所分隔[8].这种外源基因以多拷贝串联的形式在受体基因组中进行整合,使其以重复序列形式存在,容易导致DNA复制过程中由于复制不完全,从而丢失外源基因拷贝数.本试验的结果也表明了这种外源基因丢失的现象.此外,通过RT-PCR检测转基因猪胃、垂体和下丘脑3个组织中外源基因的表达情况,结果显示外源基因在多数组织中可以表达,表明外源基因不仅能整合进入猪基因组,并且能转录表达.
但少数组织中也存在没有表达的现象,而影响这种外源基因(如GFP)未能表达的因素很多.(1)外源基因的整合位点会影响其整合结果和表达效率.比如重复序列诱导的基因沉默效应(Repeat-in-duced gene silencing,RIGS),它是指多拷贝的外源基因在同一整合位点会形成重复序列,这些重复序列会相互影响,使外源基因呈低水平表达.换言之,单拷贝的或有限几个拷贝的外源基因的整合形式有可能会利于外源基因的表达.Williams等[9]的研究结果与此相似.外源基因的整合位点还与其遗传稳定性相关.Migliaccio等[10]筛选转基因细胞K256时发现,外源基因可以在细胞基因组的许多位点进行整合并表达,但只有整合在少数几个特别的位点时才会稳定地遗传,绝大多数其他整合位点上的外源基因丢失.而Wang等[11]却发现在转基因小鼠中,外源基因能稳定地传到第7代,而且通过自交或回交,小鼠体内外源基因的拷贝数能累积并逐渐增加,遗传效率表现良好.(2)外源基因表达沉默与DNA甲基化有关.Doerfler[12]检测外源基因的启动子及编码区的甲基化水平时发现,在转基因动物中,甲基化会降低外源基因的表达.Hofmann等[13]研究发现,在得到的转基因猪G1代中,有1/3个体外源基因表达低下,这是由于超甲基化引起的.Baup等[14]研究发现,由于外源基因甲基化水平在不同的细胞中是不一样的,所以在转基因小鼠的同种组织中,外源基因的表达也会存在差异.虽然慢病毒载体可以较方便地制备转基因动物,但也容易被受体基因组甲基化,而导致外源基因表达沉默.(3)位置效应也是基因沉默的主要原因,一般是指由于外源基因整合进入受体基因时,所处的染色体位置不一样,因而其表达存在差异的现象.Singh等[15]利用MuDR转座子载体对玉米进行研究,发现位置效应对外源基因的表达有非常重要的影响.当外源基因的整合位点与异染色质相临时,花斑效应就会出现;但当外源基因的整合位点位于转录比较活跃的位点,花斑效应便会消失.这些结果说明外源基因的表达沉默与基因自身无关,而是由与其整合位点的位置所决定[16-17].
本研究的结果初步揭示了在精子与慢病毒孵育法制备的转基因猪中,外源基因可以遗传给后代,但随着代数的增加,外源基因存在丢失现象,且代数多丢失也增加;同时也存在表达沉默的现象.造成这些现象的具体分子机制还有待于进一步的探讨.
| [1] |
BRACKETT B G, BARANSKA W, SAWICKI W, et al. Uptake of heterologous genome by mammalian spermatozoa and its transfer to ova through fertilization[J]. Proc Natl Acad Sci USA, 1971, 68(2): 353-357. DOI:10.1073/pnas.68.2.353 (  0) 0) |
| [2] |
PFEIFER A, KESSLER T, YANG M, et al. Transduction of liver cells by lentiviral vectors:Analysis in living animals by fluorescence imaging[J]. Mol Ther, 2001, 3(3): 319-322. DOI:10.1006/mthe.2001.0276 (  0) 0) |
| [3] |
LOIS C, HONG E J, PEASE S, et al. Germline transmission and tissue-specific expression of transgenes delivered by lentiviral vectors[J]. Science, 2002, 295(5556): 868-872. DOI:10.1126/science.1067081 (  0) 0) |
| [4] |
ZHANG Y L, XI Q Y, DING J H, et al. Production of transgenic pigs mediated by pseudotyped lentivirus and sperm[J]. PLoS One, 2012, 7(4): e35335. DOI:10.1371/journal.pone.0035335 (  0) 0) |
| [5] |
蔡伟光, 习欠云, 肖敏, 等. 精子与慢病毒共孵育条件的优化以及转基因猪的制备[J]. 中国农业科学, 2013, 46(9): 1903-1914. DOI:10.3864/j.issn.0578-1752.2013.09.018 (  0) 0) |
| [6] |
AIGNER A, JÄGER M, PASTERNACK R, et al. Purification and characterization of cysteine-S-conjugate Nacetyltransferase from pig kidney[J]. Biochem J, 1996, 317(1): 213-218. DOI:10.1042/bj3170213 (  0) 0) |
| [7] |
NAKANISHI T, KUROIWA A, YAMADA S, et al. FISH analysis of 142 EGFP transgene integration sites into the mouse genome[J]. Genomics, 2002, 80(6): 564-574. DOI:10.1006/geno.2002.7008 (  0) 0) |
| [8] |
吴波, 朱作言. 转基因动物整合位点的研究进展[J]. 遗传, 2003, 25(1): 77-80. (  0) 0) |
| [9] |
WILLIAMS A, HARKER N, KTISTAKI E, et al. Position effect variegation and imprinting of transgenes in lymphocytes[J]. Nucleic Acids Res, 2008, 36(7): 2320-2329. DOI:10.1093/nar/gkn085 (  0) 0) |
| [10] |
MIGLIACCIO A R, BENGRA C, LING J, et al. Stable and unstable transgene integration sites in the human genome:Extinction of the green fluorescent protein transgene in K562 cells[J]. Gene, 2000, 256(1/2): 197-214. (  0) 0) |
| [11] |
WANG Y, SONG Y T, LIU Q, et al. Quantitative analysis of lentiviral transgene expression in mice over seven generations[J]. Transgenic Res, 2010, 19(5): 775-784. DOI:10.1007/s11248-009-9355-z (  0) 0) |
| [12] |
DOERFLER W. DNA methylation:Eukaryotic defense against the transcription of foreign genes?[J]. Microb Pathog, 1992, 12(1): 1-8. DOI:10.1016/0882-4010(92)90060-2 (  0) 0) |
| [13] |
HOFMANN A, KESSLER B, EWERLING S, et al. Epigenetic regulation of lentiviral transgene vectors in a large animal model[J]. Mol Ther, 2006, 13(1): 59-66. DOI:10.1016/j.ymthe.2005.07.685 (  0) 0) |
| [14] |
BAUP D, FRAGA L, PERNOT E, et al. Variegation and silencing in a lentiviral-based murine transgenic model[J]. Transgenic Res, 2010, 19(3): 399-414. DOI:10.1007/s11248-009-9318-4 (  0) 0) |
| [15] |
SINGH J, FREELING M, LISCH D. A position effect on the heritability of epigenetic silencing[J]. PLoS Genet, 2008, 4(10): e1000216. DOI:10.1371/journal.pgen.1000216 (  0) 0) |
| [16] |
EBERT A, SCHOTTA G, LEIN S, et al. Su(var)genes regulate the balance between euchromatin and heterochromatin in Drosophila[J]. Genes Dev, 2004, 18(23): 2973-2983. DOI:10.1101/gad.323004 (  0) 0) |
| [17] |
CLARK A J, BISSINGER P, BULLOCK D W, et al. Chromosomal position effects and the modulation of transgene expression[J]. Reprod Fertil Dev, 1994, 6(5): 589-598. DOI:10.1071/RD9940589 (  0) 0) |
 2015, Vol. 36
2015, Vol. 36


