2. 广东省动植物与食品进出口技术措施研究重点实验室,广东 广州 510623;
3. 广州出入境检验检疫局,广东 广州 510623;
4. 华南农业大学 兽医学院,广东 广州 510642
2. Guangdong Key Laboratory of Import and Export Technical Measures of Animal, Plant and Food, Guangzhou 510623, China;
3. Guangzhou Entry-Exit Inspection and Quarantine, Guangzhou 510623, China;
4. College of Veterinary Medicine, South China Agricultural University, Guangzhou 510642, China
牛脊椎畸形综合征(Complex vertebral malformation,CVM)是由常染色体上SLC35A3基因单碱基突变(G→T)引起的隐性遗传病,该基因隐性纯合(CV /CV)时奶牛可致死.病牛的主要临床症状为脊椎弯曲畸形、两前腿筋腱缩短、颈短和心脏畸形,同时出现一系列综合症状[1].妊娠奶牛还会发生早期流产、胎牛死胎、早产,严重影响荷斯坦牛的繁殖力,延长产犊间隔,增加母牛淘汰率.CVM的遗传缺陷最早由丹麦科学家于2001年发现报道[2],引起了各国奶牛育种协会和育种工作者的重视.美国、英国、日本、瑞典、波兰和德国等国家相继报道了在荷斯坦牛群中存在CVM有害基因[3-8].近年,在中国荷斯坦牛群中也检出一定数量的CVM基因携带者[9-10].
CVM基因隐性纯合时表现病征,CVM基因携带者表现正常,但CVM基因携带者公牛在人工授精时会传播CVM缺陷基因,一旦两杂合体交配,隐性缺陷基因传播风险将大大提高,将给奶牛业造成巨大经济损失.因此有必要建立快速、准确的方法从DNA水平筛查出表型正常的隐性遗传缺陷杂合子,通过剔除CVM基因携带者公牛有效地控制CVM遗传缺陷,减少畜群养殖的经济损失.
目前,CVM有害基因的分子检测方法主要有等位基因特异性扩增方法(Allele-specific polymerase chain reaction,ASPCR)和聚合酶链式反应-引物诱导限制性分析(Polymerase chain reaction-primer introduced restriction analysis,PCR-PIRA)方法[11-12],但成本较高,不适合规模化检测.李艳华等[9]采用聚合酶链式反应-单链构象多态性(PCR-Single strand conformation polymorphism,PCR-SSCP)方法对荷斯坦牛进行诊断分析,但PCR扩增产物需要同时进行琼脂糖凝胶电泳和聚丙烯酰胺电泳来判定基因型.王帅等[10]应用错配PCR突变分析技术(PCR mismatch amplification mutation assay,PCR-MAMA)从国内荷斯坦公牛中检出CVM基因携带者,该方法同样需要采用琼脂糖凝胶电泳判定结果.本研究根据GenBank已发表的CVM基因的SLC35A3序列,设计合成了1对特异性引物和2条TaqMan探针,经过反应条件的优化,建立了奶牛脊椎畸形综合征TaqMan探针荧光PCR检测方法,该方法只需1次加样便可直接从仪器中判定结果、避免了电泳等繁琐操作.
1 材料与方法 1.1 仪器SLAN96P荧光定量PCR仪器为上海宏石医疗科技有限公司产品;高速离心机为湖南湘仪科教仪器有限公司产品;高压蒸汽灭菌器为上海博讯生物仪器有限公司产品.
1.2 样本采集某口岸隔离期的荷斯坦种母牛的全血样品292份,采用天根生化科技(北京)有限公司的血液、细胞、组织基因组DNA提取试剂盒(货号:DP304-02),从血液中提取基因组DNA.按试剂盒说明操作,提取后的基因组DNA于-20 ℃条件下冻存备用.
1.3 质粒CVM基因的野生型质粒(G/G型)、突变型质粒(T/T型)和杂合型质粒(G/T型)由中国检验检疫科学研究院构建与保存.
1.4 引物、探针的设计与合成根据GenBank已发表的奶牛遗传缺陷CVM基因的SLC35A3序列,应用DNAstar软件设计特异性PCR的引物和分别针对CVM基因野生型(SLC35A3-W)和突变型(SLC35A3-M)的探针.引物由上海捷瑞生物科技有限公司合成,探针由上海基康生物技术有限公司合成.具体序列如表 1.
|
|
表 1 荧光PCR的引物与探针 Table 1 Primers and probes for real-time PCR |
分别取1 μL制备的T /T基因型、G/G基因型、G/T基因型混合质粒DNA作为模板,荧光定量PCR反应体系包含2 × Mix buffer 15 μL,上、下游引物各0.5 μL(10 μmol·L-1),2条探针各1 μL(10 μmol·L-1),加入超纯水至30 μL.反应条件为94 ℃预变性3 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸30 s(采集荧光信号FAM和VIC),40个循环;反应在SLAN96P上进行.采用SLAN96P自带的分析软件进行分析.根据循环结束后终点荧光信号值的差异,以FAM和VIC的荧光值为变量,构建双变量散点图,鉴定样品是否为野生型、突变型或杂合型.
1.6 特异性试验为验证此方法检测特异性,以CVM基因的突变型、野生型和杂合型的质粒标准品作为模板,进行TaqMan荧光PCR检测.
1.7 敏感性试验将质粒标准样品测定浓度后,按10倍的倍比稀释,以系列稀释度样品做为模板进行扩增,直至某一稀释度样品检测不出信号,将该浓度作为样品的检测下限,以测试该方法的灵敏度.
1.8 临床血液样品的检测采用所建立的TaqMan探针检测方法对292份荷斯坦种母牛血液样品提取的DNA进行检测.
1.9 探针法的准确性比较对TaqMan探针法分析为G/T基因型的样品以及部分为G/G基因型的样品进行普通测序分析,验证该方法与测序法的符合率,从而评价探针法的准确性.
2 结果与分析 2.1 特异性分析用双探针和引物在优化的反应程序下检测时,突变型、野生型和杂合型质粒标准品能够很好地区分,具体结果如图 1所示.以野生纯合型质粒为模板扩增的结果显示野生型探针出现典型扩增,突变型探针未有荧光信号(图 1A);以突变纯合型质粒为模板扩增的结果显示突变型探针出现典型扩增,野生型探针未有荧光信号(图 1B).杂合型模板2种探针的信号都得到了有效的扩增,混合引物和探针能将突变型和野生型的模板有效地区分开来(图 1C).针对阴性对照模板2种探针均未有起跳信号(图 1D).
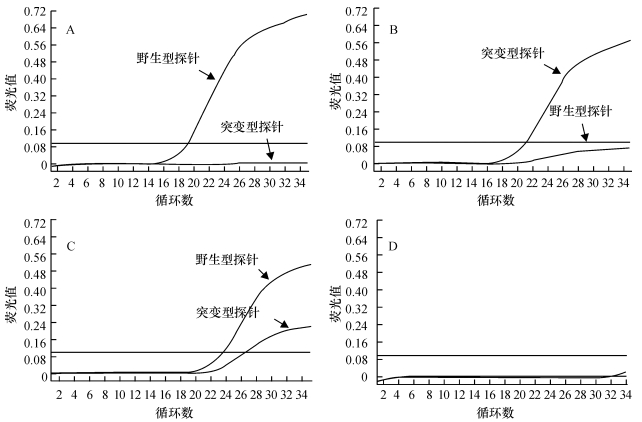
|
图 1 CVM基因特异性试验 Figure 1 Specificity tests for CVM gene A:野生型模板的扩增;B:突变型模板的扩增;C:杂合型模板的扩增;D:阴性模板的扩增. |
采用质粒提取试剂盒纯化阳性克隆质粒DNA,用微量核酸分光光度计测纯化质粒DNA样品的浓度.根据公式:质粒拷贝数=质粒DNA质量浓度(μg·μL-1)/(质粒分子碱基数× 10-15 μg),将纯化质粒DNA质量浓度换算成单位体积对应基因拷贝数.由此计算得到的各种基因型的检测灵敏度为:突变型质粒拷贝数1 × 103 μL-1(图 2A),杂合型质粒拷贝数1 × 104 μL-1(图 2B),野生型质粒拷贝数5 × 103μL-1(图 2C).
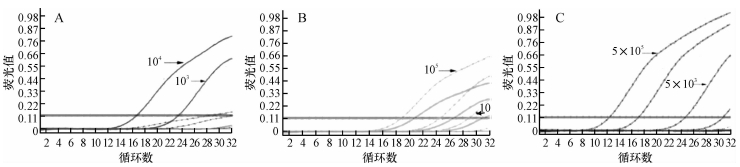
|
图 2 CVM基因敏感性试验 Figure 2 Sensitivity tests for CVM gene A:突变型模板;B:杂合型模板;C:野生型模板. |
在临床样品检测中,设G/G、T /T和T /G基因型质粒为对照,将TaqMan探针法的荧光PCR曲线分成3组.3组基因型区别明显,有3种熔解曲线,292例样品中286份为G/G基因型,6份为T /G基因型.
2.4 准确性分析从检测的临床样品中,挑选3个杂合型样品和5个野生型样品做测序分析,测序结果和TaqMan探针法分型结果吻合(图 3),说明TaqMan探针检测方法准确可靠.
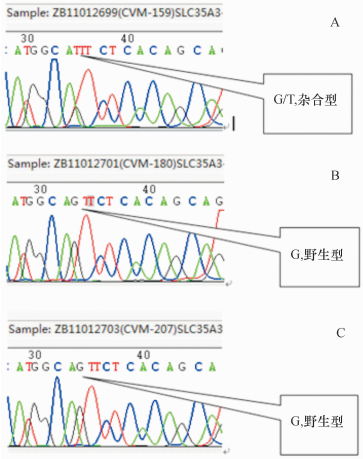
|
图 3 临床样品基因组DNA测序图 Figure 3 Graphics of genomic DNA sequencing of clinical samples A:G/T杂合型;B、C:G/G野生型. |
近年,我国多份研究报告显示,从国外引进的荷斯坦牛及国内奶牛厂养殖的荷斯坦牛中检出CVM基因携带者[9-10, 13-14].说明目前国内存在一定数量的CVM基因携带者.要防止CVM给养牛业带来损失最彻底的办法是在奶牛群体中彻底剔除CVM隐性有害基因,采用传统的数量遗传学方法———测交,需要进行长期的后裔测验,既费力又费钱.采用分子生物学方法可快速、准确、经济地筛查出种牛基因型. PCR-SSCP法、PCR-MAMA法与PCR-PIRA是有效筛查CVM致病基因的分子生物学方法,但PCR扩增产物需要同时进行酶切、琼脂糖电泳或聚丙烯酰胺凝胶电泳来判断基因型[9-10, 13],电泳检测环节相对繁琐.
实时荧光PCR方法可以直接检测核酸,准确性高,且不需后续电泳,操作简便.探针法的优胜在于在上、下游引物之间引入了1条与目的模板特异性结合的荧光标记探针,增加了实时荧光PCR的特异性和敏感性[15].本研究根据GenBank已发表的CVM基因的SLC35A3序列,设计合成了1对特异性引物和2条TaqMan探针,建立了奶牛脊椎畸形综合征TaqMan探针检测方法.检测方法能有效地区分突变型、野生型和杂合型.各种基因型质粒拷贝数的检测灵敏度为:突变型质粒拷贝数1 × 103 μL-1,杂合型质粒拷贝数1 × 104 μL-1,野生型质粒拷贝数5 × 103 μL-1.
李艳华等[9]采用PCR-SSCP法对北京地区242头公牛样品和403头母牛样品进行检测分析,CVM基因携带率为母牛5.71%和公牛8.82%.王帅等[10]利用PCR-MAMA法检测154头公牛,发现23头CVM基因携带者,携带率为15.58%.范学华等[13]建立PIRA-PCR法从587头种公牛中检出携带者56头,携带率为9.54%.本研究建立的TaqMan探针检测方法从292头母牛样品中检出6头CVM基因携带者,携带率为2.29%.
为控制CVM对我国奶业生产带来的负面影响,首先应从牛群中筛选出CVM基因携带者,在选种、选配上尽量不使用CVM基因携带者公牛和CVM基因携带者母牛配种,以减少隐性有害基因纯合致死给奶业生产带来的危害.另一方面,从国外引进种牛、牛遗传物质时应严防CVM基因携带者的潜入.本研究建立的方法简便快捷、准确率高,适用样品宽泛,适合大批样本筛选和口岸现场快速筛查.该方法对进一步提高我国检验检疫系统的检疫把关能力、防止不合格的国外相关产品进入国内,保护我国适当的动物卫生健康水平,促进我国奶牛业的健康发展,具有重要意义.
| [1] |
NIELSEN U S, AAMAND G P, ANDERSEN O, et al. Effects of complex vertebral malformation on fertility traits in Holstein cattle[J]. Livestock Prod Sci, 2003, 79(2): 233-238. (  0) 0) |
| [2] |
DUNCAN R B, CARRIG C B, AGERHOLM J S, et al. Complex vertebral malformation in a Holstein calf:Report of a case in the USA[J]. J Vet Diagn Invest, 2001, 13(4): 333-336. DOI:10.1177/104063870101300409 (  0) 0) |
| [3] |
AGERHOLM J S, BENDIXEN C, ANDERSEN O, et al. Complex vertebral malformation in a Holstein calves[J]. J Vet Diagn Invest, 2001, 13(4): 283-289. DOI:10.1177/104063870101300401 (  0) 0) |
| [4] |
REVELL S. Complex vertebral malformation in a Holstein calf in the UK[J]. Vet Rec, 2001, 149(21): 659-660. (  0) 0) |
| [5] |
NAGAHATA H, OOTA H, NITANAI A, et al. Complex vertebral malformation in a Stillborn Holstein Calf in Japan[J]. J Vet Med Sci, 2002, 64(12): 1107-1112. DOI:10.1292/jvms.64.1107 (  0) 0) |
| [6] |
BERGLUND B, PERSSON A, STALHAMMAR H. Effects of complex vertebral malformation on fertility in Swedish Holstein cattle[J]. Acta Vet Scand, 2004, 45(3): 161-165. DOI:10.1186/1751-0147-45-161 (  0) 0) |
| [7] |
RUSC A, KAMINSKI S. Prevalence of complex vertebral malformation carriers among Polish Holstein-Friesian bulls[J]. J Appl Genet, 2007, 48(3): 247-252. DOI:10.1007/BF03195219 (  0) 0) |
| [8] |
SCHUTZ E, SCHARFENSTEIN M, BRENIG B. Implication of complex vertebral malformation and bovine leukocyte adhesion deficiency DNA-based testing on disease frequency in the Holstein population[J]. J Dairy Sci, 2008, 91(12): 4854-4859. DOI:10.3168/jds.2008-1154 (  0) 0) |
| [9] |
李艳华, 乔绿, 张胜利, 等. 北京地区荷斯坦牛脊柱畸形综合征的调查研究[J]. 中国奶牛, 2011(8): 4-7. DOI:10.3969/j.issn.1004-4264.2011.08.002 (  0) 0) |
| [10] |
王帅, 赵学明, 朱化彬. 部分中国荷斯坦种公牛脊柱畸形综合征携带状况的检测和分析[J]. 畜牧兽医学报, 2011, 42(7): 1027-1031. (  0) 0) |
| [11] |
GHANEM M E, AKITA M, SUZUKI T, et al. Complex vertebral malformation in Holstein cows in Japan and its inheritance to crossbred F1 generation[J]. Anim Reprod Sci, 2008, 103(3): 348-354. (  0) 0) |
| [12] |
KANAE Y, ENDOH D, NAGAHATA H, et al. A method for detecting complex vertebral malformation in Holstein calves using polymerase chain reaction-primer introduced restriction analysis[J]. J Vet Diagn Invest, 2005, 17(3): 258-262. DOI:10.1177/104063870501700309 (  0) 0) |
| [13] |
范学华, 张毅, 公维嘉, 等. 我国荷斯坦种公牛CVM遗传缺陷基因的分子检测[J]. 中国畜牧杂志, 2010, 46(19): 14-17. (  0) 0) |
| [14] |
王洪梅, 李建斌, 侯明海, 等. 牛脊椎畸形综合征检测方法的建立与应用[J]. 遗传, 2008, 30(9): 1223-1227. DOI:10.3321/j.issn:0253-9772.2008.09.020 (  0) 0) |
| [15] |
翟新验, 卢胜明, 刘钧. 实时TaqMan PCR技术在兽医学中的应用[J]. 实验动物科学与管理, 2002, 19(2): 50-53. (  0) 0) |
 2015, Vol. 36
2015, Vol. 36



