2. 国家生猪种业工程技术研究中心,广东 新兴 527439;
3. 华南农业大学 动物科学学院,广东 广州 510642
2. National Engineering Research Center for Breeding Swine Industry, Xinxing 527439, China;
3. College of Animal Science, South China Agricultural University, Guangzhou 510642, China
仔猪腹泻是养猪生产中的常见传染病,尤其是这几年,我国频频暴发,仔猪腹泻导致的死亡率达11.5% ~29.5%,其中由大肠埃希菌Escherichia coli引起的发病率和死亡率分别占腹泻发病率和死亡率的56.2%和24.7% [1],给养猪业造成了巨大经济损失.目前,针对仔猪腹泻,虽然可以通过改善饲养环境、缓慢断奶、添加维生素和外源蛋白、使用药物、隔离扑杀、免疫接种等措施防治,但由于新传染病的发生和流行,病毒发生变异快,超强毒株不断出现,疫苗效力不够,并有药物残留等问题,使仔猪腹泻病无法得到有效防治[2-5].此外,用疫苗和药物治疗的方法会导致猪抵抗力下降,耐药性增强,猪群健康度无法提高.因此,通过分子育种的方法培育抗病品种,增强猪只抗病力,从而解决由于片面追求高产而导致的高产与健康等性状间的遗传拮抗[6].遗传学家和育种学家们通过研究鉴定了由大肠埃希菌引起的仔猪腹泻的主效基因黏蛋白13(Mucin 13,MUC13)和α(1,2)岩藻糖转移酶(Alpha(1,2)fucosyltransferase,FUT1)的主效位点[7-10].这2个基因分别影响断奶前仔猪腹泻和断奶后仔猪腹泻,且2003年在法国和西班牙利用FUT1基因分子辅助选择育种首先推出了能特异抵抗F18ab仔猪腹泻的新品系.
温氏集团2005年,在法系皮特兰群体中针对断奶后仔猪腹泻进行过猪FUT1基因的检测,发现猪只对腹泻的抵抗力有差异.2009年针对杜洛克群体断奶前仔猪腹泻进行过MUC13基因位点的检测,发现其抗腹泻等位基因频率达80%左右.本研究利用群体基因频率分析、基因型与性状相关分析等方法,通过估算这2个抗腹泻基因在温氏杜洛克专门化父系S22和皮特兰专门化父系S11核心群中的差异表现,制定特异的抗腹泻集成育种方案,进行不同的集成分子育种筛选,辅助选留,为培育抗病品系奠定基础.
1 材料与方法 1.1 试验动物试验动物来自温氏集团国家生猪种业工程技术研究中心研发基地沙湖原种场杜洛克核心群体和清远一区原种场皮特兰核心群体.样本总数1 178头,其中核心群杜洛克666头,核心群皮特兰512头.利用耳号钳剪取已经利用乙醇消毒过耳尖的待检猪只耳样2 ~3 g,放入装有1.5 mL体积分数为75%乙醇的离心管内,低温保存.
1.2 基因型判型采取的耳组织样本送至江西农业大学动物生物技术国家重点实验室培育基地检测2个抗腹泻基因,检测方法如下:采用酚-三氯甲烷法手工提取基因组DNA.2个基因位点的引物序列及具体扩增情况见表 1,PCR反应体系总体积为20 μL,包括20 ng·μL-1模板DNA 2 μL,10 × buffer 2.0 μL,25 mmol· L-1 MgCl2 1.2 μL,10 mmol · L-1 dNTP 0.3 μL,PF 0.4 μL,PR 0.4 μL,Taq聚合酶0.5 μL,超纯水13.2 μL.PCR循环参数:94 ℃变性3 min,退火温度见表 1,72℃延伸10 min,共36个循环.
|
|
表 1 引物与扩增情况 Table 1 PCR primers and the amplicons |
PCR-SNaPshot的基本原理是通过引物在标有不同荧光的ddNTP(A、G、C、T)反应体系内进行一步延伸后,使用3130XL型DNA测序仪检测不同的荧光信号.MUC13的基因型利用PCR-SNaPshot法进行检测,其SNaPshot反应体系为5 μL,其中含2 μL SNaPshot multiplexmix(含Taq聚合酶和荧光标记的ddNTPs),1.5 μL纯化后PCR产物,1.2 μL去离子水,0.3 μL SNaPshot引物.SNaP- shot反应体系的扩增程序为:96 ℃ 10 s,50 ℃ 5 s,60 ℃ 30 s,25个循环.反应后,在SNaPshot反应产物中加0.57 μL 1 × NEB buffer和0.1 μL的CIP,37 ℃反应60 min以清除荧光标记的ddNTPs,随后75 ℃反应15 min灭活纯化酶;最后,每1 μL SNaPshot反应产物加8 μL Hi-Di formamide与GeneScan 120 LIZ size standard混合物(两者体积比为20: 1),经变性后上样于ABI3130XL自动遗传分析仪(ABI,USA)进行电泳,利用GeneMapper Software version 4.0(ABI,USA)进行基因型判定和数据收集.
1.2.2 PCR-RFLP法FUT1主效位点是通过Hin6I PCR-RFLP判定基因型,检测酶切反应体系与条件如下:反应体系总体积为15 μL,其中10 × Buffer 1.5 μL,HinP1I内切酶0.3 μL,DDW 8.2 μL,PCR产物5 μL.37 ℃水浴反应,过夜.PCR产物通过0.02 g·mL-1的琼脂糖凝胶电泳来判型:A等位基因可酶切为3 283和93 bp 2条带,G等位基因表现为241、93、87 bp 3条带.
1.3 统计分析通过R软件进行表型与基因型相关分析,模型为:y = 1μ + Zα + e.其中,y为所测得的表型值,μ为总体平均值,Z为基因型的指示矩阵,α为SNP随机加性遗传效应向量,且α-N(0,Gσ2α)(G为分子血缘相关矩阵,σ2α为加性遗传方差),Zα为基因型效应,e为残差.
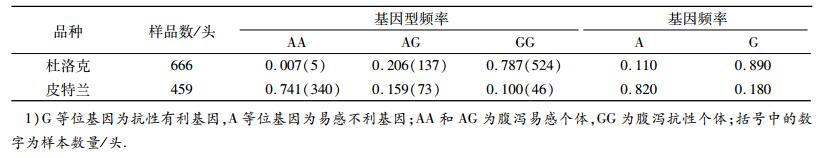
2 结果与分析 2.1 基因频率和基因型频率 2.1.1 MUC13的基因型及基因频率从2个父系群体的MUC13的基因型频率及基因频率分布(表 2)来看,2个群体表现差异较大,G等位基因为抗性有利基因,杜洛克群体有利等位基因频率高达0.890,而皮特兰群体仅为0.180.
|
|
表 2 MUC13基因型及基因频率的分布1) Table 2 MUC13 genotypes and allele frequencies in tested breeds |
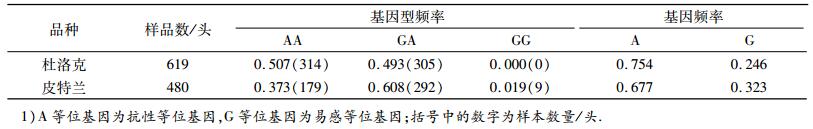
从2个父系群体的FUT1基因型频率及基因频率分布(表 3)来看,2个群体在此位点表现差异不大,A等位基因为抗性有利等位基因,有利等位基因频率偏高;杜洛克猪未检测到GG基因型个体,皮特兰猪GG频率也相对较低.
|
|
表 3 FUT1基因型及基因频率的分布1) Table 3 FUT1 genotypes and allele frequencies in tested breeds |
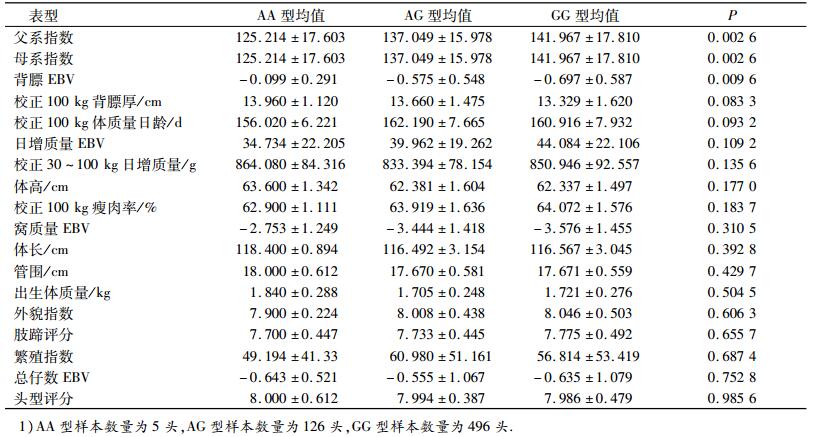
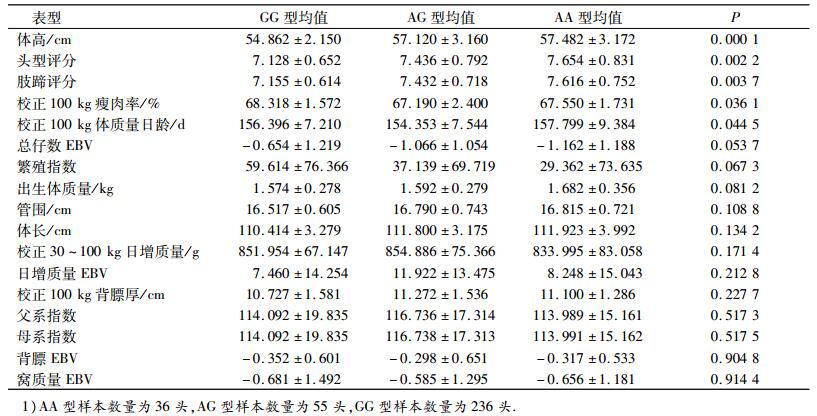
由MUC13与杜洛克群体18个选育性能的相关分析结果(表 4)可以看出,在父系指数、母系指数以及背膘育种估计值(EBV)选育性能中,GG型个体要明显优于AA和AG型个体;而在其余的性能中,则3种基因型表现差异不大.
|
|
表 4 MUC13的基因型与杜洛克生长性能的关系1) Table 4 The relationship between MUC13 genotype and growth performance in Duroc |
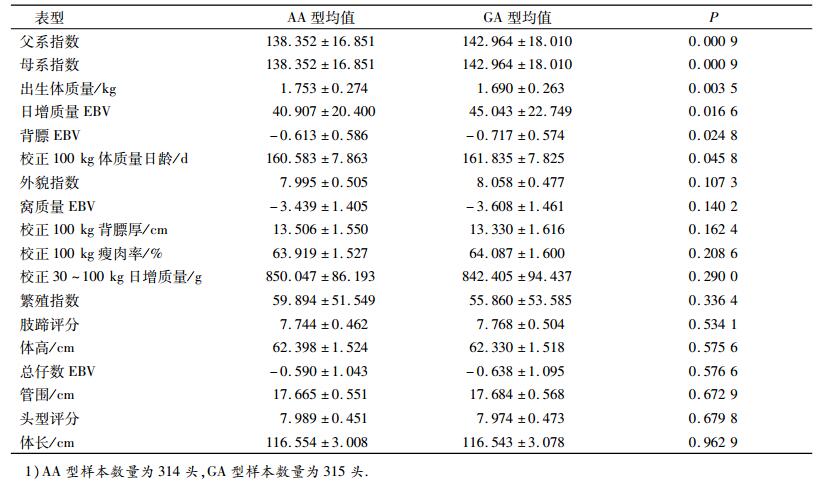
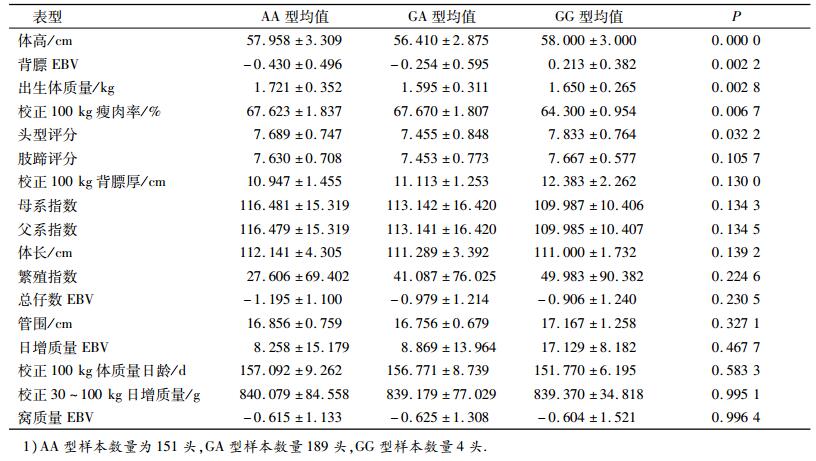
由FUT1与杜洛克群体18个选育性能的相关分析结果(表 5)可以看出,在父系指数、母系指数、日增质量EBV以及背膘EBV等选育性能中,GA型个体要明显优于AA型个体;而在出生体质量和100 kg体质量日龄性能上,AA型个体要明显优于GA型个体;2种基因型在其余性能中表现差异不大.
|
|
表 5 FUT1的基因型与杜洛克生长性能的关系1) Table 5 The relationship between FUT1 genotype and growth performance in Duroc |
由MUC13的基因型与皮特兰群体18个选育性能的相关分析结果(表 6)可以看出,在体高、头型评分、肢蹄评分等体貌表型上,GG型个体要明显差于AA和AG型个体;而GG型个体的校正100 kg瘦肉率明显高于AA和AG型个体;AG杂合子在校正100 kg体质量日龄方面要明显优于AA和GG型个体;3种基因型在其他性能中表现差异不大.
|
|
表 6 MUC13基因型与皮特兰生长性能的关系1) Table 6 The relationship between MUC13 genotype and growth performance in Pietrain |
由FUT1的基因型与皮特兰群体18个选育性能的相关分析结果(表 7)可以看出,在体高、头型评分体貌表型上,GG型个体要明显优于AA和GA型个体;背膘EBV和出生体质量等选育的生长性能方面,AA型个体要明显优于其他2种基因型个体;而杂合子GA型个体在校正100 kg瘦肉率方面表现较好;在其他性能中基因型间表现差异不大.
|
|
表 7 FUT1基因型与皮特兰生长性能的关系1) Table 7 The relationship between FUT1 genotype and growth performance in Pietrain |
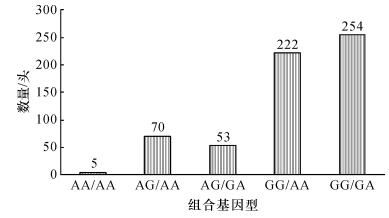
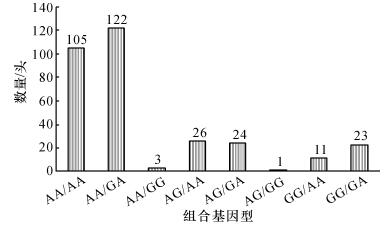
图 1表明:杜洛克群体有5种组合类型,其中MUC13与FUT1基因均为优势基因型组合GG/AA的个体数为222头,占群体总量的36.75%(222 /604).图 2结果表明:皮特兰群体有8种组合类型,仅有11头是优势基因型组合个体,占群体总量的3.49%(11 /315).
|
图 1 杜洛克群体MUC13与FUT1基因型组合分布 Figure 1 Distributions of MUC13 and FUT1 genotypic combina- tions in Duroc pigs |
|
图 2 皮特兰群体MUC13与FUT1基因型组合分布 Figure 2 Distributions of MUC13 and FUT1 genotypic combina- tions in Pietrain pigs |
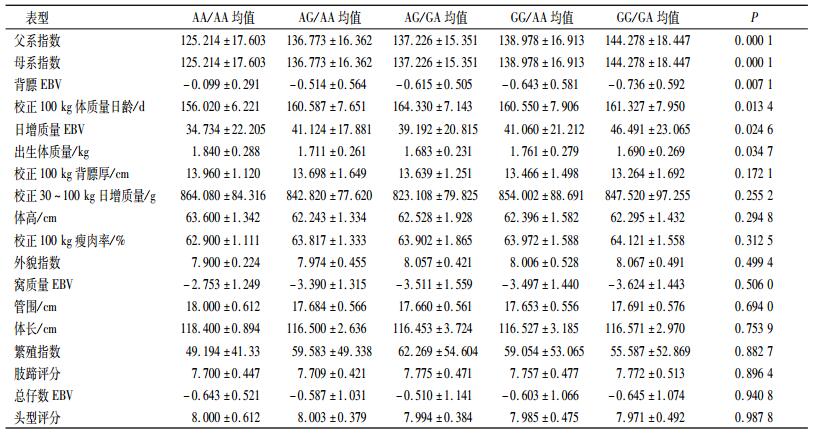
MUC13、FUT1组合基因型与杜洛克群体18个选育性能的相关分析结果(表 8)可以看出,在父系指数、母系指数、背膘EBV以及日增质量EBV选育性能中,GG/GA型个体要明显优于其他组合型个体;在校正100 kg体质量日龄、出生体质量方面,AA /AA组合型要比其他组合型占优势.其余性状中,几个组合型表现无差异.
|
|
表 8 MUC13、FUT1组合基因型与杜洛克生长性能的关系 Table 8 The relationship between MUC13, FUT1 genotypic combination and growth performance in Duroc |
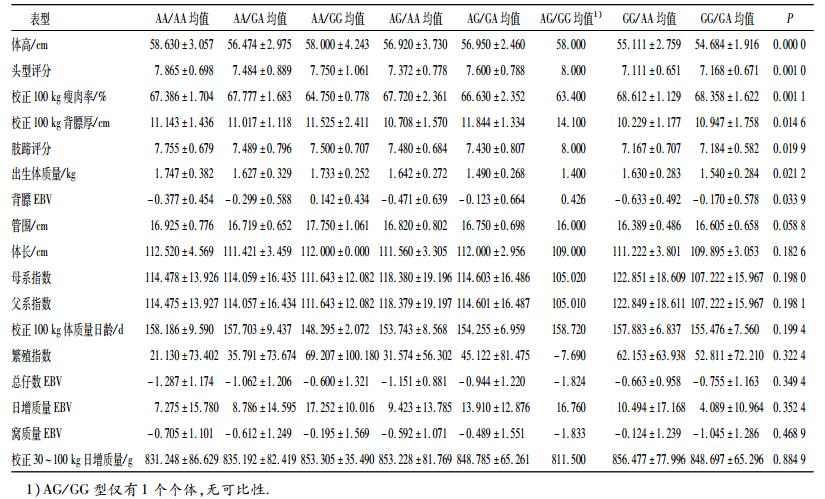
MUC13、FUT1组合基因型与皮特兰群体18个选育性能的相关分析结果见表 9.由于仅有1头AG/GG组合型个体,数据准确性不够,不做比较.由表 9可以看出,在体高、头型评分、肢蹄评分以及出生体质量表型上,AA /AA组合型要比其他组合型占优势;在校正100 kg瘦肉率、校正100 kg背膘厚以及背膘EBV性能中,GG/AA组合型个体要明显优于其他组合型个体;在其余性能中各组合型间表现差异不大.
|
|
表 9 MUC13、FUT1组合基因型与皮特兰生长性能的关系 Table 9 The relationship between MUC13, FUT1 genotypic combination and growth performance in Pietrain |
杜洛克作为商业猪种中的重要父系品种,其与生产相关的各性状都经过了严格的选育,尤其是核心育种群.本研究在温氏杜洛克核心育种群中检测抗大肠埃希菌引起的断奶前仔猪腹泻基因MUC13以及抗大肠埃希菌引起的断奶后仔猪腹泻基因FUT1的主效位点变异情况,结果显示:在温氏集团杜洛克核心育种群中,MUC13优势等位基因频率高达89.0%,这与阮国荣等[11]在福建省杜洛克群体中的检测结果一致,也与温氏集团生产实践中杜洛克种猪群体断奶前仔猪腹泻比例低的情况一致,只需对少量的劣势等位基因纯合个体及公猪杂合个体进行淘汰,对杂合母猪根据现场选育情况减少选留,慎重选配,经过3个世代左右可育成抗断奶前仔猪腹泻纯系.而FUT1基因的检测中,有利等位基因频率大于0.75,与其他商业种猪群体的检测结果相比偏高[12-13],这与温氏杜洛克群体多年的群体选育息息相关,断奶后仔猪腹泻情况较轻微.2个基因均与父系指数、母系指数极显著相关,说明这2个基因在温氏杜洛克群体中具备应用于辅助选择的潜力.
通过分析2个抗腹泻基因组合基因型分布情况发现:在温氏杜洛克群体中,GG/GA型组合(254头)所占群体比例最高达42.05%.各种组合基因型与各生产性状的相关分析显示,GG/GA组合型个体的综合生产性能表现最好,这与本文的单基因相关分析结果一致.由于2个基因有利基因型所占比例相差较大,在兼顾性能测定和表型的现场实践选育的情况下,2个基因聚合育种需要经过多个世代才能实现优化.鉴于此,在杜洛克群体中,MUC13基因的优势等位基因频率近90%,建议先实现MUC13基因的辅助选育纯化,再启动FUT1基因的选育工作,相对来讲,较同时选育更容易育成抗腹泻纯合品系.
3.2 温氏皮特兰核心群体的抗腹泻基因分子辅助选择方案温氏皮特兰核心群体MUC13基因的表现与杜洛克群体完全不同,皮特兰MUC13优势等位基因频率仅0.180,这与当时的产房生产状况相吻合,断奶前腹泻仔猪偏多.MUC13基因型与生产性能相关分析结果显示,有利基因型与体高、头型评分、肢蹄评分等体型外貌性状呈极显著负相关,仅与100 kg瘦肉率生产性状呈正相关.根据目前市场需求分析制定的皮特兰选育方案中,体型外貌为重点选育方向.因此,综合考虑群体优势等位基因频率低及与体型外貌的负相关等因素,MUC13基因暂不列为皮特兰群体的选育候选基因.
FUT1基因在皮特兰群体中的表现与在杜洛克群体中的表现相差不大,优势等位基因频率达0.677,其中杂合子个体偏多.FUT1基因型与生产性能相关分析结果显示,优势基因型(AA)在体高、头型评分等体型外貌方面表现不如GG型个体,但在背膘EBV和出生体质量方面表现出正相关趋势.综合现场育种实践,FUT1基因在皮特兰群体中的优化较难实现,基础群杂合子过多造成优化周期加长,且基因型选择与现场育种实践指标不一致(体型外貌)或相关性不高(父系指数等),因此,皮特兰群体暂时不宜对FUT1基因进行纯化,仅需保持对基因频率进行长期监控即可,在市场以及群体频率变化适宜时,再开展优化工作.
3.3 关于基因聚合育种的群体特异性在本研究中,同为终端父本的杜洛克和皮特兰群体的抗腹泻基因频率表现不同,基因型和生产性状的相关分析也表现出较大差异.由于核心育种群都是以固定的育种目标长期选育的群体,因此同样的基因在不同的核心育种群的频率分布和对各生产性状的影响表现不同.若是多个基因聚合在不同的育种群中,可能由于不同基因频率、不同的基因型组合分布、不同相关性、不同连锁关系以及不同选育方向等综合因素表现出更加复杂的特异性,所以基因聚合育种不能通用任何群体,而是需要针对不同群体进行分析和验证,具有群体特异性.在本研究中2个抗腹泻基因辅助选择育种,可应用于温氏杜洛克群体,却并不适用于皮特兰群体,与2个群体长期向着不同的选育方向选育有关.其基因型组合分布呈现很大差异,根据相关分析及现场育种的实践出发,制定适宜的抗腹泻基因的分子辅助选择方案.
致谢:感谢江西农业大学省部共建猪遗传改良与养殖技术国家重点实验室任军教授和黄路生院士给予的支持和帮助!
| [1] |
施启顺. 猪肠毒性大肠杆菌(ETEC)病抗病育种研究[J]. 中国畜牧兽医, 1999, 26(4): 51-54. (  0) 0) |
| [2] |
许建民, 赵德明. 仔猪肠毒素性大肠杆菌病的防治[J]. 中国兽医杂志, 2005, 41(2): 48-50. DOI:10.3969/j.issn.0529-6005.2005.02.028 (  0) 0) |
| [3] |
刘勇, 顾正明, 王克虎, 等. 微生态制剂促康生Ⅱ号治疗大肠杆菌抗药性菌株引起的仔猪黄痢白痢临床效果观察[J]. 中国兽医杂志, 2001, 37(7): 14. DOI:10.3969/j.issn.0529-6005.2001.07.006 (  0) 0) |
| [4] |
曹国文, 姜永康, 周淑兰, 等. 中草药抗猪大肠杆菌制剂的研制与效果观察[J]. 黑龙江畜牧兽医, 2002, 26(10): 33-34. DOI:10.3969/j.issn.1004-7034.2002.10.025 (  0) 0) |
| [5] |
李书华, 李艳琴, 冯敏, 等. 生态制剂--制痢康防治仔猪下痢的试验研究[J]. 黑龙江畜牧兽医, 1999, 23(5): 19-20. (  0) 0) |
| [6] |
SCHAEFER L R. Strategy for applying genome-wide selection in dairy cattle[J]. J Anim Breed Genet, 2006, 123(4): 218-223. DOI:10.1111/jbg.2006.123.issue-4 (  0) 0) |
| [7] |
VÖGELI P, BERTSCHINGER H U, STAMM M, et al. Genes specifying receptors for F18 fimbriated Escherichia coli, causing oedema disease and postweaning diarrhoea in pigs, map to chromosome 6[J]. Anim Genet, 1996, 27(5): 321-328. (  0) 0) |
| [8] |
ZHANG B, REN J, YAN X M, et al. Investigation of the porcine MUC13 gene: Isolation, expression, polymor- phisms and their strong association with susceptibility to enterotoxigenic E.coli F4ab/ac[J]. Anim Genet, 2008, 39(3): 258-266. DOI:10.1111/age.2008.39.issue-3 (  0) 0) |
| [9] |
REN J, YAN X M, AI H S, et al. Susceptibility towards enterotoxigenic Escherichia coli F4ac diarrhea is governed by the MUC13 gene in pigs[J/OL]. PloS One, 2012, 7 (9): e44573[2014-11-11]. http://journals.plos.org/plosone/article?id=10.1371/journal.pone.0044573.
(  0) 0) |
| [10] |
GOETSTOUWERS T, POUCKE M V, COPPIETERS W, et al. Refined candidate region for F4ab/ac enterotoxigen- ic Escherichia coli susceptibility situated proximal to MUC13 in pigs[J/OL]. PloS One, 2014, 9(8): e105013[2014-11-11]. http://journals.plos.org/plosone/article?id=10.1371/journal.pone.0105013.
(  0) 0) |
| [11] |
阮国荣, 肖石军, 徐盼, 等. 福建省商业猪种MUC13、IGF2和RYR1基因主效位点的遗传变异分析[J]. 江西农业大学学报, 2012, 34(5): 997-1002. DOI:10.3969/j.issn.1000-2286.2012.05.026 (  0) 0) |
| [12] |
CODDENS A, VERDONCK F, MULINGE M, et al. The possibility of positive selection for both F18 + Escherichia coli and stress resistant pigs opens new perspectives for pig breeding[J]. Vet Microbiol, 2008, 126(1/2/3): 210-215. (  0) 0) |
| [13] |
晏学明, 郭源梅, 丁能水, 等. 不同品种猪a1岩藻糖转移酶基因遗传变异初析[J]. 中国畜牧杂志, 2004, 40(3): 8-10. DOI:10.3969/j.issn.0258-7033.2004.03.004 (  0) 0) |
 2015, Vol. 36
2015, Vol. 36