 种群遗传结构分析
种群遗传结构分析2. 清远市兴渔水产科技有限公司,广东 清远 511510
 Cranoglanis 3个种群的野生资源状况,并对3个种群的物种有效性进行分析。【方法】 采用线粒体控制区Dloop基因序列测定技术, 分析了珠江水系、海南水系和越南红河水系长臀
Cranoglanis 3个种群的野生资源状况,并对3个种群的物种有效性进行分析。【方法】 采用线粒体控制区Dloop基因序列测定技术, 分析了珠江水系、海南水系和越南红河水系长臀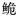 种群的群体遗传结构及其变异。【结果】 在检测的84个个体中共得到43个单倍型,呈现出较高的单倍型多样性与较为贫乏的核苷酸多样性,其中海南长臀
种群的群体遗传结构及其变异。【结果】 在检测的84个个体中共得到43个单倍型,呈现出较高的单倍型多样性与较为贫乏的核苷酸多样性,其中海南长臀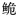 C. multiradiatus群体的单倍型多样性(Hd=0.871)和核苷酸多样性(Pi=0.006 4)最低;Tajima’s D中性检验以及核苷酸不配对分析均表明,3个长臀
C. multiradiatus群体的单倍型多样性(Hd=0.871)和核苷酸多样性(Pi=0.006 4)最低;Tajima’s D中性检验以及核苷酸不配对分析均表明,3个长臀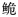 群体趋于稳定,未经历过大规模的种群扩张。Fst分析发现,海南长臀
群体趋于稳定,未经历过大规模的种群扩张。Fst分析发现,海南长臀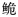 同珠江长臀
同珠江长臀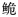 C. bouderius、红河长臀
C. bouderius、红河长臀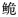 C. henrici产生了一定的遗传分化,而珠江和红河群体未发现明显遗传分化,从遗传距离来看,珠江和红河长臀
C. henrici产生了一定的遗传分化,而珠江和红河群体未发现明显遗传分化,从遗传距离来看,珠江和红河长臀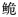 净遗传距离为0.000。【结论】 长臀
净遗传距离为0.000。【结论】 长臀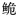 野生资源较为贫乏,且海南群体最为严重;认为应将珠江长臀
野生资源较为贫乏,且海南群体最为严重;认为应将珠江长臀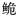 和红河长臀
和红河长臀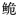 归为同一亚种长臀
归为同一亚种长臀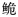 C. bouderius,而海南长臀
C. bouderius,而海南长臀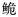 作为长臀
作为长臀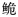 的另一个亚种。
的另一个亚种。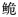 线粒体控制区 Dloop基因序列 物种有效性 遗传结构 资源状况
线粒体控制区 Dloop基因序列 物种有效性 遗传结构 资源状况 2. Qingyuan Xingyu Fishery Science Co., Ltd., Qingyuan 511510, China
鱼类线粒体基因是遗传信息重要的载体,具有母系遗传、进化速度快等特点,是研究种群遗传结构的理想材料[1-2];线粒体控制区又称D-环区(Dloop),其进化速度最快,是mtDNA其他区段的5~10倍[3],遗传变异大,是种群遗传多样性研究的常用标记之一,很适合做种内、种群或个体间的遗传分化研究[4]。
长臀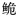
自1927年Koller[11]在海南发现一新种Pseudeutropichthys multiradiatus Koller以来,长臀
珠江长臀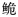
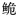
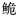
组织样品DNA提取试剂盒为TIANGEN生化科技有限公司产品、Tap DNA聚合酶为TaKaRa有限公司产品,其他试剂均为分析纯。引物由上海捷瑞生物公司合成,测序由上海英骏公司完成。引物根据NCBI上已有长臀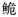

取30 mg左右鳍条样品,于蒸馏水中漂洗数次,用滤纸吸干表面水分,放入1.5 mL离心管中,用剪刀剪碎,加裂解液,在55 ℃水域中直至裂解完全,按TIANGEN组织DNA提取试剂盒说明书进行操作,然后琼脂糖凝胶电泳检验提取效果,于-20 ℃条件保存供后续试验。
1.2.2 长臀
PCR反应体系总体积为50 μL,其中包括10×Buffer 5 μL,dNTPs (2.5 mmol·L-1) 4 μL,上、下游引物各1 μL,Ex Taq酶(1 U·μL-1) 2 μL,模板1 μL,用双蒸水补足50 μL。Dloop PCR反应条件:94 ℃预变性5 min;94 ℃变性30 s,54 ℃退火30 s,72 ℃延伸1 min,35个循环;72 ℃延伸10 min。反应结束后,取2 μL PCR产物做琼脂糖凝胶电泳,检验目的基因质量,然后送上海英骏生物公司测序鉴定。
1.2.3 序列分析测序结果同NCBI上序列比对分析后,用Clustal X软件进行排序,并进行手工校正[17]。MEGA 5.0软件统计序列的碱基组成、变异位点数、简约信息位点以及群体间的遗传距离[18];用DnaSP 5.0计算单倍型多态性和核苷酸多样性等遗传多样性指数[19]。利用Arlequin3.5中性检验和核苷酸不配对分布2种方法来检测长臀

采用Clustal X软件分别对长臀
|
|
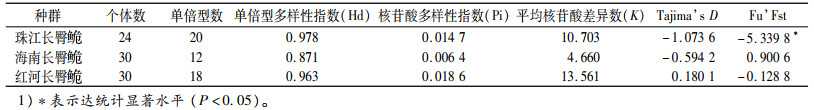
表 1 长臀 |

采用DNAsp5.0和Arlequin3.5软件对3个长臀
|
|
表 2 43个单倍型在长臀 |
|
|
表 3 长臀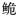 |
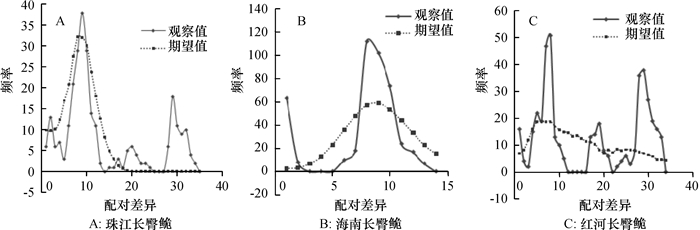
|
图 1 长臀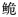 |

采用Mega5.0对长臀
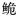
|
|
表 4 长臀 |
|
图 2 基于Dloop基因序列构建的长臀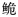 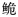 C. bouderius;H:海南长臀 C. bouderius;H:海南长臀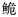 C. multiradiatus;Y:红河长臀 C. multiradiatus;Y:红河长臀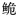 C. henrici。 C. henrici。 |

遗传多样性是物种不断进化适应环境的基础,是评价生物资源状况的重要依据,遗传多样性的降低或丧失, 会对野生群体的资源状况造成极大威胁。采用Dloop基因序列对长臀

长臀
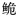
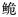
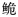
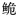

长臀
| [1] |
唐文乔, 胡雪莲, 杨金权. 从线粒体控制区全序列变异看短颌鲚和湖鲚的物种有效性[J]. 生物多样性, 2007, 15(3): 224-231. DOI:10.3321/j.issn:1005-0094.2007.03.002 (  0) 0) |
| [2] |
牛素芳, 苏永全, 钟声平, 等. 福建近海2个鲐鱼群体遗传结构与遗传多样性分析[J]. 台湾海峡, 2012, 31(4): 509-515. DOI:10.3969/J.ISSN.1000-8160.2012.04.010 (  0) 0) |
| [3] |
ROSEL P E, DIZON A E, HAYGOOD M G. Variability of the mitoehondrial control region in populations of the harbour porpoise, Phoeoena, on interoeeanic and regional scales[J]. Can J Fish Aquat Sci, 1995, 52(6): 1421-1429. (  0) 0) |
| [4] |
乐小亮. 中国野生香鱼(Plecogzossus altivelis)遗传多样性分析[D]. 广州: 暨南大学, 2010.
(  0) 0) |
| [5] | |
| [6] | |
| [7] |
乐佩琦, 陈宜瑜. 中国濒危动物红皮书:鱼类[M]. 北京: 科学出版社, 1998, 240-243.
(  0) 0) |
| [8] |
乐小亮, 赵爽, 刘海林, 等. 基于线粒体Cytb基因对南渡江海南长臀  遗传多样性分析[J]. 生态科学, 2010, 29(5): 247-250. ( 遗传多样性分析[J]. 生态科学, 2010, 29(5): 247-250. ( 0) 0) |
| [9] |
高志远, 章群, 夏月恒, 等. 松涛水库长臀鮠遗传多样性研究[J]. 广东农业科学, 2013, 40(3): 98-100. DOI:10.3969/j.issn.1004-874X.2013.03.033 (  0) 0) |
| [10] |
程飞, 谢松光, 叶卫. 长臀鮠的AFLP分析[J]. 水生生物学报, 2009, 33(3): 539-545. (  0) 0) |
| [11] |
KOLLER O. Fishe von der Insel Hainan[J]. Anna des Naturh Mus Wien, 1927, 41: 25-49. (  0) 0) |
| [12] |
MYERS G S. On the fishes described by Koller from Hainan in 1926 and 1927[J]. Lingnan Sci J, 1931, 10: 255-262. (  0) 0) |
| [13] |
JAYARAM K C. Taxonomic status of the Chinese catfish family Cranoglanideae Myers[J]. Proc Natn Inst Sci India, 1995, 15: 85-87. (  0) 0) |
| [14] |
褚新洛.郑葆珊, 成定远. 中国动物志:硬骨鱼纲·鲇形目[M]. 北京: 科学出版社, 1999, 74-77.
(  0) 0) |
| [15] |
NG H H, KOTTELAT M. Cranoglanis henrici (Vaillant, 1893), a valid species of cranoglanidid catfish from Indochina (Teleostei, Cranglanididae)[J]. Zoosystema, 2000, 22(4): 847-852. (  0) 0) |
| [16] |
刘彩霞. 长臀  属与鲚属的物种有效性分析及系统学研究[D]. 武汉: 中科院水生生物研究所, 2007. http://cdmd.cnki.com.cn/Article/CDMD-80119-2008010704.htm
( 属与鲚属的物种有效性分析及系统学研究[D]. 武汉: 中科院水生生物研究所, 2007. http://cdmd.cnki.com.cn/Article/CDMD-80119-2008010704.htm
( 0) 0) |
| [17] |
THOMPSON J D, GIBSON T J, PLEWNIAK F, et al. The CLUSTAL_X window s interface: Flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Res, 1997, 25(24): 876-4882. (  0) 0) |
| [18] |
TAMURA K, PETERSON D, PETERSON N, et al. MEGA5:Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Mol Biol Evol, 2011, 28(10): 2731-2739. DOI:10.1093/molbev/msr121 (  0) 0) |
| [19] |
LIBRADO P, ROZAS J. DnaSPv5: A software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25(11): 1451-1452. DOI:10.1093/bioinformatics/btp187 (  0) 0) |
| [20] |
EXCOFFIER L, LISCHER H E L. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows[J]. Mol Ecol Resour, 2010, 10(3): 564-567. DOI:10.1111/men.2010.10.issue-3 (  0) 0) |
| [21] |
陈大庆, 张春霖, 鲁成, 等. 青海湖裸鲤繁殖群体线粒体基因组Dloop区序列多态性[J]. 中国水产科学, 2006, 13(5): 800-806. DOI:10.3321/j.issn:1005-8737.2006.05.017 (  0) 0) |
| [22] |
袁希平, 严莉, 徐树英, 等. 长江流域铜鱼和圆口铜鱼的遗传多样性[J]. 中国水产科学, 2008, 15(3): 380-383. (  0) 0) |
| [23] |
杨金权, 胡雪莲, 唐文乔, 等. 长江口邻近水域刀鲚的线粒体控制区序列变异与遗传多样性[J]. 动物学杂志, 2008, 43(1): 8-15. DOI:10.3969/j.issn.0250-3263.2008.01.002 (  0) 0) |
| [24] |
袁娟, 张其中, 李飞, 等. 铜鱼线粒体控制区的序列变异和遗传多样性[J]. 水生生物学报, 2010, 34(1): 13-16. (  0) 0) |
| [25] |
ROGERS A R, HARPENDING A. Population growth makes waves in distribution of pairwise genetic differences[J]. Mol Biol Evol, 2006, 9: 552-569. (  0) 0) |
 2016, Vol. 37
2016, Vol. 37






 人工繁殖技术探讨[J]. 水利渔业, 2005, 25(2): 22-23.
人工繁殖技术探讨[J]. 水利渔业, 2005, 25(2): 22-23.  人工繁殖与胚胎发育的研究[J].
人工繁殖与胚胎发育的研究[J].