伪狂犬病病毒(Pseudorabies virus,PRV)属疱疹病毒科,α疱疹病毒亚科,对世界养猪业危害巨大。疫苗免疫是防控猪伪狂犬病的重要措施。在我国,许多猪场采用Bartha株疫苗预防猪伪狂犬病且取得了显著效果,但自2011年以来,许多Bartha株疫苗免疫猪场重新爆发了猪伪狂犬病疫情[1]。将新流行株接种Bartha株疫苗免疫的猪和羊,结果显示猪和羊均发病,表明Bartha株疫苗对新毒株不能提供完全保护[1-5]。
伪狂犬病毒的糖蛋白不仅在病毒的侵入、释放以及细胞间扩散等过程中起重要作用,而且也是参与机体免疫反应的主要靶蛋白[6]。因此,对这些糖蛋白进行深入分析有助于了解新毒株的致病性及免疫原性。本研究在成功分离猪伪狂犬病毒新流行株的基础上,对其进行了遗传进化分析及主要糖蛋白gB、gC、gD、gE的序列分析,以期从分子水平上解析现阶段猪伪狂犬病疫情爆发的可能原因。
1 材料与方法 1.1 材料 1.1.1 病料和细胞病料采自安徽某猪场流产胎儿的脑组织,经PCR检测为PRV gE阳性。PK-15细胞由华南农业大学兽医微生物学与免疫学教研室保存。
1.1.2 试剂LA Taq酶、DNA Marker、琼脂糖、pMD18-T载体均为TaKaRa公司产品,DMEM培养液为Gibco公司产品,胎牛血清为Hyclone公司产品。
1.1.3 试验动物5周龄的雌性昆明小鼠购自南方医科大学实验动物中心。
1.2 方法 1.2.1 病毒的分离将病料在DMEM中充分研磨后,于4 ℃条件下6 800 r·min-1离心15 min,收集上清液。将上清液用0.22 μm的滤器过滤,接种至PK-15单层细胞中,观察到细胞出现明显病变时,将细胞反复冻融3次后收集病毒,通过PCR鉴定病毒。
1.2.2 致病性试验将30只5周龄的昆明小鼠分成6组,分别接种10-1、10-2、10-3、10-4稀释度的PRV AH病毒液100 μL,另外2组分别接种未稀释的病毒液100 μL作阳性对照和DMEM 100 μL作阴性对照,每天对小鼠的临床症状和死亡情况进行观察,最后计算出病毒的LD50。
1.2.3 引物设计参照GenBank发布的PRV序列(GI:32187339)设计4对引物分别扩增PRV gB、gC、gD、gE基因序列,引物由生工生物工程股份有限公司合成。
1.2.4 PCR扩增用LA Taq酶扩增gB、gC、gD、gE 4段基因序列,其引物见表 1。
|
|
表 1 扩增主要糖蛋白基因的引物序列 Table 1 Primer sequences of genes encoding the major glycoproteins |
PCR反应条件为:94 ℃预变性5 min;94 ℃变性45 s,退火温度条件下45 s,72 ℃延伸2 min,循环30次;72 ℃末次延伸10 min,4 ℃保存。gB、gC、gD、gE基因的PCR扩增最适退火温度分别为:50、56、58、55 ℃。
1.2.5 遗传进化分析用MEGA5.0软件将PRV AH和部分国内外经典的PRV毒株的gC、gE基因序列进行比对分析,并采用邻位相连法绘制系统发育树。
1.2.6 主要糖蛋白的序列分析将PRV AH与PRV其他毒株的gB、gC、gD、gE氨基酸序列进行比对分析,以解析新流行株与其他毒株的差异。
2 结果与分析 2.1 病毒分离将流产胎儿脑组织样品接种到PK-15单层细胞上,在接种48 h后观察到了明显细胞病变,提取病变细胞总DNA为模板,利用PRV gE特异性引物进行PCR检测,结果显示可以扩增出gE特异性条带(图略),证实病料中含有伪狂犬病毒,并命名为PRV AH。
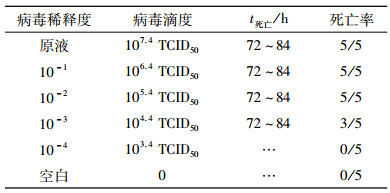
2.2 致病性试验所有接种10-1和10-2稀释度病毒液的小鼠都在72~84 h内死亡,有3只接种10-3稀释度病毒液的小鼠在72~84 h内死亡。接种10-4稀释度病毒液的小鼠未发生死亡。所有接种死亡的小鼠都表现典型的伪狂犬病症状,如神经症状和奇痒症状,小鼠在接种不同稀释度的病毒液后的死亡情况见表 2。在PK-15细胞中测得PRV AH分离株的TCID50为10-7.4/0.1 mL。经寇氏法测得病毒对小鼠的LD50为104.3TCID50。
|
|
表 2 分离株PRV AH对小鼠的致病性试验 Table 2 The pathogenicity of PRV AH to mice |
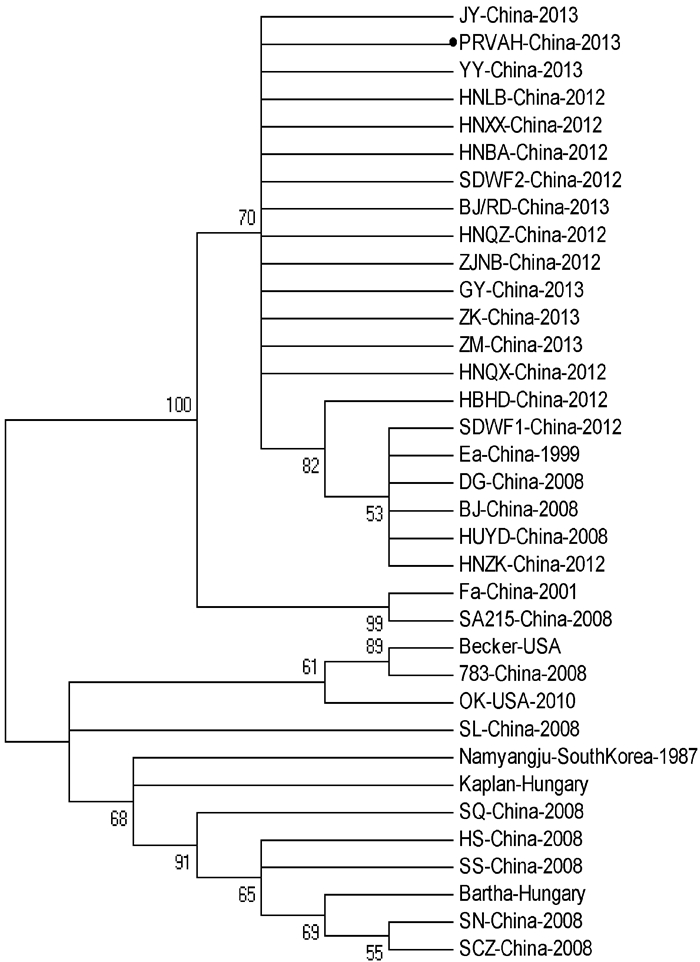
分别以PRV gC和gE基因对新流行株与其他毒株进行遗传进化分析。在PRV gC基因的遗传进化分析中,采用了30株国内不同年代或不同地点分离的PRV毒株和5株国外分离的PRV毒株,结果表明, 除2008年分离的7株毒株外,其他国内毒株都位于同一个分支,且2011年PRV重新大流行以来新分离毒株的遗传关系更近,这些毒株与国外毒株及疫苗毒株Bartha株遗传关系较远(图 1),这与Fonseca等[7]的结果相符。本研究中所分离的毒株PRV AH与2011年PRV重新大流行以来分离的PRV毒株位于同一分支,因此归属于新流行株。
|
图 1 PRV gC基因的遗传进化分析 Figure 1 Phylogenetic analysis based on PRV gC |
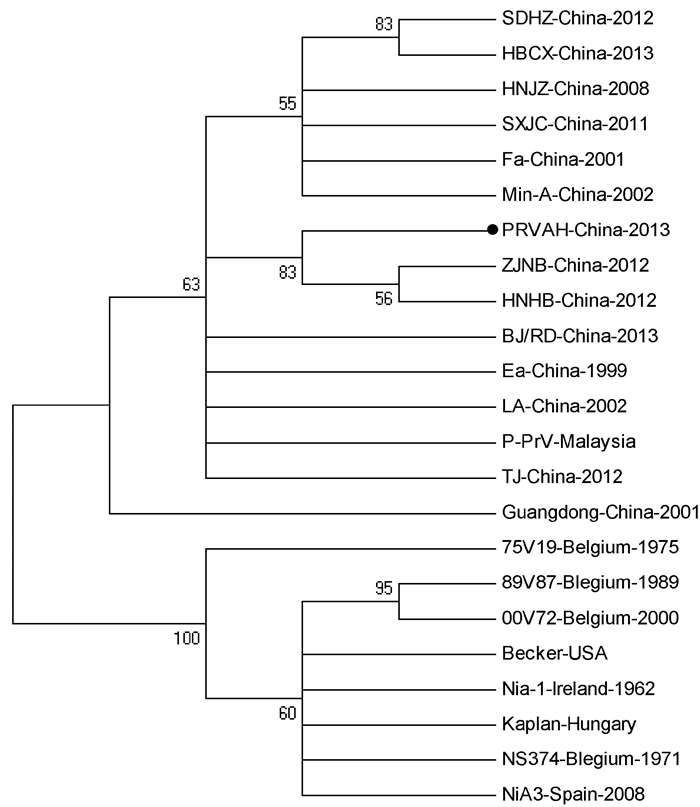
PRV gE基因的遗传进化分析结果与gC基因的相似。在PRV gE基因的遗传进化分析中,采用了14株国内不同年代或不同地点分离的PRV毒株和9株国外PRV毒株,结果显示,所有国内毒株与国外毒株(P-PrV-Malaysia株除外)分属于不同分支,说明国内分离毒株遗传关系较近,而与国外毒株存在较大差异(图 2)。
|
图 2 PRV gE基因的遗传进化分析 Figure 2 Phylogenetic analysis based on PRV gE |
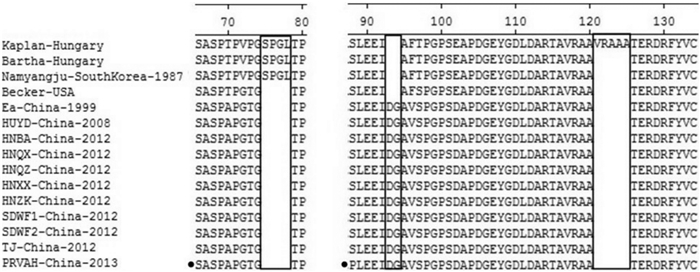
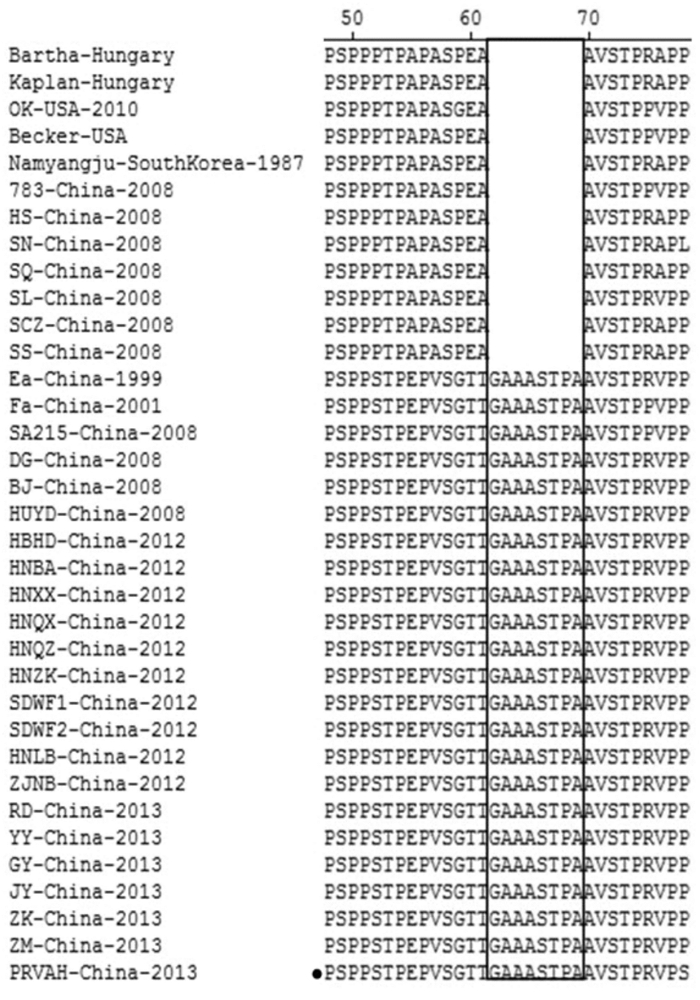
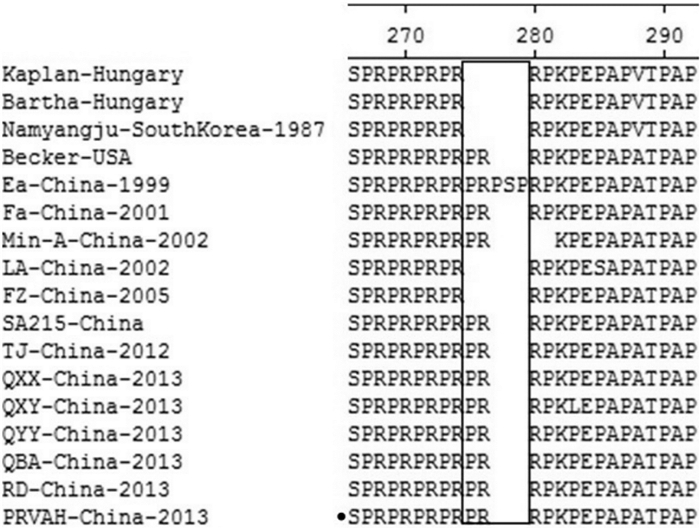
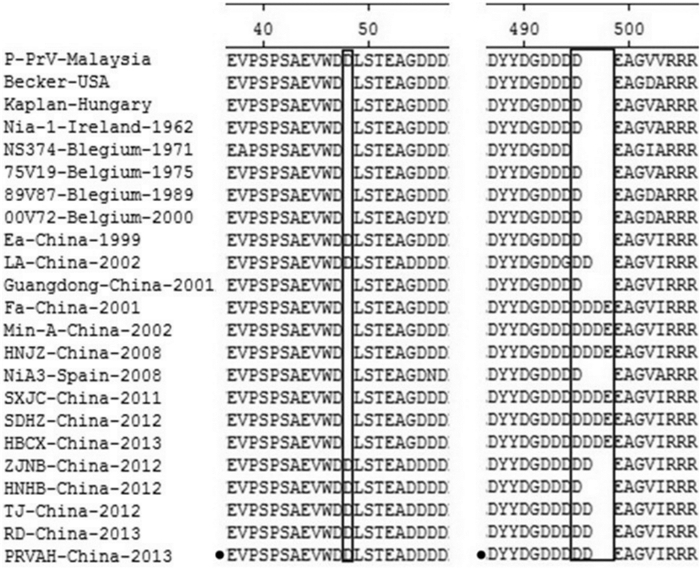
将PRV国内分离株的主要糖蛋白序列与Bartha株及国外分离株进行比较,结果显示, 国内株都存在明显的氨基酸的缺失或插入。在糖蛋白gB区域,所有国内株在75~78氨基酸处存在4个氨基酸的缺失,在93~94氨基酸处存在2个氨基酸的插入(图 3)。在糖蛋白gC区域,国内毒株(除2008年分离的7株毒株外)在62~69氨基酸处都存在8个连续氨基酸的插入(图 4)。与Bartha株相比较,多数国内株在糖蛋白gD的275~276氨基酸处存在2个氨基酸的插入,而PRV Ea株在275~279氨基酸处存在5个连续氨基酸的插入(图 5)。在糖蛋白gE区域,不同分离株的区别主要位于48氨基酸和495~498氨基酸处,如与国外经典毒株Kaplan株和Becker株相比较,国内新分离株(除HNHB株外)在496~498位置均存在1~3个氨基酸的插入,部分国内株在48氨基酸处还存在1个氨基酸的插入(图 6)。
|
图 3 不同PRV毒株gB氨基酸的序列比对分析 Figure 3 Comparison of gB glycoprotein amino acid sequences of different PRV strains |
|
图 4 不同PRV毒株gC氨基酸的序列比对分析 Figure 4 Comparison of gC glycoprotein amino acid sequences of different PRV strains |
|
图 5 不同PRV毒株gD氨基酸的序列比对分析 Figure 5 Comparison of gD glycoprotein amino acid sequences of different PRV strains |
|
图 6 不同PRV毒株gE氨基酸的序列比对分析 Figure 6 Comparison of gE glycoprotein amino acid sequences of different PRV strains |
PRV AH株从安徽某Bartha株疫苗免疫猪场的流产胎儿脑组织中分离得到,经动物试验测得其毒力较强,对小鼠LD50可达104.3 TCID50。为了了解PRV AH株和国内外其他PRV毒株的关系,我们分别以PRV gC和gE基因进行了遗传进化分析,结果显示PRV AH株和2011年造成国内PRV大流行的毒株十分相似。
伪狂犬病毒的糖蛋白不仅在病毒的侵入、释放以及细胞间传播等过程中起重要作用,而且也是参与机体免疫反应的主要靶蛋白。因此,对这些糖蛋白进行深入分析有助于了解新毒株的致病性及免疫原性等。对PRV gB、gC、gD、gE糖蛋白的序列分析表明,国内不同时期分离的毒株,特别是新毒株序列基本相似,但与Bartha株存在明显差异,主要表现为多个连续氨基酸的插入或缺失,且部分差异区域位于这些糖蛋白的重要功能区。
在PRV gB糖蛋白N端的59~126氨基酸序列存在3个连续的抗原表位区,这一区域的氨基酸序列具有高柔韧性、易接近性和亲水性,具有抗体结合位点(ABD)的特征[8]。在本研究中,与Bartha株相比,所有国内毒株均存在4个氨基酸的缺失(75~78氨基酸)和2个氨基酸的插入(93~94氨基酸),这些突变都在59~126氨基酸区域,可能会影响抗原-抗体的结合。
PRV gC基因在诱导感染动物体液免疫和细胞免疫中起着重要作用。在猪群,无论是野毒感染还是弱毒疫苗免疫,机体诱导产生的PRV中和抗体主要是针对gC糖蛋白。此外,gC糖蛋白是介导病毒与靶细胞的黏附所必须,主要通过其表面的3个肝素结合区域(HBDs)与靶细胞表面硫酸乙酰肝素糖胺聚糖链结合。HBDs区域同时也是gC糖蛋白N端的抗体结合区域(44~290氨基酸)的一部分,用gC特异性抗体结合该区域可干扰病毒的黏附作用[9]。在本研究中,与Bartha株相比,国内新毒株的gC氨基酸序列在ABD区域均存在8个连续氨基酸的插入,这可能会改变该区的环状结构,从而影响gC抗体与其有效结合。这可能也是造成Bartha株疫苗对当前PRV流行株免疫效果低下的原因之一。
PRV gD糖蛋白可介导细胞间的融合,主要通过与细胞表面被称为疱疹病毒侵入介质(Herpesvirus entry mediators, Hve)的受体发生结合,以稳定病毒与细胞的相互作用。gD糖蛋白也是诱导产生中和抗体的靶蛋白之一,这种抗体可减少PRV侵入细胞,从而在抗病毒免疫中发挥重要作用[10]。在本研究中,与Bartha株相比,国内PRV新毒株的gD氨基酸序列在267~276氨基酸处存在(Pro-Arg)5 序列,而在Bartha株是(Pro-Arg)4序列,且在这一区域脯氨酸的连续间隔排列可以在蛋白表面形成严格的亲水结构,从而有利于蛋白之间的相互作用[11]。这些特殊氨基酸序列在PRV感染中的具体作用尚不清楚。国内毒株在gD蛋白中的这些突变是否会影响其与Bartha株疫苗抗体的有效结合尚需进一步证实。
PRV gE糖蛋白在介导细胞融合、病毒扩散和释放等方面起着重要作用[12-13],同时gE基因也与PRV的毒力相关[14-15]。在本研究中,国内毒株gE氨基酸序列的主要区别在于48位和496~498位存在不同数量氨基酸的插入,这些插入可能会导致病毒毒力的改变。
综上所述,与Bartha株相比,国内PRV毒株的主要免疫原性蛋白gB、gC、gD均存在明显差异,这也可能是影响Bartha株疫苗在国内猪场免疫效果不佳的原因之一。在2011年后国内分离的PRV毒株中,除gE蛋白外,糖蛋白gB、gC、gD无明显突变,且与国内经典毒株Ea、Fa?#199;A215等基本相似。因此,国产疫苗,如由Ea株致弱的HB-98毒株,可能可以提供更好的免疫保护,但国产疫苗生产工艺等方面仍需改进以提高疫苗的质量。此外,疫苗的免疫效果很大程度上取决于猪群本身的状况,例如疾病、营养缺乏和药物滥用等因素导致动物机体的免疫抑制也可能导致疫苗免疫失败。
| [1] |
AN T Q, PENG J M, TIAN Z J, et al. Pseudorabies virus variant in Bartha-K61-vaccinated pigs, China, 2012[J]. Emerg Infect Dis, 2013, 19(11): 1749-1755. DOI:10.3201/eid1911.130177 (  0) 0) |
| [2] |
YU X, ZHOU Z, HU D, et al. Pathogenic pseudorabies virus, China, 2012[J]. Emerg Infect Dis, 2014, 20(1): 102-104. DOI:10.3201/eid2001.130531 (  0) 0) |
| [3] |
WU R, BAI C, SUN J, et al. Emergence of virulent pseudorabies virus infection in Northern China[J]. J Vet Sci, 2013, 14(3): 363-365. DOI:10.4142/jvs.2013.14.3.363 (  0) 0) |
| [4] |
WANG C, YUAN J, QIN H, et al. A novel gE-deleted pseudorabies virus (PRV) provides rapid and complete protection from lethal challenge with the PRV variant emerging in Bartha-K61-vaccinated swine population in China[J]. Vaccine, 2014, 32(27): 3379-3385. DOI:10.1016/j.vaccine.2014.04.035 (  0) 0) |
| [5] |
彭金美, 安同庆, 赵鸿远, 等. 猪伪狂犬病病毒新流行株的分离鉴定及抗原差异性分析[J]. 中国预防兽医学报, 2013, 35(1): 1-4. DOI:10.3969/j.issn.1008-0589.2013.01.01 (  0) 0) |
| [6] |
POMERANZ L E, REYNOLDS A E, HENGARTNER C J. Molecular biololgy of pseudorabies virus: Impact on neurovirology and veterinary medicine[J]. Microbiol Mol Biol R, 2005, 69(3): 462. DOI:10.1128/MMBR.69.3.462-500.2005 (  0) 0) |
| [7] |
FONSECA A J, CAMARGOS M F, DE OLIVEIRA A M, et al. Molecular epidemiology of Brazilian pseudorabies viral isolates[J]. Vet Microbiol, 2010, 141(3/4): 238-245. (  0) 0) |
| [8] |
ZARIPOV M M, MORENKOV O S, FODOR N, et al. Distribution of B-cell epitopes on the pseudorabies virus glycoprotein B[J]. J Gen Virol, 1999, 80(3): 537-541. DOI:10.1099/0022-1317-80-3-537 (  0) 0) |
| [9] |
OBER B T, TEUFEL B, WIESMUELLER K, et al. The porcine humoral immune response against pseudorabies virus specifically targets attachment sites on glycoprotein gC[J]. J Virol, 2000, 74(4): 1752-1760. DOI:10.1128/JVI.74.4.1752-1760.2000 (  0) 0) |
| [10] |
ELOIT M, BOUZGHAIA H, TOMA B. Identification of antigenic sites on pseudorabies virus glycoprotein gp50 implicated in virus penetration of the host cell[J]. J Gen Virol, 1990, 71(9): 2179-2183. DOI:10.1099/0022-1317-71-9-2179 (  0) 0) |
| [11] |
PETROVSKIS E A, TIMMINS J G, ARMENTROUT M A, et al. DNA sequence of the gene for pseudorabies virus gp50, a glycoprotein without N-linked glycosylation[J]. J Virol, 1986, 59(2): 216-223. (  0) 0) |
| [12] |
ZSAK L, METTENLEITER T C, SUGG N, et al. Release of pseudorabies virus from infected cells is controlled by several viral functions and is modulated by cellular components[J]. J Virol, 1989, 63(12): 5475-5477. (  0) 0) |
| [13] |
ZSAK L, ZUCKERMANN F, SUGG N, et al. Glycoprotein gI of pseudorabies virus promotes cell fusion and virus spread via direct cell-to-cell transmission[J]. J Virol, 1992, 66(4): 2316-2325. (  0) 0) |
| [14] |
KIMMAN T G, DE WIND N, OEI-LIE N, et al. Contribution of single genes within the unique short region of Aujeszky's disease virus (suid herpesvirus type 1) to virulence, pathogenesis and immunogenicity[J]. J Gen Virol, 1992, 73(2): 243-251. DOI:10.1099/0022-1317-73-2-243 (  0) 0) |
| [15] |
TIRABASSI R S, TOWNLEY R A, ELDRIDGE M G, et al. Characterization of pseudorabies virus mutants expressing carboxy-terminal truncations of gE: Evidence for envelope incorporation, virulence, and neurotropism domains[J]. J Virol, 1997, 71(9): 6455-6464. (  0) 0) |
 2016, Vol. 37
2016, Vol. 37