2. 华南农业大学 动物科学学院/广东省农业动物基因组学与分子育种重点实验室,广东 广州 510642
2. College of Animal Science, South China Agricultural University/Guangdong Provincial Key Laboratory of Agro-animal Genomics and Molecular Breeding, Guangzhou 510642, China
FAM213B (Family with sequence similarity 213, member B),又称为前列腺酰胺合成酶(Prostamide F synthase,PGFS),属于硫氧还蛋白超家族成员,较早在小鼠和猪的大脑中发现,并推测在中枢神经系统起着重要的作用[1]。FAM213B也是前列腺素(Prostaglandin, PG)合成过程中一种重要的催化酶。PG的生物合成是一个复杂的过程,涉及到多个酶促反应以及中间产物的形成(http://www.kegg.jp/pathway/hsa00590):首先,作为PG合成的起始物之一,花生四烯酸(Arachidonic acid,ARA)在酶催化下被转变成前列腺素H2(Prostaglandin H2,PGH2),PGH2作为一种不稳定的中间产物,很快在FAM213B的作用下生成更为稳定的花生四烯酸酯或者前列腺素E2α(Prostaglandin E2,PGE2),以及向前列腺素F2α(Prostaglandin F2α,PGF2α)的转变;FAM213B还能够直接催化PGH2生成PGF2α[2],因此FAM213B具有双重催化作用。
在FAM213B催化PG合成过程中,PGE2、PGH2与PGF2α是重要的中间产物。PG是哺乳动物重要的生殖激素,参与母猪发情周期、妊娠识别和建立等生理活动。不同来源、不同类型的PG具有不同的生物学作用,在动物繁殖过程中具有调节作用的主要是PGE2和PGF2α[3]。PGE2和PGF2α之所以受到关注是因为两者具有相反的生理功能,PGF2α是一种溶解黄体因子[4],而PGE2则能够抑制黄体溶解[5-6], 因此PGE2/PGF2α须受到严格控制,恰当的比例维持着母猪的发情周期和妊娠[3, 7]。较高比例的PGE2/PGF2α对于母猪妊娠的建立和维持具有重要作用,比如中国高繁殖力母猪梅山猪[8]和二花脸猪[9],相比外种猪,均表现较高的PGE2/PGF2α比例。由于PGF2α具有溶解黄体作用,因此在怀孕母猪体内,PGF2α的表达量受到严格控制。从上述PGF2α的生成过程来看,FAM213B可能间接影响PGE2/PGF2α比例。
目前,FAM213B作为催化PGH2直接生成PGF2α的酶相关研究较少。显然,PGH2直接生成PGF2α,比PGH2生成PGE2再生成PGF2α过程更为简单,对于PGF2α的高效快速生成具有重要意义。鉴于PGF2α的溶解黄体作用,可以推测FAM213B可能在母猪的发情周期中起着重要的催化作用,或者FAM213B含量在母猪怀孕阶段可能受到严格的调控。但是,FAM213B也能够催化PGH2向PGE2的转化,而PGE2与PGF2α对黄体具有相反的生理功能,因此FAM213B的表达可能会受到更严格的调控。为了深入研究FAM213B在母猪PG合成中的作用,本研究对母猪FAM213B基因进行了全长mRNA的克隆,并在此基础上对启动子进行了克隆和转录活性的初步分析,期望为进一步研究猪FAM213B基因的表达调控和功能奠定基础。
1 材料与方法 1.1 试验材料动物组织与细胞:用于提取DNA和RNA的耳组织以及猪子宫内膜组织,均来源于妊娠12 d(GD12) 的第3胎长大杂交母猪(LL),样品采自阳江市某猪场;猪子宫内膜细胞的采集来源于广州市某屠宰场长大淘汰母猪。
菌株、质粒和限制性内切酶及其他:大肠埃希菌菌株DH5α和pMD20T载体购自TaKaRa公司,pGL3-Basic载体、pRL-TK载体、限制性内切酶购自Promega公司。Taq DNA聚合酶、dNTPs、DL2000分子量标准、DNA凝胶回收试剂盒等购自广东东盛生物技术公司;T4 DNA连接酶、RNA提取试剂Trizol、反转录试剂盒、RACE kit试剂盒购自TaKaRa公司。无内毒素质粒小提试剂盒购自OMEGA公司。
1.2 猪FAM213B基因完整mRNA序列的克隆根据GenBank上公布的猪FAM213B基因(XM_005664949.2) CDS区的序列,设计引物,用于完整mRNA序列的克隆。使用5′-Full RACE kit获得基因的5′UTR序列,主要步骤包括:使用烟草酸焦磷酸酶(Tobacco acid pyrophosphatase,TAP)去掉mRNA的5′帽子结构,使用T4 RNA Ligase将5′RACE Adaptor连接到mRNA的5′端后,使用5′RACE Adaptor上的引物(p-5′Outer、p-5′Inner)与已知序列部分的引物(5′GSP1、5′GSP2) 进行巢式PCR反应,特异性地扩增cDNA 5′末端的全长序列,最后进行测序。3′UTR的获得过程包括:由ReverseTranscriptaseM-MLV(RNaseH-)将poly(A)+RNA反转录成cDNA,然后再使用TaKaRaLATaq,以反转录的cDNA为模板,利用试剂盒自带引物(p-3′Outer、p-3′Inner)和已知序列部分引物(3′GSP1、3′GSP2) 进行巢式PCR反应,上述过程均按照使用说明书进行,获得完整3′UTR序列。将获得序列进行拼接,获得完整mRNA序列,提交GenBank,获得基因登录号。根据获得序列设计引物:p-mRNA-F和p-mRNA-R,以猪子宫内膜总RNA为模板,PCR反应后,进行T-A克隆后测序,以此验证所拼接序列的正确性。具体的引物序列见表 1。
|
|
表 1 猪FAM213B基因mRNA全序列和启动子克隆所用引物信息 Table 1 The primers for cloning the mRNA and promoter of porcine FAM213B gene |
参照本实验室获得的猪FAM213B基因mRNA序列(KX444503) 以及5′调控区序列(GenBank登录号:100134955),设计引物(表 1引物:p-promoter-F和p-promoter-R),扩增获得猪FAM213B基因启动子区2 261 bp,将此构建至T载体,测序后,命名为T-FAM213B-P。以此载体为模板,以带有酶切位点的引物(p0-promoter-F和p0-promoter-R)扩增启动子区,PCR扩增反应程序为:98 ℃预变性1 min;98 ℃变性10 s,67 ℃退火10 s,72 ℃延伸2.5 min,33个循环;72 ℃后延伸10 min。
将上述PCR扩增获得DNA片段用DNA凝胶回收试剂盒进行回收纯化后,用Mlu I和Xho I进行双酶切,同时对pGL-Basic载体进行酶切,酶切体系为:10×Buffer 10 μL,PCR产物或者载体50 μL,Mlu I和Xho I各5 μL,ddH2O 30 μL,37 ℃反应3 h。将双酶切后的DNA片段和pGL-Basic载体分别用DNA凝胶回收试剂盒进行回收纯化,2个DNA片段进行连接,10 μL连接反应体系中,依次加入1 μL Ligation Buffer,2 μL pGL3-basic载体双酶切产物,6.8 μL启动子片段,0.2 μL T4 DNA连接酶,22 ℃连接4 h。转化大肠埃希菌DH5α,涂布氨苄青霉素琼脂平板,37 ℃过夜培养,然后挑取单克隆进行测序,阳性质粒命名为P0。
1.4 猪子宫内膜细胞的分离与培养屠宰场采集卵泡期子宫,所采样品于切口处结扎后,放入冰盒,迅速带回实验室。纵向剪开子宫角,置于无菌培养皿中,用含有双抗的PBS中清洗数次,直至液体清亮。剪取子宫内膜上皮组织于含有双抗的PBS中,漂洗数次。
用眼科剪将子宫内膜上皮组织剪成1 mm3大小。将剪碎的组织块转移至培养瓶中。加入2倍体积的1 g·L-1胶原酶I,37 ℃水浴摇床消化,每隔0.5 h混摇1次。2.5 h后加入等体积含100 g·L-1 FBS的DMEM/F12的终止液进行终止消化。把消化液和终止液的混合液用150 μm(100目)细胞筛过滤,吸取滤液至离心管中,500 r·min-1离心5 min(沉淀主要为上皮细胞,上清主要为基质细胞)。
上皮细胞与基质细胞通过1 000 r·min-1离心5 min和PBS重悬后,将沉淀细胞计数,然后以2×105个细胞·mL-1接种于细胞培养瓶中,于37 ℃,CO2培养箱中静置培养(含体积分数为5%的CO2和95%的空气)。原代细胞在接种后24 h左右贴壁,约3~5 d达到90 %融合。此时,吸出培养液,用PBS漂洗2次后,加入2.5 g·L-1胰蛋白酶消化3~5 min。在显微镜下观察,待大部分细胞变圆时,立即加入等体积的终止液终止消化,轻轻吹打数次。将细胞悬液转移至离心管中,1 000 r·min-1离心5 min后弃上清,再加入5 mL PBS重悬细胞,1 000 r·min-1离心5 min后弃上清,接种于新的培养瓶继续培养,备用。
将上述构建好的载体用OMEGA无内毒素试剂盒提取后,与pRL-TK质粒用阳离子脂质体法转染细胞,48 h后,用双荧光素酶报告基因系统进行Luciferase活性检测,收集数据分析启动子片段转录活性。
1.5 生物信息学分析蛋白基本理化性质预测:http://www.expasy.org/cgi-bin/protscale.pl,ExPASy的ProtScal程序;蛋白质三级结构预测:http://swissmodel.expasy.org/;蛋白质结构域和基序预测:http://smart.embl-heidelberg.de的UniProt程序;序列比对工具:Kalign和Clustal Omega。
2 结果与分析 2.1 猪FAM213B基因完整mRNA序列的克隆将提取的猪子宫内膜组织总RNA经过去磷酸化处理和去帽反应,连接5′RACE adaptor后反转录成cDNA,以该cDNA为模板,进行引物p-5′Outer和5′GSP1的PCR扩增,此时产物条带弥散不清,如图 1A。再以此PCR产物为模板,以引物p-5′Inner和5′GSP2进行PCR扩增,此时得到单一的目的条带209 bp,如图 1B。将目的条带连接载体后测序,将所得序列与已知序列比对,拼接后得到了67 bp的5′UTR,即转录起始位点位于ATG前的67 bp处。同样,以总RNA为模板,用ployT引物将RNA反转录成cDNA,接着用p-3′Outer和3′GSP1引物进行PCR扩增,此时产物条带弥散不清,如图 1C。再以此为模板,以p-3′Inner和3′GSP2引物进行PCR扩增,此时得到单一目的条带600 bp,如图 1D。将目的条带连接载体后测序,所得序列与已知序列比对,拼接后得到了132 bp的3′UTR,以及10 bp的ployA序列。
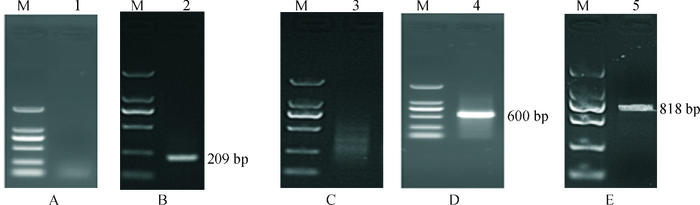
|
图 1 猪FAM213B基因完整mRNA序列的RACE结果 Figure 1 The RACE results of porcine FAM213B mRNA sequence M为DL2000 Marker,从上到下为:2 000、1 000、750、500、250和100 bp;1: 5′RACE Outer引物扩增弥散带;2: 5′RACE Inner引物扩增特异性条带;3:3′RACE Outer引物扩增弥散带;4:3′RACE Outer引物扩增特异性条带;5:FAM213B基因mRNA验证扩增结果。 |
经过测序后,以5′UTR和3′UTR序列设计引物p-mRNA-F和p-mRNA-R,以长大杂母猪子宫内膜组织cDNA为模板,通过PCR扩增获得完整的mRNA序列,并经过测序确定与RACE技术获得的mRNA序列完全一致,为808 bp,polyA长度为10 bp(见图 1E)。将该序列提交GenBank,获得登录号为:KX444503。
2.2 猪FAM213B性质以及不同转录本和蛋白质结构比较用ExPASy的ProtScal程序分析获得猪FAM213B基因mRNA CDS编码蛋白的理化性质,发现该蛋白的分子量为21 546.8,等电点为8.87,半衰期为30 h,不稳定系数为61.27(大于40为不稳定),为一不稳定性蛋白。用UniProt程序分析该蛋白的结构域,发现在17~106位氨基酸之间存在硫氧还蛋白折叠结构域。GO分析表明,该蛋白作为细胞质成分,参与了氧化还原反应过程(GO:0055114),具有催化活性。
猪FAM213B基因定位于6号染色体,基因全长3 138bp(GenBank登录号:NC_010448.3), NCBI数据库公布的猪FAM213B基因的mRNA序列有3个,其中1个序列为609 bp(NM_001113441.1),是一个完整的阅读框,不含非翻译区,将其序列与本研究获得的mRNA的CDS区进行比对,两者序列一致。另外2个序列是根据生物信息学计算得出的转录本,分别为940 bp(XM_013998922.1) 和824 bp(XM_005664949.2)。鉴于人FAM213B基因存在多个转录本,进而将本研究获得猪FAM213B基因mRNA序列与940和824 bp转录本序列进行比较(在线软件:Kalign),结果表明所获得的FAM213B基因mRNA与另外2个转录本的非翻译区、CDS区长度存在差异,结果如图 2所示。808、824、940 bp的转录本CDS区分别长:609、555和711 bp,编码氨基酸数目为:203、185和237。进一步与基因组序列进行比对分析, 发现猪FAM213B基因由7个外显子组成,本研究所获得的mRNA序列与其他2个较长的mRNA序列相比,发现在2、3和4外显子不存在差异,而在第1、5、6和7外显子存在差异,1和7外显子的差异在于UTR长度不同;此外,本研究获得序列第5、6外显子分别为69、132 bp,而940 bp序列第5外显子159 bp,而824 bp序列第5、6外显子分别为72和94 bp。
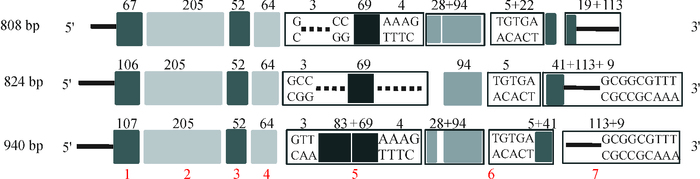
|
图 2 猪FAM213B基因mRNA不同转录本序列与结构分析 Figure 2 The genetic structures of three transcripts of porcine FAM213B gene 实横线:UTR;方框:编码区;虚线:该处不存在碱基;上方数字表示碱基数目,下方红色数字1~7表示外显子序号;808、824和940 bp分别表示3个转录本碱基数目。 |
通过在线工具预测3个转录本蛋白质三级结构(http://swissmodel.expasy.org/),结果如图 3所示。3个蛋白均有α螺旋、β折叠、转角等结构组成,由于3个转录本编码区前108个氨基酸序列一致,因此三级结构存在类似空间构象,主要由α螺旋组成;而三者的氨基酸序列、数目不一致,因此蛋白质三级结构差异较大。在这些氨基酸44位处为半胱氨酸(Cys44),这是该蛋白的活性位点;同时,三者都存在硫氧还蛋白折叠结构域。
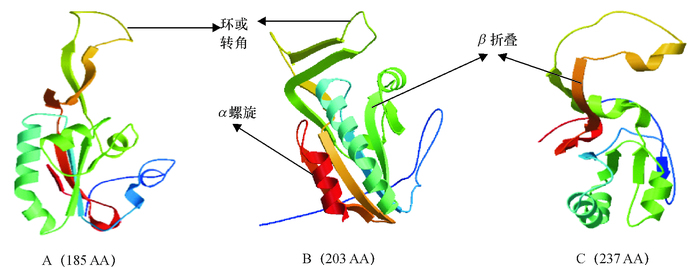
|
图 3 猪FAM213B基因不同转录本的蛋白质三级结构分析 Figure 3 The protein tertiary structures of three transcripts of porcine FAM213B gene A、B、C分别对应于图 2中3个转录本的蛋白结构,括号内数字表示3个转录本编码氨基酸数目。 |
通过Clustal Omega软件,将人(NP_689584.2)、猪(KX444503)、牛(NP_001035688.1)、山羊(XP_005690822.2)、绵羊(XP_014954752)、小鼠(NP_079858.2)、大鼠(NP_001102167.1)、斑马鱼(NP_998478.1)、爪蟾(NP_001087128.1)9个物种FAM213B基因开放阅读框转录所得氨基酸序列进行蛋白质同源性分析,猪与山羊、牛和绵羊有更高的相似性,相似性分别为94.03%、93.03%和91.54%;与人、小鼠和大鼠相似性为88.38%、86.57%和85.57%;与爪蟾、斑马鱼相似性分别为61.19%和59.70%。根据氨基酸相似性分析结果,用邻接法构建FAM213B蛋白质同源进化分支图(图 4)。
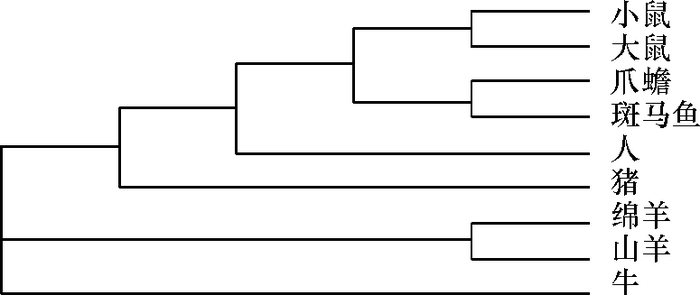
|
图 4 基于FAM213B蛋白质序列的聚类分析 Figure 4 Cluster analysis based on FAM213B protein sequences |
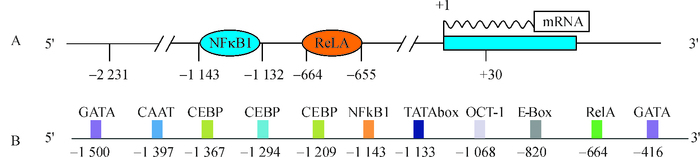
将5′RACE试验获得的mRNA序列的第1个碱基确定为+1,通过PCR扩增获得2 261 bp(-2 231/+30) 的猪FAM213B基因5′启动子序列(图 5A)。将猪FAM213B基因启动子区序列通过在线生物学软件进行分析,预测启动子区潜在的顺式作用元件和结合反式作用因子,发现FAM213B基因启动子区存在潜在的转录因子结合位点(图 5B),如典型的TATA box、CAAT box等顺式作用元件;存在与下列转录因子如cAMP应答元件结合蛋白(cAMP-response element binding protein,CREB)、CCAAT增强子结合蛋白(CCAAT/enhancer binding protein, C/EBP)、E-box结合因子(E-box binding factors)、核因子κB(Nuclear factor κB, NFκB)、八聚体转录因子(Octamer transcription factor-1, OCT-1) 等潜在结合位点。其中,NFκB成员NFκB1和RelA分别位于:-1 143/-1 132和-664/-655区域(图 5A)。根据在线预测结果,以2 261 bp(-2 231/+30) 的片段(图 6A)为模板,用p0-promoter-F和p0-promoter-R引物对扩增-2 175/+30区域(图 6B),并将其连接构建报告基因重组载体,酶切鉴定结果符合预期(图 6C)。
|
图 5 猪FAM213B基因mRNA结构示意图及启动子顺式作用元件与转录因子预测结果 Figure 5 The structure of the FAM213B mRNA and prediction of cis-acting elements and transcription factors in FAM213B gene promoter A:猪FAM213B基因启动子与mRNA结构示意图;B:猪FAM213B基因启动子长顺式作用元件及结合因子预测结果。 |

|
图 6 猪FAM213B基因启动子报告基因重组载体的构建 Figure 6 The bioinformatics analysis and the construction of the reporter gene recombinant vector of FAM213B gene promoter M为DS5000 Marker,从上到下为:5 000、3 000、2 000、1 500、1 000、750、500、250和100 bp;1:2 261 bp启动子片段;2:2 205 bp启动子片段;3:2 205 bp片段重组载体双酶切结果。 |
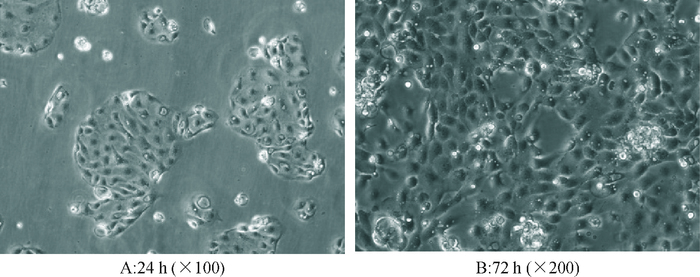
采用胶原酶消化法,分离培养上皮细胞和间质细胞。猪子宫内膜上皮细胞大概在24 h左右开始贴壁并成旋涡状排列的致密单层细胞集落,采用离心的方法并不能够将上皮细胞和间质细胞完全分离,因而偶见散在的间质细胞插入性生长(图 7A)。虽然间质细胞在数小时内开始贴壁成单个生长,但并不影响启动子活性的检测。子宫内膜上皮细胞在3~5 d后可达到80%融合,上皮细胞呈梭形,边界清楚,排列紧密,胞质呈颗粒状,胞核大而圆,铺满瓶底时,细胞集落扩大并相互融合成片(图 7B)。
|
图 7 猪子宫内膜上皮细胞培养形态 Figure 7 Appearance of the cultured endometrial epithelial cells |
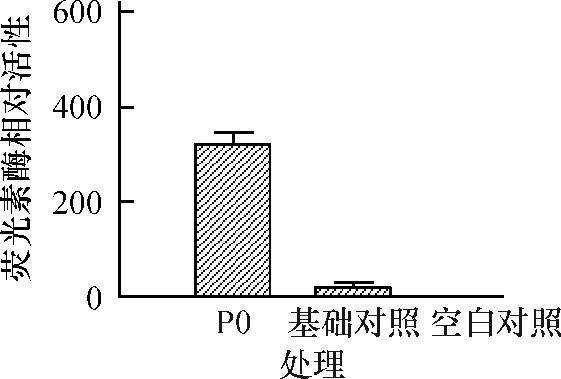
采用瞬时转染方法,将猪FAM213B基因启动子报告基因重组载体P0转染猪子宫内膜上皮细胞,同时共转染内参pGL-TK质粒。分别检测萤火虫荧光素酶和海肾荧光素酶的活性,萤火虫荧光素酶与海肾荧光素酶活性的比值即表示启动子片段的活性,启动子报告基因瞬时转染结果如图 8所示:重组质粒P0转录活性远高于对照,表明克隆所获得片段具有启动子转录活性。
|
图 8 FAM213B基因启动子在猪子宫内膜细胞转录活性 Figure 8 The transcriptional activity of the promoter of procine FAM213B gene |
前列腺酰胺合成酶GFS存在3种同工酶,分别是PGFS I、PGFS Ⅱ和FAM213B。前2种是醛酮还原酶(Aldo-keto reductase, AKR)超家族,FAM213B则属于硫氧还蛋白样超家族成员[10]。FAM213B是近年来新发现的一种氧化还原酶,这种新型酶不同于之前所报道的PGF酶。FAM213B蛋白质在NADPH作为质子供体情况下,能直接将PGH2做为底物一步合成PGF2α,并且FAM213B合成PGF2α效率优于其他PGF酶[1]。在通过5′RACE和3′RACE技术获得了猪FAM213B基因完整mRNA序列(GenBank序列号:KX444503) 的基础上,并确定了该基因的转录起始位点位于其起始密码子前67 bp。由于FAM213B基因在人上存在多个转录本,推测在猪上该基因也可能存在多个转录本。将获得的转录本序列与GenBank提交的FAM213B基因其他2个转录本(940 bp,XM_013998922.1;824 bp,XM_005664949.2) 进行了基因结构和蛋白三维结构比较,发现三者在1~108位氨基酸序列相同,而这在17~106位氨基酸之间存在硫氧还蛋白折叠功能结构域,这可能与其共同的催化功能有关。蛋白质三级结构虽然有较大差异,但是功能域形成的α螺旋基本一致,因此推测其不同的转录本的翻译蛋白其催化功能是一致的。不过本研究在猪子宫内膜仅发现了1种转录本,其他2个计算模拟的转录本尚未检测到,但是这并不意味着猪FAM213B基因只有1种转录本。目前,发现小鼠FAM213B基因只有1个转录本。对人、猪、牛、山羊、绵羊、小鼠、大鼠、斑马鱼、爪蟾等9个物种的FAM213B氨基酸序列相似性进行分析,猪与山羊、牛和绵羊有更高的相似性。同时,猪、人和小鼠的FAM213B基因结构相似,都由7个外显子和6个内含子组成,第2、第3、第4和第5外显子的长度在3个物种相同,第7外显子的长度差异较大。而比较有趣的现象是在公共数据库中尚未检索到家禽如鸡、鸭等物种FAM213B基因信息。
3.2 猪FAM213B基因启动子在猪子宫内膜的转录调控妊娠的建立需要功能性黄体持续分泌孕酮,而孕酮又可促进子宫内膜的分泌活动,这对胚胎附植及发育具有关键作用[11],PG与孕酮的溶解、维持密切相关。前人研究表明猪和小鼠FAM213B mRNA存在脑组织与脊髓中,而且主要在细胞质中表达,表明FAM213B在中枢神经系统发挥着重要的作用[1];前期研究发现,FAM213B在怀孕猪子宫内膜中表达[9],推测FAM213B基因在子宫内膜中对母猪妊娠和胚胎发育起着调控作用。FAM213B和CBR1/2均可以催化PGE2向PGF2α转变[12],但FAM213B也能够直接催化PGH2向PGF2α的转变,因此推测FAM213B在母猪妊娠期间的低催化效率对于较高PGE2/PGF2α的比率可能是必需的,FAM213B基因的mRNA和蛋白表达量在猪子宫内膜可能会受到严格控制。因此,本研究在RACE技术获得完整FAM213B基因mRNA基础上,精准定位其转录起始位点,并且发现获得的FAM213B基因启动子序列(-2 175/+30) 在猪子宫内膜中具有转录下游报告基因的活性。通过对猪FAM213B基因的启动子序列生物信息学分析,发现除了典型的TATA box、CAAT box等顺式作用元件外,还存在其他的一些潜在转录因子结合位点,如E-box、NFκB、OCT-1等,而这些因子也决定了FAM213B基因在猪子宫内膜中的表达规律。例如,母猪的妊娠建立类似一种炎性过程[13],而炎性因子NFκB对于妊娠相关基因的表达则具有促进或者抑制作用,在该启动子-1 143和-664位点预测到了NFκB1和RelA结合位点,NFκB1和RelA均为NFκB家族成员[14]。有研究表明,分泌期人类子宫NFκB1和RelA表达上调[15];RelA表达量在胚胎植入窗口期表达量也会升高[13]。有文献报道,在母猪胚胎植入前NFκB就被开始被激活[16],NFκB常常作为转录因子激活炎性相关基因的表达[17]。
本研究发现FAM213B基因的启动子上存在潜在的NFκB和RelA结合位点,推测FAM213B基因可能会受到NFκB的调控,不过需要进行深入研究。总之,初步检测到所获启动子片段具有转录活性,而且发现与母猪妊娠密切相关的潜在顺式作用元件,这些结果为进一步分析猪FAM213B基因转录活性区域、特异性转录因子结合位点鉴定等研究奠定了基础。
| [1] |
MORIUCHI H, KODA N, OKUDA-ASHITAKA E, et al. Molecular characterization of a novel type of prostamide/prostaglandin F synthase, belonging to the thioredoxin-like superfamily[J]. J Biol Chem, 2008, 283(2): 792-801. DOI:10.1074/jbc.M705638200 (  0) 0) |
| [2] |
KOMOTO J, YAMADA T, WATANABE K, et al. Prostaglandin F2alpha formation from prostaglandin H2 by prostaglandin F synthase (PGFS): Crystal structure of PGFS containing bimatoprost[J]. Biochemistry, 2006, 45(7): 1987-1996. DOI:10.1021/bi051861t (  0) 0) |
| [3] |
WACLAWIK A, ZIECIK A J. Differential expression of prostaglandin (PG) synthesis enzymes in conceptus during peri-implantation period and endometrial expression of carbonyl reductase/PG 9-ketoreductase in the pig[J]. J Endocrinol, 2007, 194(3): 499-510. DOI:10.1677/JOE-07-0155 (  0) 0) |
| [4] |
CHRISTENSON L K, FARLEY D B, Anderson L H, et al. Luteal maintenance during early pregnancy in the pig: Role for prostaglandin E2[J]. Prostaglandins, 1994, 47(1): 61-75. DOI:10.1016/0090-6980(94)90075-2 (  0) 0) |
| [5] |
ZIECIK A J. Old, new and the newest concepts of inhibition of luteolysis during early pregnancy in pig[J]. Domest Anim Endocrinol, 2002, 23(1/2): 265-275. (  0) 0) |
| [6] |
GADSBY J E, LOVDAL J A, BRITT J H, et al. Prostaglandin F2 alpha receptor concentrations in corpora lutea of cycling, pregnant, and pseudopregnant pigs[J]. Biol Reprod, 1993, 49(3): 604-608. DOI:10.1095/biolreprod49.3.604 (  0) 0) |
| [7] |
WEEMS C W, WEEMSY S, RANDELR D. Prostaglandins and reproduction in female farm animals[J]. Vet J, 2006, 171(2): 206-228. DOI:10.1016/j.tvjl.2004.11.014 (  0) 0) |
| [8] |
BAZER F W, THATCHER W W, MATINAT-BOTTEF, et al. Composition of uterine flushings from Large White and prolific Chinese Meishan gilts[J]. Reprod Fertil Dev, 1991, 3(1): 51-60. DOI:10.1071/RD9910051 (  0) 0) |
| [9] |
ZHANG H, WANG S, LIU M, et al. Differential gene expression in the endometrium on gestation day 12 provides insight into sow prolificacy[J]. Bmc Genomics, 2013, 14: 45. DOI:10.1186/1471-2164-14-45 (  0) 0) |
| [10] |
SUZUKI-YAMAMOTO T, TOIDA K, SUGIMOTO Y, et al. Colocalization of prostaglandin F(2alpha) receptor FP and prostaglandin F synthase-I in the spinal cord[J]. J Lipid Res, 2009, 50(10): 1996-2003. DOI:10.1194/jlr.M800543-JLR200 (  0) 0) |
| [11] |
ZIECIK A J, WACLAWIK A, KACZMAREK M M, et al. Mechanisms for the establishment of pregnancy in the pig[J]. Reprod Domest Anim, 2011, 46(Suppl 3): 31-41. (  0) 0) |
| [12] |
WACLAWIK A, JABBOUR H N, BLITEK A, et al. Estradiol-17beta, prostaglandin E2 (PGE2), and the PGE2 receptor are involved in PGE2 positive feedback loop in the porcine endometrium[J]. Endocrinology, 2009, 150(8): 3823-3832. DOI:10.1210/en.2008-1499 (  0) 0) |
| [13] |
KING A E, COLLINS F, KLONISCH T, et al. An additive interaction between the NFkappaB and estrogen receptor signalling pathways in human endometrial epithelial cells[J]. Hum Reprod, 2010, 25(2): 510-518. DOI:10.1093/humrep/dep421 (  0) 0) |
| [14] |
LINDSTROM T M, BENNETT P R. The role of nuclear factor kappaB in human labour[J]. Reproduction, 2005, 130(5): 569-581. DOI:10.1530/rep.1.00197 (  0) 0) |
| [15] |
LAIRD S M, TUCKERMAN E M, CORK B A, et al. Expression of nuclear factor kappaB in human endometrium; role in the control of interleukin 6 and leukaemia inhibitory factor production[J]. Mol Hum Reprod, 2000, 6(1): 34-40. DOI:10.1093/molehr/6.1.34 (  0) 0) |
| [16] |
MATHEW D J, SELLNER E M, GREEN J C, et al. Uterine progesterone receptor expression, conceptus development, and ovarian function in pigs treated with RU 486 during early pregnancy[J]. Biol Reprod, 2011, 84(1): 130-139. DOI:10.1095/biolreprod.110.086843 (  0) 0) |
| [17] |
INOUE J, GOHDA J, AKIYAMA T, et al. NF-kappaB activation in development and progression of cancer[J]. Cancer Sci, 2007, 98(3): 268-274. DOI:10.1111/cas.2007.98.issue-3 (  0) 0) |
 2017, Vol. 38
2017, Vol. 38


