流溪河流域是广州市饮用水的主要水源地之一,近年来,随着工业、城市污水以及周边畜禽养殖业污染的加剧,排放到流溪河水域中的可溶性无机氮含量严重超标,产生了水体富营养化等诸多问题[1]。反硝化作用作为生态系统中氮素循环的关键步骤,对水体中氮素的循环降解起着重要的调控作用[2]。因此,研究反硝化作用的过程及相关的微生物机制对缓解河流生态系统中的富营养化至关重要。
反硝化作用是指反硝化细菌还原硝酸盐,将其逐步转化为气态氮或氮氧化物的过程,主要由硝酸还原酶、亚硝酸还原酶、NO还原酶和N2O还原酶4种酶催化完成[3]。其中亚硝酸还原酶为关键限速酶,主要由nirS和nirK基因编码合成[3-4]。这2个基因经常被用作标志基因来研究环境中的反硝化作用,据报道在河流生态系统中nirS基因比nirK基因分布更广泛,更适合用于沉积物中反硝化作用的研究[5-8]。
本文以反硝化细菌亚硝酸还原酶的nirS基因作为标志基因,构建基因文库对流溪河流域沉积物中反硝化细菌的群落结构和多样性进行分析,结合实时荧光定量PCR(Real-time quantitative PCR, RT-qPCR)对反硝化细菌的丰度进行研究,并通过主坐标分析和冗余度分析等多变量统计分析方法研究反硝化细菌的群落结构、丰度与环境因子之间的相关性,探讨不同采样点反硝化细菌群落结构的差异性,了解反硝化细菌对环境因子的响应机制,为流溪河生态系统中氮素循环研究提供科学依据。
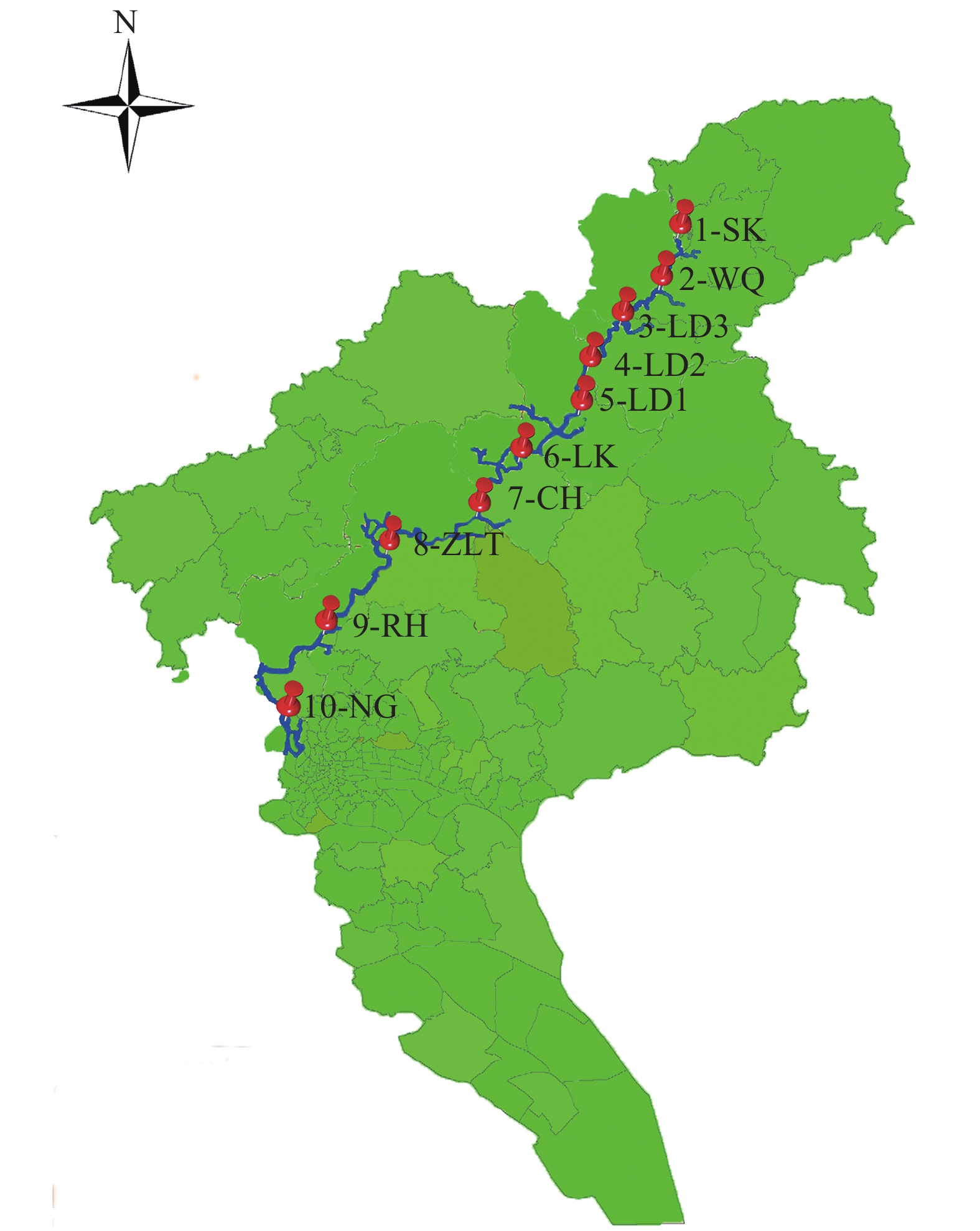
1 材料与方法 1.1 材料本试验在流溪河干流及支流上选取了10个采样点,从上游向下游依次标记为:水库(1–SK)、温泉(2–WQ)、绿道–3(3–LD3)、绿道–2(4–LD2)、绿道–1(5–LD1)、良口(6–LK)、从化(7–CH)、钟落潭(8–ZLT)、人和(9–RH)、南岗(10–NG)(图1)。使用自制的底泥采样器,在各个采样点采取水底表层(0~5 cm)的沉积物,采集后立刻放入无菌自封袋内,用干冰低温保存。每个采样点采集沉积物样品1 kg左右,返回实验室后保存于–80℃超低温冰箱。
|
图 1 采样点分布 Figure 1 Location of sampling sites in the Liuxi River |
|
|
表 1 流溪河中各采样点底泥理化性质 Table 1 Physical and chemical properties of sediments in sampling sites within the Liuxi River |
水温及pH采用便携式通用测量仪Multiline P4在现场进行测量,底泥中化学需氧量(Chemical oxygen demand,COD)、氨氮(NH4+–N)、亚硝酸盐氮(NO2––N)、硝酸盐氮(NO3––N)和总氮(Total nitrogen,TN)含量均参考中华人民共和国国家标准进行测定分析(表1)。COD、NH4+–N和TN分别参照GB11914—89、LY/T1231—1999和LY/T1228—1999/3进行测定;NO2––N和NO3––N参照HJ501—2009及HJ/T84—2001进行测定。
1.2.2 流溪河沉积物宏基因组DNA提取流溪河各采样位点沉积物样品的宏基因组DNA,采用强力土壤®DNA提取试剂盒(PowerSoil® DNA Isolation Kit,USA)提取,每个位点采集3个平行样,使用FD–1E–50型真空冷冻干燥机(Bilon)冻干,3个平行样混匀后称取0.25 g干土样品进行DNA提取。将提取的DNA用NanoDrop核酸蛋白仪测定浓度及质量,置于–20℃冰箱保存。
1.2.3 nirS基因PCR扩增使用cd3aF(5′-GTSAACGTSAAGGARACSGG-3′)和R3cd(5′-GASTTCGGRTGSGTCTTGA-3′)作为nirS基因序列PCR扩增的引物序列[9]。PCR反应程序为:95℃预变性2 min;95 ℃变性30 s,53 ℃退火1 min,72℃延伸1 min,35个循环;72 ℃延伸10 min。PCR扩增产物用10 g·L–1的琼脂糖凝胶电泳进行检验,目的条带大小在420 bp左右。
1.2.4 反硝化细菌nirS基因克隆及测序分析PCR扩增产物使用琼脂糖凝胶回收试剂盒(E.Z.N.A®Gel Extraction Kit,Omega)进行纯化,纯化后的产物采用pMD19-T Simple Vector连接试剂盒进行连接。将连接产物转化至大肠杆菌DH5α感受态细胞中,涂在LB固体选择培养基(含有100 μg·mL–1氨苄青霉素、40 μg·mL–1 IPTG及24 μg·mL–1X-gal)上,置于37 ℃条件下培养12~16 h后进行蓝白斑筛选,每个培养平板中随机挑取约100个白色菌斑,接种于含100 μg·mL–1氨苄青霉素的液体培养基中扩大培养,采用通用引物M13-D(5′-AGGGTTTTCCCAGTCACGACG-3′)和RV-M(5′-GAGCGGATAACAATTTCACACAGG-3′)进行菌落PCR验证,然后送往生工生物工程(上海)股份有限公司进行序列测定。
1.2.5 nirS基因丰度RT-qPCR分析nirS基因实时荧光定量采用绝对定量法,利用iQ5(Bio-Rad,USA)仪器测定分析。按照“1.2.4”方法进行nirS基因克隆,筛选阳性克隆子,挑取单菌落培养,采用质粒小提试剂盒(E.Z.N.A® Plasmid Miniprep Protocol,Omega)提取质粒。通过10倍稀释法将阳性克隆质粒稀释成6个浓度梯度,每个梯度设置3次重复,并设置阴性对照组,以不同浓度标准品作为模板进行RT-qPCR反应,绘制nirS基因荧光定量PCR标准曲线。引物序列与“1.2.3”相同。反应体系为20 μL:10 μL荧光染料SYBR Premix Mix Ex Taq Ⅱ,0.8 μL上、下游引物(10 μmol·L–1),7.4 μL ddH2O,1 μL模板DNA。反应条件为:94 ℃ 2 min (酶启动);95 ℃预变性 30 s,57 ℃退火30 s,72 ℃延伸45 s (36个循环,前6个循环退火温度每个循环降低1 ℃,后30个循环退火温度为52 ℃);72 ℃延伸10 min,最后以0.1 ℃·S–1速率从65 ℃升至95 ℃。nirS基因荧光定量PCR标准曲线的相关系数R2=0.995 2,扩增效率为109%。
1.2.6 数据处理采用MOTHUR软件对所有测序的nirS基因序列作OTU划分,并计算不同采样位点的nirS型反硝化细菌多样性指数,应用MEGA 6.0软件构建系统发育树。利用Canoco 4.5程序对nirS型反硝化群落分布进行主坐标分析(Principal coordinate analysis,PCoA),对群落–环境因子关系进行冗余分析(Redundancy analysis,RDA)。试验数据的相关性及方差分析采用SPSS 19.0软件进行。
2 结果与分析 2.1 流溪河nirS型反硝化细菌多样性分析本试验对流溪河10个采样点沉积物样品的宏基因组DNA进行PCR扩增及克隆子测序,共得到495条克隆序列,在83%相似度水平上共获得53个不同种类的操作分类单元(OTU)(表2)。对每个采样点进行多样性指数分析,发现各采样点的覆盖率均较高,表示测序所得到的OTU序列覆盖了沉积物中绝大部分的nirS序列。最上游水库位点的Shannon、Chao1、Pielou和ACE指数值最低,分别为0.16、2、0.23和2.0,Simpson指数值最高为0.92,表明该采样点的物种多样性最低;人和位点的Shannon、Chao1、Pielou和ACE指数值均较高,分别为2.48、62、0.87和47.8,Simpson指数值较低为0.08,表明该位点物种比较丰富。在所有采样点中,从化、良口、人和和绿道–1位点的Shannon、Chao1、Pielou和ACE指数值相对于其他采样点较高,上游2个采样点(水库和温泉)的指数值较低,表明反硝化细菌在流溪河各不同采样位点的分布存在差异,这一差异可能与不同位点底泥理化性质(表1)和还原性物质污染程度有关。
|
|
表 2 流溪河中不同采样位点nirS型反硝化细菌多样性指数 Table 2 Diversity characteristics of nirS-type denitrifiers in sampling sites in the Liuxi River |
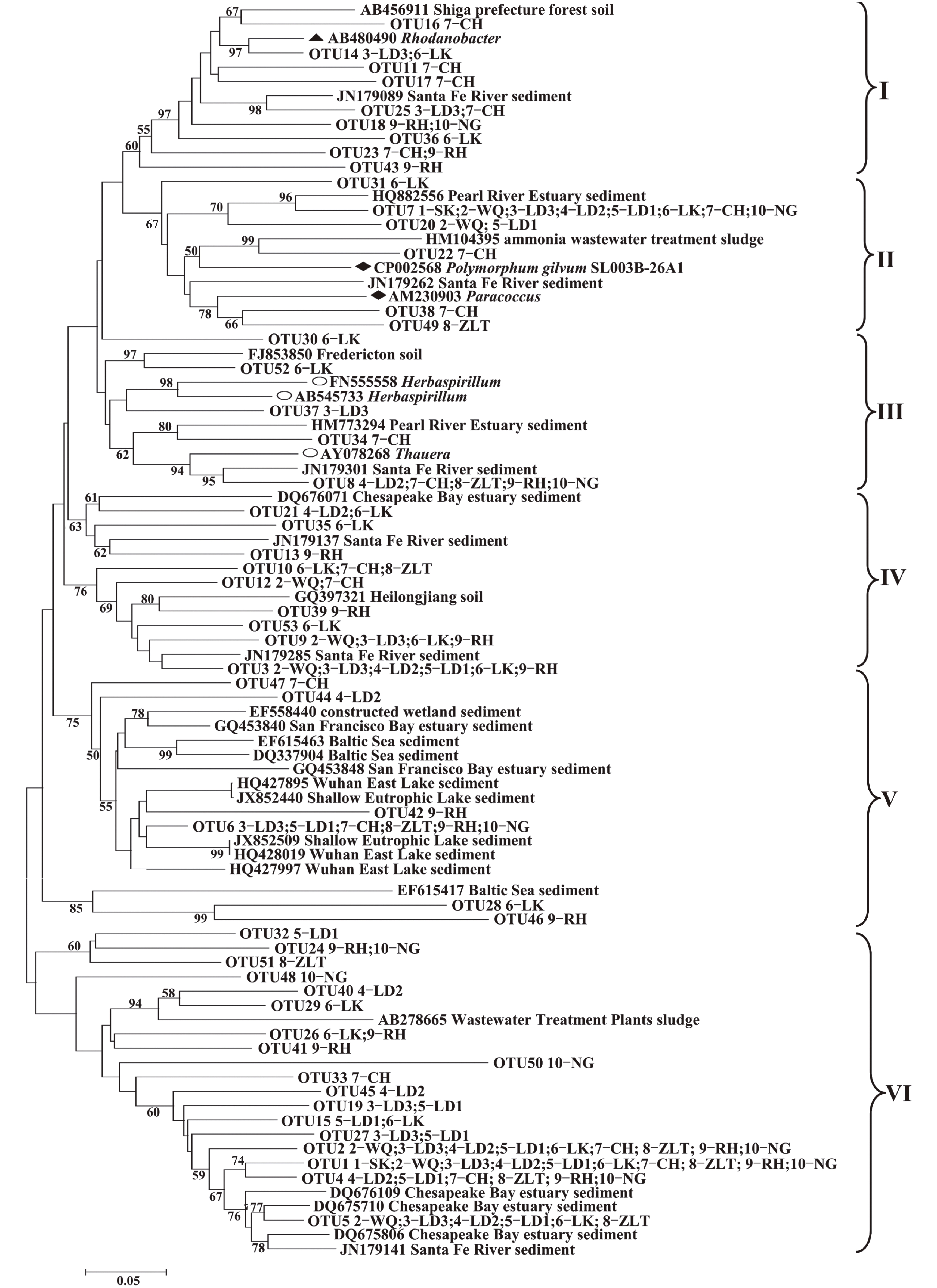
通过与NCBI数据库nirS序列比对,发现流溪河沉积物中大多数nirS序列与已知的反硝化细菌亲缘关系较远,与数据库其他环境样品中的nirS序列有较高相似性(图2)。根据邻位相接法构建系统进化树,将所有序列分成6簇,第1簇与γ–变形菌纲的产黄杆菌属Rhodanobacter有较高相似性;第2簇与α–变形菌纲的副球菌属Paracoccus以及Polymorphum gilvum的菌株SL003B–26A1有较高相似性;第3簇与β–变形菌纲的草螺菌属Herbaspirillum和陶厄氏菌属Thauera有较高相似性。其他簇序列没有亲缘关系较近的已知物种。
|
图 2 基于nirS序列的反硝化细菌系统发育树 Figure 2 Neighbor-joining phylogenetic tree of denitrifying bacteria based on nirS sequences |
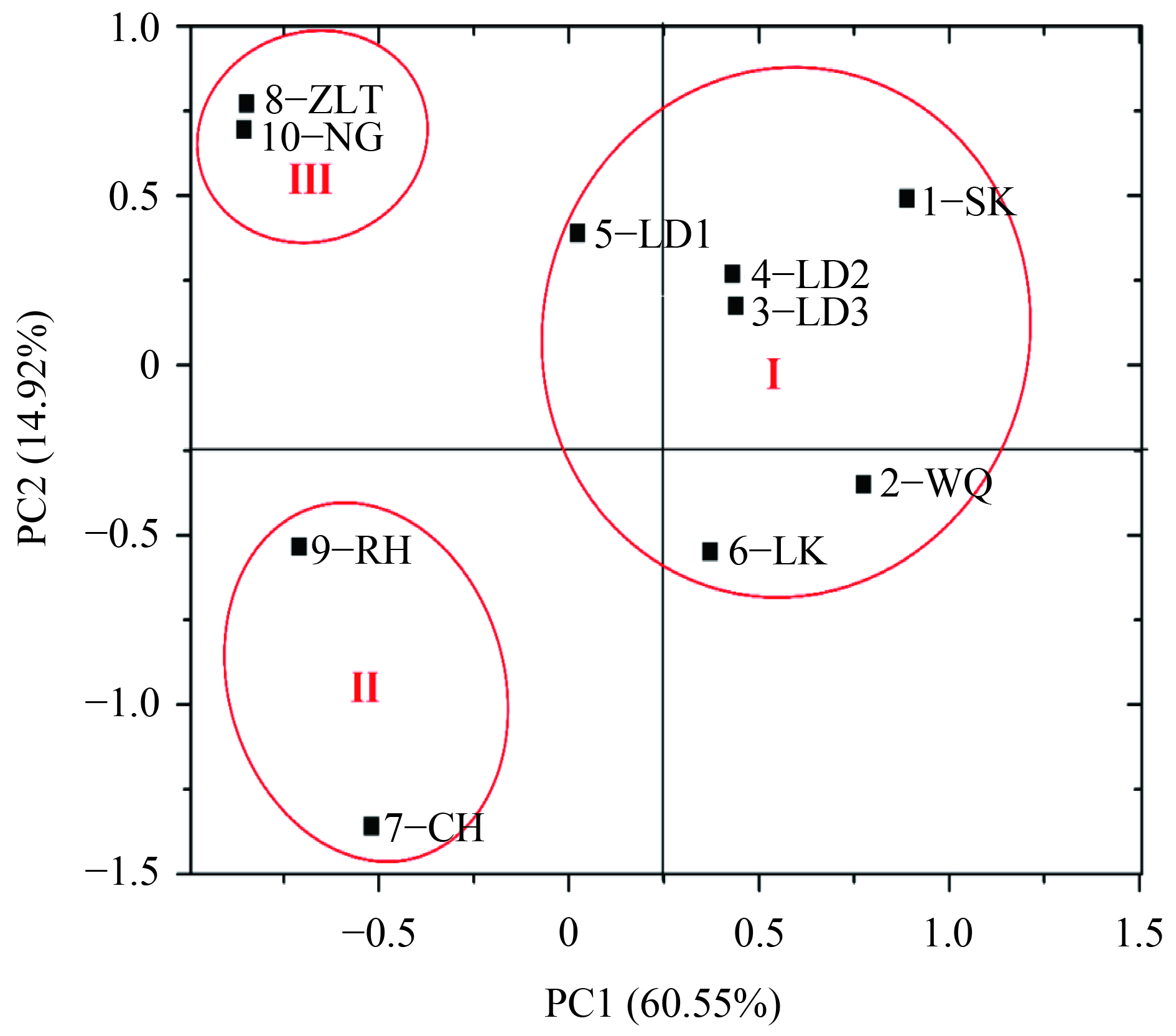
PCoA分析结果如图3所示,PC1和PC2主成分贡献率为75.47%,结合采样位点的周围环境,可以将所有nirS型反硝化细菌群落聚类为3组,即中上游源头区(包括水库、温泉、绿道–3、绿道–2、绿道–1和良口),农业养殖区(从化、人和)和城市生活区(钟落潭、南岗)。这3个区域在地理位置和环境特点等方面的差异影响了反硝化细菌群落的组成及分布。
|
图 3 nirS基因序列的主坐标分析 Figure 3 Principal coordinate analyses of nirS gene sequences |
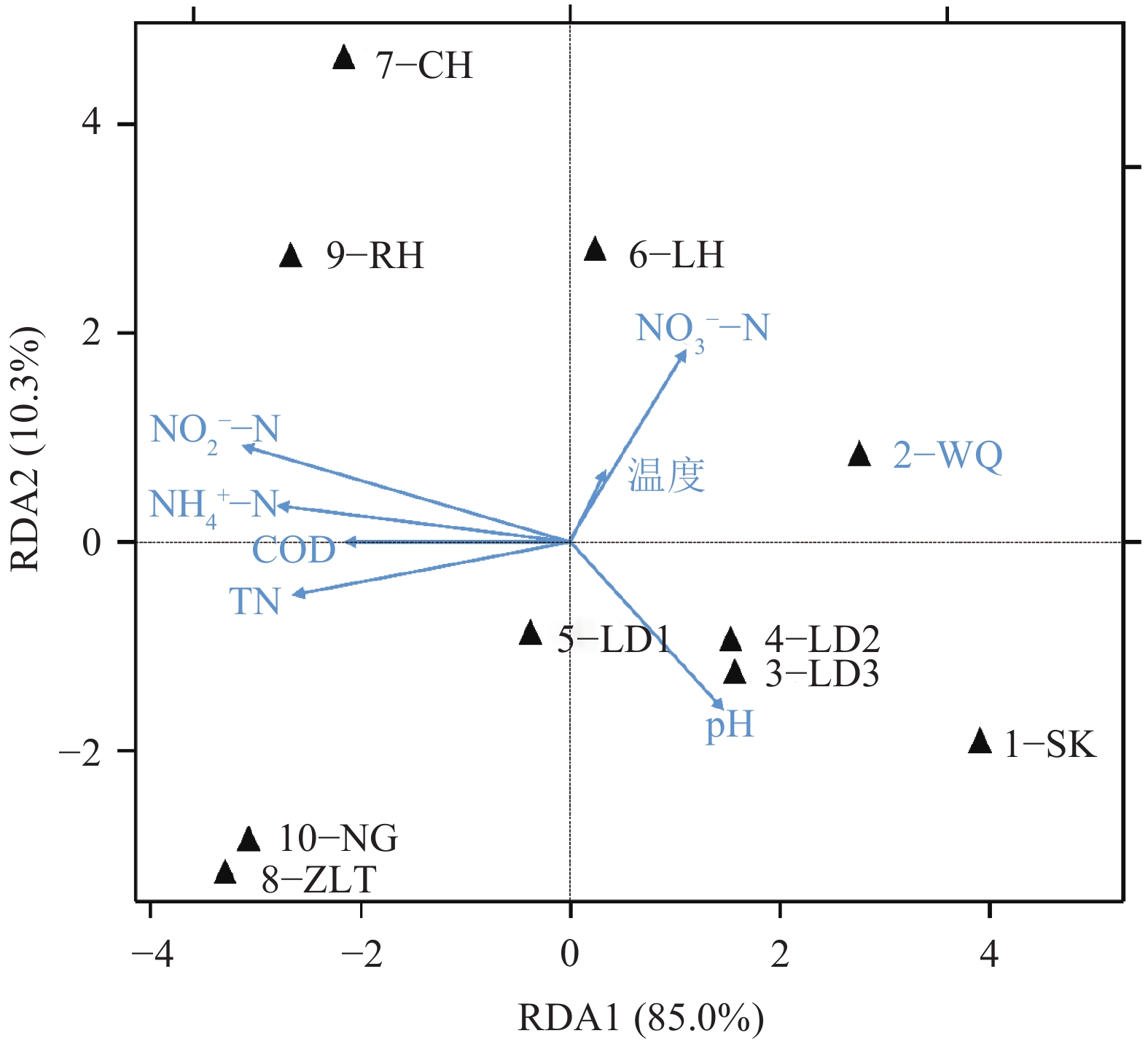
流溪河沉积物中nirS型反硝化细菌群落分布与环境因子相关性的RDA分析如图4所示。坐标轴RDA1和RDA2总解释量为95.3%,高解释量表明了环境因子对反硝化细菌群落分布影响的可靠性。结果表明,nirS型反硝化细菌群与沉积物中NO2––N(P=0.001,F=10.713)和NH4+–N(P=0.049 8,F=3.025)含量显著相关,分别占据了总解释量的57.2%和12.3%;而与TN、COD和NO3––N含量、pH和温度没有相关性(P>0.05, 1 000次蒙特卡洛排列),共占据了总解释量的30.9%。
|
图 4 nirS型反硝化细菌群落结构与环境因子冗余度分析 Figure 4 Redundancy analyses for correlations between nirS-type denitrifier community structure and environmental factors |
如图5所示,流溪河沉积物中nirS基因拷贝数呈明显的空间差异性分布,其中水库的nirS基因拷贝数最低(8.26×101 g–1),南岗最高(5.45×104 g–1),其次为从化(2.96×104 g–1),其他采样位点nirS基因拷贝数范围为7.89×103~1.34×104 g–1。
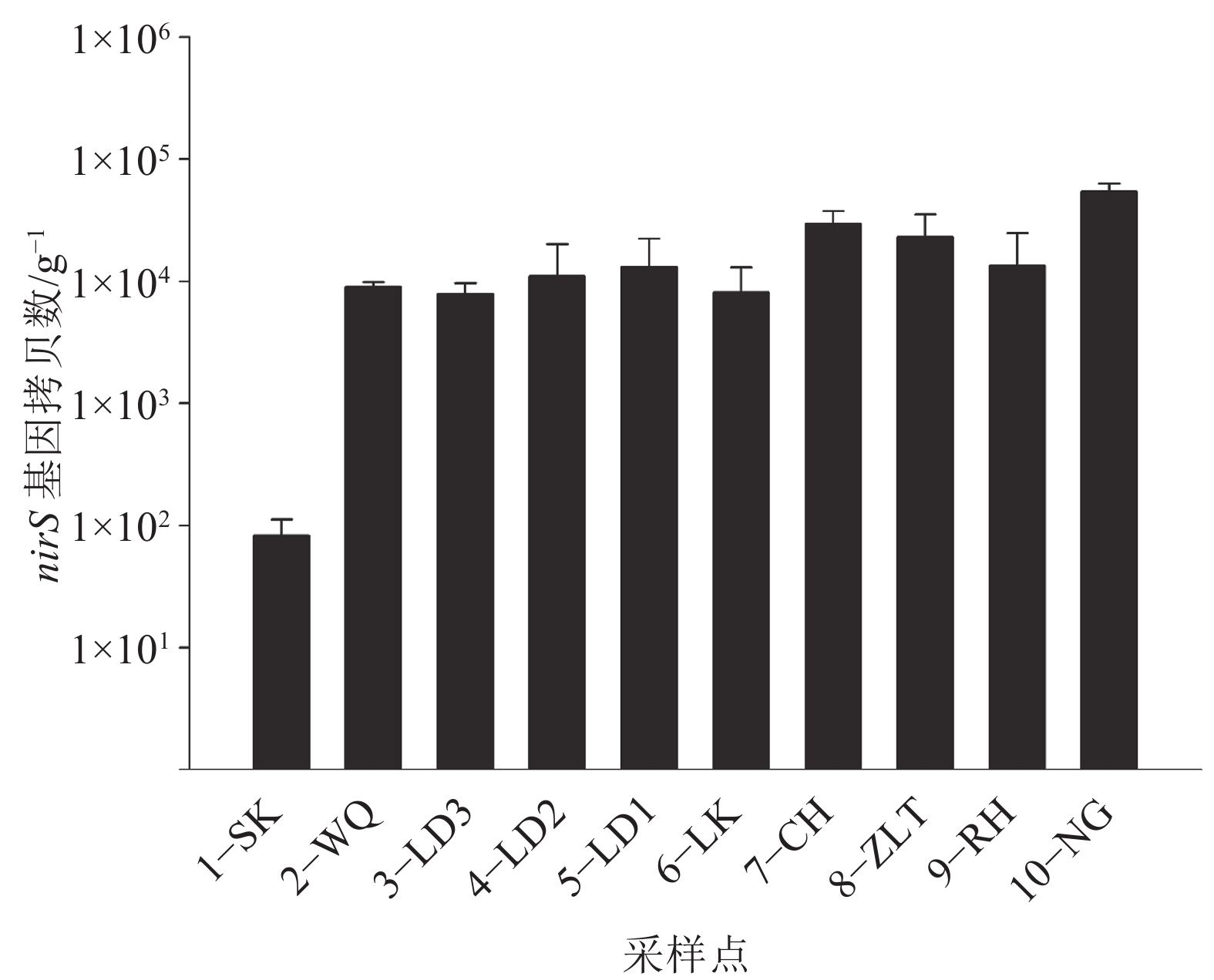
|
图 5 流溪河各采样点沉积物中nirS基因拷贝数 Figure 5 nirS gene copy numbers in sediments of sampling sites in the Liuxi River |
基因丰度和环境因子相关性分析结果如表3所示,nirS基因丰度与NH4+–N、COD和TN含量呈极显著正相关,与NO3––N含量呈显著负相关。
|
|
表 3 nirS基因丰度与环境因子相关系数 Table 3 Correlation analyses of nirS gene abundance with environmental factors |
本研究采用构建基因文库和实时荧光定量的方法研究了广州市流溪河沉积物中反硝化细菌的多样性,分析了沉积物中反硝化细菌的群落结构、基因丰度及其与环境因子的关系。研究发现流溪河中nirS型反硝化细菌主要与变形菌门中的α–,β–和γ–变形菌纲亲缘关系较近,此结果与Henry等[10]报道一致,说明了变形菌门在反硝化过程中的重要作用。同时,本研究中的nirS型反硝化细菌还与许多来自其他生态系统(包括土壤、河口沉积物以及污水处理厂污泥)的微生物亲缘关系较近,表明了流溪河沉积物中反硝化细菌群落的复杂性。Gao等[11] 以及Zheng等[7]分别报道沿海湿地和河流入海口的反硝化细菌nirS序列均与水生系统中的菌群亲缘关系较近,可能原因是海水的高盐度导致其反硝化细菌群落与其他生态系统存在差异。
大量研究表明环境因子会对反硝化细菌群落结构产生影响。Gao等[11]发现NH4+–N含量与反硝化细菌群落多样性呈正相关,与本研究结果一致,这可能是由于NH4+–N含量的升高为反硝化过程提供了更多的电子受体[12]。Francis等[13]发现NO2––N含量显著影响nirS型反硝化细菌群落,并且NO2––N作为氮循环中的重要物质,可以影响nirS基因编码的亚硝酸还原酶的mRNA水平[14]。此外温度、盐度和pH同样会显著影响反硝化细菌群落结构的组成[11, 15-17],但本试验由于采样地点及时间的限制,未发现相关现象。在本试验中,发现nirS型反硝化细菌群落与TN、COD和NO3––N含量存在显著的相关性,这表明流溪河沉积物中反硝化细菌群落对特定环境具有一定的适应性。
流溪河沉积物中nirS基因丰度呈现显著的空间异质性分布(P<0.01)。根据采样点周围环境的不同,我们发现城市生活区的nirS基因丰度要高于源头区和农业养殖区。本研究中nirS基因的拷贝数为8.26×101~5.45×104 g–1,明显低于中国沿海湿地沉积物(4.3×105~3.7×107 g–1)、长江河口沉积物(1.01×106~9.00×107g–1)和旧金山湾沉积物(5×105~5×107 g–1)的拷贝数[11, 18-19]。本研究发现流溪河中NO3––N含量与nirS基因丰度呈显著负相关,Dong等[20]也发现河口中NO3––N含量增加会导致nirS基因丰度的降低。Mosier等[19]发现COD和TN含量对nirS基因丰度有显著影响,与本研究结果一致。此外,Dang等[8]发现NH4+–N含量对nirS细菌群落的组成和丰度具有显著影响。因此,NH4+–N含量是决定反硝化菌数量、组成和分布的关键因素。除此之外,还有一些研究报道pH、温度和NO2––N含量可以改变nirS基因的丰度[8, 11, 14, 21],但本试验没有发现明显的相关性。这些结果意味着进入水生系统中的化学污染物或活性氮(如NO3––N和NH4+–N)会显著影响反硝化细菌群落的基因丰度。
本研究调查了广州市流溪河沉积物中nirS型反硝化细菌的组成、分布以及基因丰度。结果发现nirS型反硝化细菌的组成和基因丰度存在明显的空间差异。总的来说,城市生活区的nirS基因丰度要高于源头区和农业养殖区,这主要是城市环境中TN、NH4+–N、COD和NO3––N含量较高影响的结果。同时,NH4+–N和NO3––N含量也是影响nirS反硝化细菌组成和分布的关键因素。这些研究结果表明河流中化学污染物以及活性氮的大量存在会显著影响水生系统中反硝化细菌群落的生存,从而导致水体富营养化等一系列环境问题。
| [1] |
XIONG W, SUN Y, DING X, et al. Antibiotic resistance genes occurrence and bacterial community composition in the Liuxi River[J]. Front Environ Sci, 2014, 2. DOI:10.3389/fenvs.2014.00061 (  0) 0) |
| [2] |
CAFFREY J M, BANO N, KALANETRA K, et al. Ammonia oxidation and ammonia-oxidizing bacteria and archaea from estuaries with differing histories of hypoxia[J]. ISME J, 2007, 1(7): 660-662. DOI:10.1038/ismej.2007.79 (  0) 0) |
| [3] |
ZUMFT W G. Cell Biology and molecular basis of denitrification[J]. Microbiol Mol Biol Rev, 1997, 61(4): 533-616. (  0) 0) |
| [4] |
PHILIPPOT L, HØJBERG O. Dissimilatory nitrate reductases in bacteria[J]. Biochim Biophys Acta, 1999, 1446(1/2): 1-23. (  0) 0) |
| [5] |
ABELL G C J, REVILL A T, SMITH C, et al. Archaeal ammonia oxidizers and nirS-type denitrifiers dominate sediment nitrifying and denitrifying populations in a subtropical macrotidal estuary
[J]. ISME J, 2010, 4(2): 286-300. DOI:10.1038/ismej.2009.105 (  0) 0) |
| [6] |
NOGALES B, TIMMIS K N, NEDWELL D B, et al. Detection and diversity of expressed denitrification genes in estuarine sediments after reverse transcription-PCR amplification from mRNA[J]. Appl Environ Microbiol, 2002, 68(10): 5017-5025. DOI:10.1128/AEM.68.10.5017-5025.2002 (  0) 0) |
| [7] |
ZHENG Y, HOU L, LIU M, et al. Diversity, abundance, and distribution of nirS-harboring denitrifiers in intertidal sediments of the Yangtze Estuary
[J]. Microb Ecol, 2015, 70(1): 30-40. DOI:10.1007/s00248-015-0567-x (  0) 0) |
| [8] |
DANG H, WANG C, LI J, et al. Diversity and distribution of sediment nirS-encoding bacterial assemblages in response to environmental gradients in the eutrophied Jiaozhou Bay, China
[J]. Microb Ecol, 2009, 58(1): 161-169. DOI:10.1007/s00248-008-9469-5 (  0) 0) |
| [9] |
THROBÄCK I N, ENWALL K, JARVIS Å, et al. Reassessing PCR primers targeting nirS, nirK and nosZ genes for community surveys of denitrifying bacteria with DGGE
[J]. FEMS Microbiol Ecol, 2004, 49(3): 401-417. DOI:10.1016/j.femsec.2004.04.011 (  0) 0) |
| [10] |
HENRY S, BRU D, STRES B, et al. Quantitative detection of the nosZ gene, encoding nitrous oxide reductase, and comparison of the abundances of 16S rRNA, narG, nirK, and nosZ genes in soils
[J]. Appl Environ Microbiol, 2006, 72(8): 5181-5189. DOI:10.1128/AEM.00231-06 (  0) 0) |
| [11] |
GAO J, HOU L, ZHENG Y, et al. nirS-Encoding denitrifier community composition, distribution, and abundance along the coastal wetlands of China
[J]. Appl Microbiol Biotechnol, 2016, 100(19): 8573-8582. DOI:10.1007/s00253-016-7659-5 (  0) 0) |
| [12] |
AVRAHAMI S, CONRAD R, BRAKER G. Effect of soil ammonium concentration on N2O release and on the community structure of ammonia oxidizers and denitrifiers
[J]. Appl Environ Microbiol, 2002, 68(11): 5685-5692. DOI:10.1128/AEM.68.11.5685-5692.2002 (  0) 0) |
| [13] |
FRANCIS C A, O'MULLAN G D, CORNWELL J C, et al. Transitions in nirS-type denitrifier diversity, community composition, and biogeochemical activity along the Chesapeake Bay estuary
[J]. Front Microbiol, 2013, 4(237). DOI:10.3389/fmicb.2013.00237 (  0) 0) |
| [14] |
WANG Y, MA X, ZHOU S, et al. Expression of the nirS, hzsA, and hdh genes in response to nitrite shock and recovery in Candidatus kuenenia stuttgartiensis
[J]. Environ Sci Technol, 2016, 50(13SI): 6940-6947. (  0) 0) |
| [15] |
ROUSK J, BÅÅTH E, BROOKES P C, et al. Soil bacterial and fungal communities across a pH gradient in an arable soil[J]. ISME J, 2010, 4(10): 1340-1351. DOI:10.1038/ismej.2010.58 (  0) 0) |
| [16] |
SZUKICS U, HACKL E, ZECHMEISTER-BOLTENSTERN S, et al. Rapid and dissimilar response of ammonia oxidizing archaea and bacteria to nitrogen and water amendment in two temperate forest soils[J]. Microbiol Res, 2012, 167(2): 103-109. DOI:10.1016/j.micres.2011.04.002 (  0) 0) |
| [17] |
莫旭华, 麻威, 史荣久, 等. 氮肥对小麦田土壤nirS型反硝化细菌多样性的影响
[J]. 微生物学报, 2009, 49(9): 1203-1208. (  0) 0) |
| [18] |
ZHANG Y, XIE X, JIAO N, et al. Diversity and distribution of amoA-type nitrifying and nirS-type denitrifying microbial communities in the Yangtze River estuary
[J]. Biogeosciences, 2014, 11(8): 2131-2145. DOI:10.5194/bg-11-2131-2014 (  0) 0) |
| [19] |
MOSIER A C, FRANCIS C A. Denitrifier abundance and activity across the San Francisco Bay estuary[J]. Environ Microbiol Rep, 2010, 2(5): 667-676. DOI:10.1111/j.1758-2229.2010.00156.x (  0) 0) |
| [20] |
DONG L F, SMITH C J, PAPASPYROU S, et al. Changes in benthic denitrification, nitrate ammonification, and anammox process rates and nitrate and nitrite reductase gene abundances along an estuarine nutrient gradient (the Colne Estuary, United Kingdom)[J]. Appl Environ Microbiol, 2009, 75(10): 3171-3179. DOI:10.1128/AEM.02511-08 (  0) 0) |
| [21] |
胡玲珍, 陈振楼. 河口沉积物反硝化反应影响因子综述[J]. 环境科学动态, 2003(4): 41-43. (  0) 0) |
 2018, Vol. 39
2018, Vol. 39


