2. 安徽农业大学 林学与园林学院,安徽 合肥 230036
2. School of Forestry and Landscape Architecture, Anhui Agricultural University, Hefei 230036, China
随着全球气候变暖,干旱发生更加频繁,持续时间更长,平均气温和极端气温都有所上升[1]。同时,其他非生物胁迫也会导致植物脱水,如冻害、干热风及盐胁迫等。当受到干旱胁迫时,植物会调动多水平的复杂响应来应对胁迫,包括形态适应、生理生化调控及基因表达的调节[1-2]。
梨是世界重要的果树作物,在中国更被称为“百果之宗”。在过去的20年里,中国梨产量增长了3倍以上,从550万t升至1 900万t,占世界梨产量的70%。目前,世界梨产业已偏向于亚洲梨产业[3]。但是,随着干旱趋势的加剧,在我国梨主产区,梨园生产对灌溉水依赖性逐步增大,有的地区已造成地下水严重超采的现象[4],水资源短缺已成为梨果生产的主要限制性因子之一。提高水分利用效率是农业节水栽培的核心问题[5]。目前,梨产区的节水途径主要是改进梨园灌水方式[4],而对梨抗旱资源的挖掘和抗旱品种的培育研究相对较少,特别是通过基因工程培育梨树新品种更是鲜有报道。本研究在前人对干旱胁迫下梨树在生理[6-7]、分子水平响应相关研究的基础上[8-9],以生产实践中发现的抗旱性较强的黄冠梨Pyrus bretschneideri ‘Xuehuali’×P. pyrifolia ‘Shinsseiki’ (Huangguan, HG)和抗旱性较弱的黄金梨P. pyrifolia ‘Niitaka’×P. pyrifolia ‘Nijisseiki’ (Hwangdeum, HK)为试验材料,借助转录组测序技术,挖掘梨树抗旱基因,明析其抗旱代谢相关调控机制,为培育梨抗旱品种提供理论基础和技术支撑。
1 材料与方法 1.1 试验材料2014年7月,以安徽农业大学实验基地所栽种的长势良好的2年生黄金梨和黄冠梨嫁接盆栽苗(杜梨为砧木)为材料,在人工气候室内进行正常浇灌[对照,土壤含水量(w)为70%±5%]和干旱处理[土壤含水量(w)为20%±5%],每个处理(黄冠梨对照:HGC,黄金梨对照:HKC,黄冠梨干旱处理:HGSD和黄金梨干旱处理:HKSD)15株树苗,取梨树中部倒数3~6片成熟功能叶进行总RNA的提取。
1.2 样品处理及转录组数据库分析梨叶片总RNA的提取,采用Li等[10]的方法进行。梨转录组序列的测定和分析参考Li等[11]的方法,获得过滤后的干净读数(Clean reads)后与梨基因组进行比对获得基因序列。在此基础上,对干旱胁迫处理前后2个梨树品种中的差异表达基因(Differentially expressed genes, DEGs)进行分析,本研究中,我们用每百万reads中来自于某基因每千碱基长度的reads数(Reads per kilobase transcriptome per million mapped reads, RPKM)值来统计基因的表达量,且界定错误发现率(False discovery rate, FDR)≤0.01,Log2Ratio的绝对值≥1(Ratio为基因在2个比较样本中表达量的比值)的基因为差异表达基因。对于差异基因的注释和功能分析,则是把基因序列分别在NCBI的非冗余蛋白质序列库(NCBI non-redundant protein sequences, Nr数据库)、基因本体库(Gene ontology, GO)和京都基因与基因组百科全书库(Kyoto encyclopedia of genes and genomes, KEGG)中进行比对。
1.3 差异表达基因RT-qPCR验证从测序结果中随机挑选 12 个DEGs用实时荧光定量PCR(RT-qPCR)法验证转录组测序数据的有效性和准确性。使用软件Primer premier 5.0设计荧光定量PCR引物 (表1),以叶片的cDNA为模板,以梨的GAPDH片段为内参,详细步骤参考董丽丽等[12]的方法,每个样品进行4次重复检测,使用2–△△Ct法求得待测样品相对表达量。
|
|
表 1 梨叶片差异表达基因荧光定量测定引物 Table 1 Primers for qRT-PCR verification of differentially expressed genes in pear leaves |
测序结果(表2)显示,2个梨树品种的对照分别得到了4 787万(HGC)和4 788万(HKC)的过滤后的干净读数,而2个干旱处理分别得到了4 777万(HGSD)和4 778万(HKSD)的过滤后的干净读数,这些数据为进一步分析提供了足够的数据源。4个样品HGC、HKC、HGSD和HKSD的转录组数据与梨基因组的比对率分别为64.52%、65.08%、64.01%和63.59%。在表达基因的数量上,4个样品中除了HGSD不足2.6万以外,其他样品的表达基因都在2.6万以上。同时,在黄冠梨的2个样品(HGC和HGSD)中都预测到了1 900个左右的新转录本,在黄金梨的2个样品(HKC和HKSD)中均预测到了1 700多个新转录本。同时,4个样品都分别得到了4万多个可变剪切和21万以上的SNPs。
|
|
表 2 梨叶片转录组分析结果 Table 2 Transcriptome analysis result of pear leaves |
不同样本间基因差异表达的分析结果如图1所示,不同浇水处理下2个品种间都有差异表达基因;进一步研究表明,正常浇水处理下2个梨品种有4 377个DEGs,干旱处理后2个梨品种有3 841个DEGs,不同浇水处理下都有的DEGs有2 501个,1 876个基因只在正常浇水处理差异表达,而1 340个DEGs仅在干旱后差异表达。进一步分析发现,这1 340个DEGs中有529个基因上调表达,而其余811个基因下调,这些基因是我们进一步研究的重点。
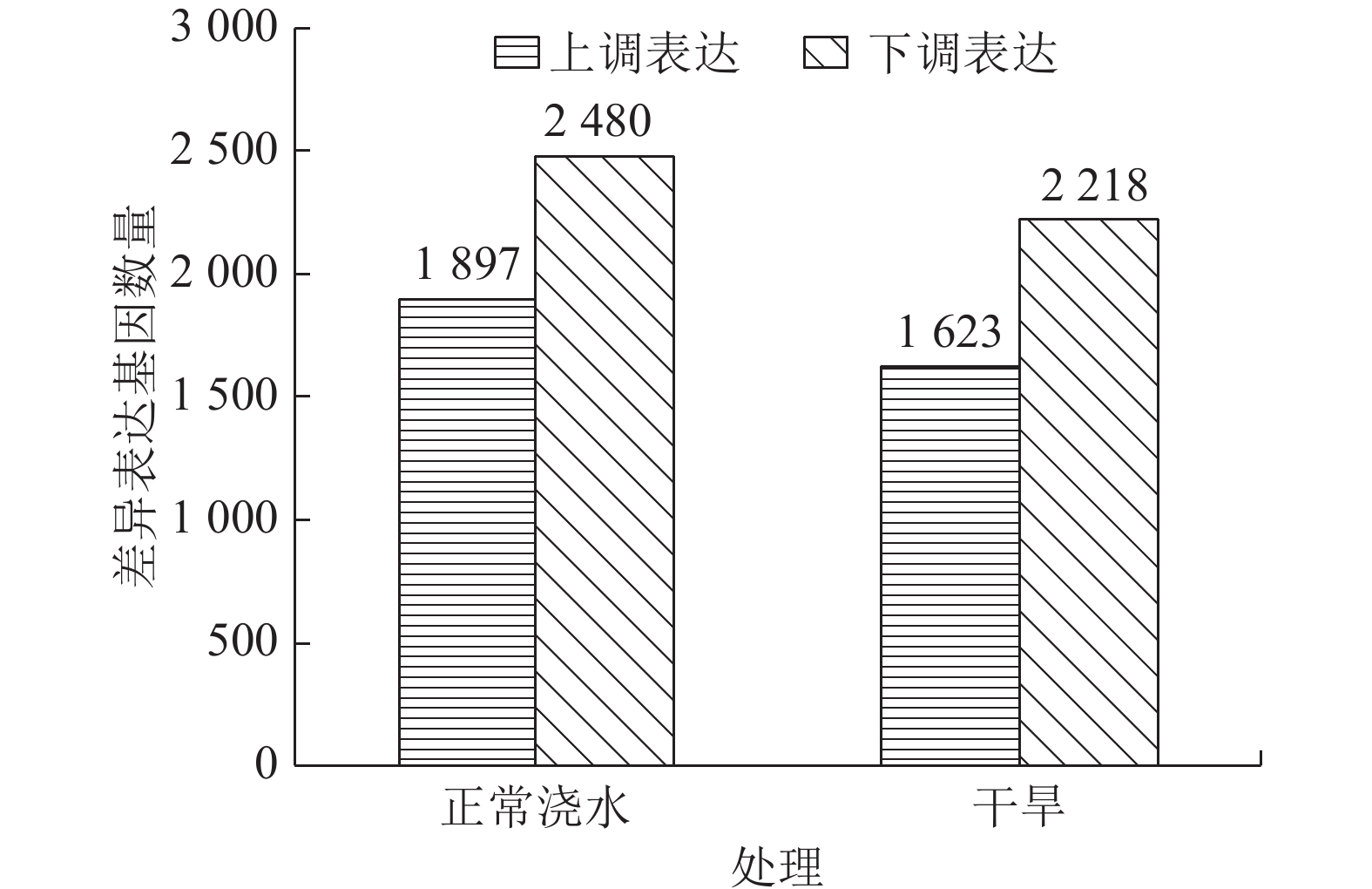
|
图 1 不同处理下2个梨品种中差异表达基因统计 Figure 1 Statistics of differentially expressed genes in two pear genotypes under different treatments |
基因本体(GO)是一个国际标准化的基因功能分类体系,提供了一套动态更新的标准词汇表来全面描述生物体中基因和基因产物的属性。将1 340个仅在干旱处理下的DEGs与GO数据库进行比对(表3),总共有3 544个DEGs至少含有一种GO功能注释(P<0.05),其中注释到生物过程、分子功能和细胞成分3个本体中的DEGs分别有1 387、922和1 253个。其中,生物过程的基因富集在18个GO本体条目,其中代谢过程、细胞过程、单一的生物过程和应激反应所富集的DEGs数量居于前4位,所占比例都在10%以上;分子功能的基因主要富集在11个GO本体条目,其中催化活性和结合所富集的基因最多,共占DEGs总数的60%以上;细胞成分被分为14个GO本体条目,其中细胞、细胞部分、细胞器和生物膜富集的基因较多,占DEGs总数的75%以上。
|
|
表 3 干旱处理下1 340个差异表达基因的基因本体分析 Table 3 Gene ontology analysis of 1 340 differentially expressed genes under drought stress |
|
|
表 4 干旱处理下1 340个差异表达基因的KEGG通路分析 Table 4 Summary of KEGG pathway for 1 340 differentially expressed genes under drought stress |
在1 340个仅在干旱处理下的DEGs中共有239个基因可以比对注释到102类KEGG的代谢通路中,其中,10个代谢通路富集达到差异显著(P<0.05)(表4)。其中,注释到苯丙素的生物合成代谢途径的DEGs最多,为11个。其次,注释到植物内源激素脱落酸(Abscisic acid, ABA)相关的类胡萝卜素生物合成途径的有8个DEGs,与茉莉酸类物质(Jasmonic acid,JA)生物合成相关的α–亚麻酸的代谢途径的有9个DEGs以及与细胞分裂素(Cytokinine,CTK)生物合成相关的玉米素合成途径有3个DEGs。其次,与糖代谢相关的途径有2条,分别是戊糖、葡萄糖醛酸转换(9个DEGs)和半乳糖代谢(8个DEGs)。另外,注释到不饱和脂肪酸生物合成和脂肪酸降解的都有7个DEGs,注释为氮代谢的有3个DEGs。
2.3.3 转录因子分析仅在干旱处理下的2个梨品种的1 340个DEGs通过Blast在Nr库中比对上1 322个,其中有37个被注释为转录因子,这些转录因子分布在17个转录因子家族中。其中注释到最多的是乙烯应答转录因子(Ethylene responsive factor,ERF)家族,有11个DEGs(2个上调、9个下调)。另外,居于前4位的还有C2H2L(2个上调、2个下调),NAC(2个上调、1个下调)和DOF(1个上调、2个下调),其他家族,除了COL,MYB分别拥有上下调基因各1个,WRKY拥有2个下调基因外,其余家族都只注释到1个差异基因,且除了HD外,都下调表达(图2)。
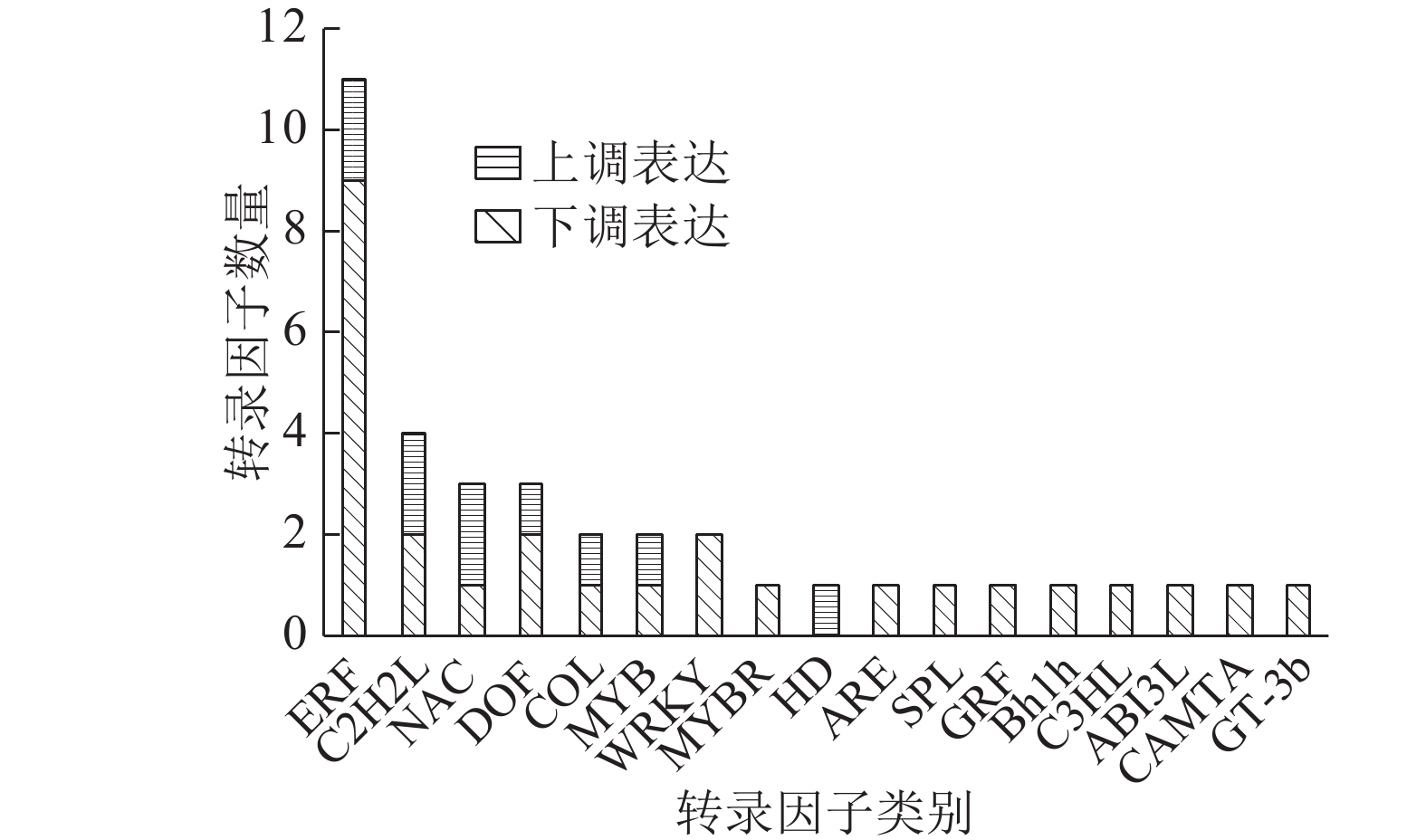
|
图 2 干旱处理下1 340 个差异表达基因中转录因子分类 Figure 2 Classification of transcription factors among 1 340 differentially expressed genes under drought stress |
植物激素与植物抗旱的关系一直是科研的热点,作为“胁迫激素”的ABA更是备受关注。本研究中,把2个品种中干旱后特异表达的DEGs与类胡萝卜素生物合成代谢途径(ko00906)比对发现,与ABA生物合成密切相关的基因有β–胡萝卜素羟化酶(Carotene hydroxylase,CtrZ,Pbr002245.1)、玉米黄质环氧化酶 (Zeaxanthin epoxidase,ZEP,Pbr025145.1)、9–顺环氧类胡萝卜素双加氧化酶(9-cis-epoxycarotenoid dioxygenase,NCED,Pbr002245.1)、ABA2(SDR类基因,编码一个短链脱氢酶,Pbr022971.1)及脱落酸醛氧化酶 (Indole-3- acetaldehyde oxidase, AAO3, Pbr029589.1、Pbr005981.1、Pbr005979.1和Pbr005978.1),这些基因的表达情况见表5。
|
|
表 5 干旱处理下ABA生物合成相关差异表达基因的表达与注释 Table 5 Gene expression and annotation of differentially expressed genes related to ABA biosynthesis under drought stress |
从不同浇水处理下的2个梨品种比较组中分别随机选取2个上调基因和4个下调基因,用qRT-PCR方法验证测序数据的有效性,结果如图3所示。这些结果与转录组分析结果一致,从另一个方面证实了2组数据中差异表达基因的数据有效性。
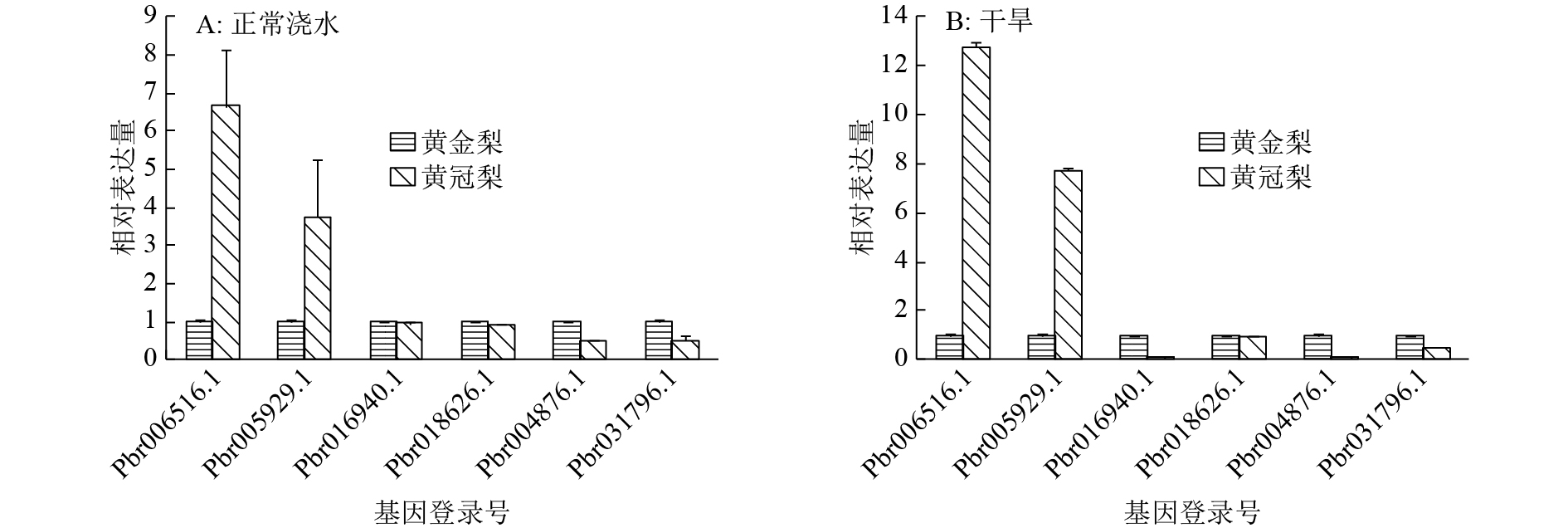
|
图 3 不同处理下2个梨品种差异表达基因qRT-PCR验证 Figure 3 qRT-PCR validation of differentially expressed genes in two pear genotypes under different treatments |
本研究以正常浇水和干旱胁迫下耐旱梨品种黄冠梨和不耐旱梨品种黄金梨的叶片为试验材料,通过转录组分析找到了跟植物抗旱有关的的差异表达基因,其中在富集达到差异显著的10个代谢通路中,与植物内源激素相关的代谢通路有3个,分别是类胡萝卜素生物合成途径(8个DEGs)、α–亚麻酸的代谢途径(9个DEGs)以及玉米素合成途径(3个DEGs)。其中,与ABA生物合成相关的类胡萝卜素生物合成途径的8个差异基因分别被注释为CtrZ、ZEP、NCED和ABA2的各有1个DEGs,注释为AAO3的有4个DEGs。
植物产生的主要激素有生长素(Auxin)、赤霉素(Aibberellin, GA)、细胞分裂素(Cytomin, CTK)、脱落酸(ABA)、乙烯(Ethylene, ETH)、水杨酸(Salicylic acid, SA)、茉莉酸(JA)和油菜素内脂(Brassinosteroid, BR)等,其中,ABA、SA、JA和ETH在介导植物对病原菌和非生物胁迫的防御反应中起着重要作用,而它们中的ABA在植物非生物抗逆中的研究较深入[13]。研究表明[14],高盐和干旱刺激能引起植物激素ABA水平的升高,接着ABA启动信号级联反应,触发诸如关闭气孔等短期反应,从而通过调节胁迫反应基因来维持水分平衡和长期生长反应。
ABA在胁迫发生时的积累是通过ABA生物合成相关基因的表达而实现的。ABA的生物合成的主要途径是C40途径,在这个过程中C40的类胡萝卜素是合成ABA的前体[15]。在合成类胡萝卜的过程中,CtrZ是关键基因之一,能提高类胡萝卜素的产量[16]。在ABA合成的过程中,ZEP促进玉米黄质形成花药黄质并进一步生成紫黄质[17],这一步是ABA的合成关键步骤之一。NCED则是9–顺式紫黄质或9′–顺式新黄质裂解为C15化合物黄质醛的限速酶[18],该步骤是ABA合成的另一个关键步骤。以上反应在质体中进行,此后,黄质醛被从质体输出到了细胞溶质内,并在SDR/ABA2催化作用下转变为ABA醛[19],ABA醛则在AAO的作用下生成ABA[20]。本研究中,在干旱胁迫下,ABA合成的8个关键基因在干旱处理下的黄冠梨和黄金梨中差异表达,这有可能是2个梨品种抗旱性差异的重要原因之一,因此这些基因的表达将协同ABA的动态变化,是下一步梨抗旱分子机理研究的重点。
转录因子在作物抗旱中的作用已屡被报道[21]。本研究中注释到的转录因子家族有17个,其中ERF家族基因数量最多。ERF转录因子家族属于AP2转录因子超家族的亚家族之一,含有一个AP2-ERF功能结构域,可特异性的结合GCC-box来调控下游胁迫响应基因表达,近年来成为植物抗逆研究的热点[22]。在水分胁迫研究中,拟南芥中AtERF71/HRE2基因被证明能调节渗透胁迫[23],芝麻[24]中的ERF基因被证明可以被干旱诱导,番茄在旱、寒胁迫或ABA刺激下,TERF1表达显著上升,并参与渗透调节[25]。ERF蛋白中含有乙烯响应元件绑定蛋白,会响应乙烯诱导[26],但也有研究表明SA[27]、JA[28]以及ABA[25]也会诱导ERF基因表达。同时,人们对大麦研究表明,转录因子HvDRF1(AP2/ERF类转录因子)与HvABI5(bZIP类转录因子)互作,提高了下游基因HVA1的表达[29]。由此可见,植物抗逆响应是由多基因参与、各途径彼此联系的复杂的网络调控过程,ERF类转录因子可能处在网络的某个交叉点,协同调控下游基因的表达从而参与植物的抗逆[30]。本研究中的11个ERF转录因子,干旱胁迫后在2个品种间有2个上调表达、9个下调表达,它们可能会通过参与内源激素信号转导或与其他转录因子互作而响应梨的水分胁迫。
| [1] |
GIORGI F, LIONELLO P. Climate change projections for the Mediterranean region[J]. Global Planet Change, 2008, 63(2/3): 90-104. (  0) 0) |
| [2] |
WU J, WANG L F, LI L, WANG S. De novo assembly of the common bean transcriptome using short reads for the discovery of drought-responsive genes[J]. PLoS One, 2014, 9(10): e109262. DOI:10.1371/journal.pone.0109262 (  0) 0) |
| [3] |
HWANG I G, KIM H Y, WOO K S, et al. Isolation and identification of the antioxidant DDMP from heated pear (Pyrus pyrifolia Nakai)
[J]. Prev Nutr Food Sci, 2013, 18(1): 76-79. DOI:10.3746/pnf.2013.18.1.076 (  0) 0) |
| [4] |
ZHANG Y C, SHEN Y J, XU X L, et al. Characteristic of the water-energy-carbon fluxes of irrigated pear orchards in the North China Plain[J]. Agr Water Manage, 2013, 128(1): 140-148. (  0) 0) |
| [5] |
山仑. 植物水分利用效率和半干旱地区农业用水[J]. 植物生理学通讯, 1994, 30(1): 61-66. (  0) 0) |
| [6] |
STRUTHERS R, IVANOVA A, TITS L, et al. Thermal infrared imaging of the temporal variability in stomatal conductance for fruit trees[J]. Intl J Appl Earth Obs Geoinf, 2015, 39: 9-17. DOI:10.1016/j.jag.2015.02.006 (  0) 0) |
| [7] |
ZARAFSHAR M, AKBARINIA M, ASKARI H, et al. Morphological, physiological and biochemical responses to soil water deficit in seedlings of three populations of wild pear tree (Pyrus boisseriana)
[J]. Biotechnol Agron Soc Environ, 2014, 18(3): 31-35. (  0) 0) |
| [8] |
HUSSAIN S, NIU Q F, QIAN M J, et al. Genome-wide identification, characterization, and expression analysis of the dehydrin gene family in Asian pear (Pyrus pyrifolia)
[J]. Tree Genet Genomes, 2015, 11(5): 110. DOI:10.1007/s11295-015-0938-y (  0) 0) |
| [9] |
LI K Q, XU X Y, HUANG X S. Identification of differentially expressed genes related to dehydration resistance in a highly drought-tolerant pear, Pyrus betulaefolia, as through RNA-Seq
[J]. PLoS One, 2016, 11(2): e0149352. DOI:10.1371/journal.pone.0149352 (  0) 0) |
| [10] |
LI T, LI X Y, TAN D M, et al. Distinct expression profiles of ripening related genes in the ‘Nanguo’ pear (Pyrus ussuriensis) fruits
[J]. Sci Hortic, 2014, 171: 78-82. DOI:10.1016/j.scienta.2014.03.054 (  0) 0) |
| [11] |
LI R Q, YU C, LI Y R, et al. SOAP2: an improved ultrafast tool for short read alignment[J]. Bioinformatics, 2009, 25(15): 1966-1967. DOI:10.1093/bioinformatics/btp336 (  0) 0) |
| [12] |
董丽丽, 龚凌燕, 陈磊, 等. 石榴肉桂酰辅酶A还原酶基因的克隆与表达分析[J]. 南京农业大学学报, 2016, 39(5): 747-753. DOI:10.7685/jnau.201602011 (  0) 0) |
| [13] |
NAKASHIMA K, YAMAGUCHI-SHINOZAKI K. ABA signaling in stress-response and seed development[J]. Plant Cell Rep, 2013, 32(7): 959-970. DOI:10.1007/s00299-013-1418-1 (  0) 0) |
| [14] |
MEHROTRA R, BHALOTHIA P, BANSAL P. Abscisic acid and abiotic stress tolerance - different tiers of regulation[J]. J Plant Physiol, 2014, 171(7): 486-496. DOI:10.1016/j.jplph.2013.12.007 (  0) 0) |
| [15] |
ROCK C D, ZEEVAART J A. The ABA mutant of Arabidopsis thaliana is impaired in epoxy-carotenoid biosynthesis
[J]. Proc Natl Acad Sci USA, 1991, 88(17): 7496-7499. DOI:10.1073/pnas.88.17.7496 (  0) 0) |
| [16] |
MISAWA N, SATOMI Y, KONDO K, et al. Structure and functional analysis of a marine bacterial carotenoid biosynthesis gene cluster and astaxanthin biosynthetic pathway proposed at the gene level[J]. J Bacteriol, 1995, 177(22): 6575-6584. DOI:10.1128/jb.177.22.6575-6584.1995 (  0) 0) |
| [17] |
AUDRAN C, GONNEAU M, NORTH H, et al. Localisation and expression of zeaxanthin epoxidase mRNA in Arabidopsis in response to drought stress and during seed development
[J]. Aust J Plant Physiol, 2001, 28(11): 1161-1173. (  0) 0) |
| [18] |
QIN X, ZEEVAART J A. Overexpression of a 9-cis-epoxycarotenoid dioxygenase gene in Nicotiana plumbaginifolia increases abscisic acid and phaseic acid levels and enhances drought tolerance
[J]. Plant Physiol, 2002, 128(2): 544-551. DOI:10.1104/pp.010663 (  0) 0) |
| [19] |
GONZÁLEZ-GUZMÁN M, APOSTOLOVA N, BELLlÉS J M, et al. The short-chain alcohol dehydrogenase ABA2 catalyzes the conversion of xanthoxin to abscisic aldehyde[J]. Plant Cell, 2002, 14(8): 1833-1846. DOI:10.1105/tpc.002477 (  0) 0) |
| [20] |
SEO M, AOKI H, KOIWAI H, et al. Comparative studies on the Arabidopsis aldehyde oxidase(AAO) gene family revealed a major role of AAO3 in ABA biosynthesis in seeds[J]. Cell Physiol, 2004, 45(11): 1694-703. DOI:10.1093/pcp/pch198 (  0) 0) |
| [21] |
吴蓓, 李梦瑶, 王广龙, 等. AgERF4 转录因子基因的克隆与表达分析
[J]. 园艺学报, 2016, 43(11): 2193-2202. (  0) 0) |
| [22] |
吴凡, 张超, 郭加, 等. 牡丹切花ERF转录因子基因的分离与表达分析
[J]. 园艺学报, 2016, 43(1): 109-120. (  0) 0) |
| [23] |
PARK H Y, SEOK H Y, WOO D H, et al. AtERF71/HRE2 transcription factor mediates osmotic stress response as well as hypoxia response in Arabidopsis
[J]. Biochem Biophys Res Commun, 2011, 414(1): 135-141. DOI:10.1016/j.bbrc.2011.09.039 (  0) 0) |
| [24] |
DOSSA K, WEI X, LI D, et al. Insight into the AP2/ERF transcription factor superfamily in sesame and expression profiling of DREB subfamily under drought stress[J]. BMC Plant Biol, 2016, 16(1): 171. DOI:10.1186/s12870-016-0859-4 (  0) 0) |
| [25] |
ZHANG X, ZHANG Z, CHEN J, et al. Expressing TERF1 in tobacco enhances drought tolerance and abscisic acid sensitivity during seedling development[J]. Planta, 2005, 222(3): 494-501. DOI:10.1007/s00425-005-1564-y (  0) 0) |
| [26] |
ZHAO Y, WEI T, YIN K Q, et al. Arabidopsis RAP2.2 plays an important role in plant resistance to Botrytis cinerea and ethylene responses
[J]. New Phytol, 2012, 195(2): 450-460. DOI:10.1111/j.1469-8137.2012.04160.x (  0) 0) |
| [27] |
ZHANG G, CHEN M, CHEN X, et al. Phylogeny, gene structures, and expression patterns of the ERF gene family in soybean (Glycine max L.)
[J]. J Exp Bot, 2008, 59(15): 4095-4107. DOI:10.1093/jxb/ern248 (  0) 0) |
| [28] |
CHAKRAVARTHY S, TUORI R P, D’ASCENZO M D, et al. The tomato transcription factor Pti4 regulates defense-related gene expression via GCC box and non-GCC box elements
[J]. Plant Cell, 2003, 15(12): 3033-3050. DOI:10.1105/tpc.017574 (  0) 0) |
| [29] |
XUE G P, LOVERIDGE C W. HvDRF1 is involved in abscisic acid-mediated gene regulation in barley and produces two forms of AP2 transcriptional activators, interacting preferably with a CT-rich element[J]. Plant J, 2004, 37(3): 326-339. DOI:10.1046/j.1365-313X.2003.01963.x (  0) 0) |
| [30] |
LICAUSI F, OHME-TAKAGI M, PERATA P. APETALA2/Ethylene responsive factor (AP2/ERF) transcription factors: Mediators of stress responses and developmental programs[J]. New Phytol, 2013, 199(3): 639-649. DOI:10.1111/nph.12291 (  0) 0) |
 2018, Vol. 39
2018, Vol. 39


