2. 温氏研究院,广东 云浮 527400
2. Wens’ Institute, Yunfu 527400, China
猪流行性腹泻病毒(Porcine epidemic diarrhea virus,PEDV)是α冠状病毒属的成员,是一种具有高感染性的单股正义链RNA病毒。其可以导致猪发生急性水样腹泻、呕吐、脱水等临床症状,尤其对新生仔猪具有高致死率[1]。1978-2018年近40年的时间内,PEDV严重危害了全球养殖业,仅在2010年冬就造成了我国南方近100万头仔猪的死亡,给我国猪业造成了不可估量的损失[2]。到2013年,美国17个州爆发PEDV,近10%的仔猪死亡,随即病毒在接下来的几年传播到欧洲国家,PEDV迅速地遍及全世界[3-4]。对于PEDV防控和治疗,现有研究主要以接种疫苗的方式防控,但是由于疫苗的有效性、安全性和毒株的变异性等问题[5-6],仍然不能使仔猪完全避免PEDV的影响。开发有效的PEDV防控手段对养猪业良性发展具有重要意义。
基因编辑是利用基因编辑工具对生物体基因组进行修改,以产生相应表型的一种精确基因修饰手段。其中簇状规律性短回文重复序列结合蛋白(Clustered regularly interspaced palindromic repeats/CRISPR-associated proteins,CRISPR/Cas)系统是一种在细菌和古细菌里发现的天然适应性免疫系统,由CRISPR序列(间隔区)和侧翼的Cas基因组成。基于参与切割的Cas蛋白将该系统分为2类:一类是多个亚单位蛋白复合发挥作用的I类系统,包含type I,type III和type IV蛋白;另一类主要是以单一蛋白发挥功能的type II(Cas9),type V(Cpf1、C2c1、C2c3)和type VI(Cas13a、Cas13b、Cas13c、Cas13d)型CRISPR系统[7-8]。近年研究发现,CRISPR/Cas系统是唯一可以靶向RNA切割的免疫系统,Cas13蛋白含有2个高等真核生物和原核生物核苷酸结合域,可以介导对RNA的精准靶向切割[9]。该系统通过CRISPR阵列转录成成熟的CRISPR RNA (crRNA),与Cas13蛋白形成Cas13-crRNA复合物靶向切割目标RNA序列。迄今为止Cas13蛋白家族中,Cas13a和Cas13b是已证明可以靶向切割RNA的有力工具[10-11]。Cox等[10]的结果显示Cas13b拥有比Cas13a更高效的RNA抑制效率。Rashid 等[12]利用Cas13a系统成功干扰芜菁花叶病毒(Turnip mosaic virus,TuMV)的感染,首次证实了该系统对植物RNA病毒的抑制能力。基于这些对CRISPR/Cas13系统的研究,本研究以 CRISPR/Cas13b 系统为基础,根据PEDV基因组的保守区域设计多个 CRISPR/Cas13b 系统打靶crRNA,旨在筛选出有效的crRNA以实现对PEDV体外增殖的有效抑制。本研究为后续开发有效的PEDV治疗手段,建立PEDV抗性动物提供了一种新的候选研究手段。
1 材料与方法 1.1 材料质粒:CRISPR/Cas13b基因编辑载体pC0043-PspCas13b crRNA backbone (Addgene编号:103854)、pC0046-EF1a-PspCas13b-NES-HIV(Addgene编号:103862)购于Addgene公司,无PEDV污染的Vero细胞由温氏研究院保存;PEDV ST2毒株由温氏研究院分离保存。
试剂:质粒小提试剂盒(Biomiga,广州),胶回收试剂盒(OMEGA,广州),病毒RNA抽提试剂盒(Magen,上海),Premix Ex Taq DNA聚合酶、SYBR Green Ex Taq酶、Primer Script RT Master Mix(Takara Biotechnology,大连),T4连接酶(New England Biolabs,美国),BbsI内切酶(Thermo Fisher,美国),感受态细胞TOP10(唯地生物,上海),Lipofetamine 3000转染试剂、Alexa Fluor 594标记羊抗兔二抗(Invitrogen,美国),PEDV单克隆抗体(JENO,韩国),无EDTA胰酶、Opti-MEM、DMEM、FBS等细胞培养试剂(Gibco,美国)。
1.2 试验方法 1.2.1 crRNA的设计与合成通过查找近年国内流行PEDV的序列,进行多重比对,寻找其保守序列,设计出多条基于Cas13b打靶系统的crRNA。crRNA长度28 nt,无特定PAM位点需求。将所设计的crRNA序列及其互补链序列分别加上与BbsI酶切位点相匹配的黏性末端,合成相应的寡聚核苷酸。
1.2.2 CRISPR/Cas13b系统打靶质粒的制备用BbsI酶切crRNA骨架质粒。酶切体系为:BbsI酶2 μL,10×buffer 2 μL,质粒(717 ng/μL)3 μL,无菌水补至20 μL,37 ℃条件下反应2 h。酶切产物经电泳鉴定酶切完全后,切胶回收线性化载体片段用于下游连接。
合成的crRNA寡聚核苷酸经过退火复性形成双链DNA。退火体系和程序如下:互补寡聚核苷酸(10 nmol/L)各取5 μL混合,95 ℃ 1 min,10 ℃ 3 min,3个循环;95 ℃ 3 min。退火后形成的双链DNA与BbsI酶切后线性化的骨架质粒以T4连接酶进行连接,反应体系如下:T4连接酶1 μL,线性化载体1 μL,退火双链2 μL,补充无菌水至10 μL,16 ℃条件下连接过夜。连接后的质粒转化感受态细胞Top10,挑取单菌落扩大培养后进行鉴定。由于crRNA片段很短,无法通过酶切进行鉴定,因此我们采用合成的crRNA作为一侧引物,并在载体上设计一段序列作为另一侧引物,采用二者进行PCR扩增时,阳性质粒可以扩增出片段。我们通过菌液PCR鉴定质粒是否构建正确,菌液PCR鉴定引物如表1,PCR反应体系如下:菌液1 μL,上下游引物(10 nmol/L)各0.6 μL,2×Ex Taq酶10 μL,补充无菌水至20 μL,引物序列如表1所示。PCR反应程序:94 ℃ 5 min;94 ℃ 20 s,58 ℃ 20 s,72 ℃ 20 s,30个循环;72 ℃,5 min,4 ℃保存,菌液PCR鉴定为阳性的菌落进行序列测定以确定质粒序列是否正确。阳性质粒分别命名为U6-crRNA1、U6-crRNA2、U6-crRNA3和U6-crRNA4。大量抽提质粒用于后续转染试验。
|
|
表 1 用于PCR鉴定crRNA插入的引物序列 Table 1 Primer sequences for identifying positive crRNA insertion in PCR |
PEDV在Vero细胞上以含7%(φ)胰酶的无血清DMEM培养,至细胞完全病变后收取全部上清和细胞,反复冻融3次以促进病毒释放,再分装后冻存于−80 ℃备用。测定病毒滴度时,将长势良好的Vero细胞接种于96孔板,加入100 μL以含7%(φ)胰酶的无血清DMEM培养基进行10倍梯度稀释的PEDV病毒液,病毒液稀释梯度为10−1~10−7,每个稀释度接种5孔,观察一周的病变情况,应用Reed-Muench法计算病毒的TCID50。
1.2.4 CRISPR/Cas13b转染Vero细胞在转染前1天消化铺板。将长势良好的细胞消化后均匀铺至24孔板,用含 10%(φ)FBS的 DMEM 培养基在37 ℃、CO2体积分数为5%的培养箱中培养,待细胞汇合度达到70%~90%时,弃掉原有培养皿中的培养液,每孔加入300 μL新鲜培养液以备转染。按照Lipofectamine 3000转染试剂说明书以如下步骤转染:将1.5 μL Lipofectamine 3000用25 μL Opti-MEM稀释,另取25 μL Opti-MEM稀释转染质粒(每孔500 ng)和P3000(每孔1 μL),将二者混合后室温下静置10~15 min,混合物加入含300 μL培养基的24孔板中,混匀后静置培养。其中构建好的打靶质粒U6-crRNA和Cas13b质粒共转染Vero细胞为试验组,对照组则转入空白crRNA和Cas13b质粒,每个转染组重复3孔。
1.2.5 病毒感染转染48 h后,弃掉含脂质体培养液并用PBS缓冲液清洗转染细胞3遍,以每孔400 μL含有感染复数(Multiplicity of infection,MOI)=0.001的PEDV病毒液接种细胞,置于37 ℃培养箱中感染1 h,然后弃去病毒液,以PBS缓冲液洗涤细胞3遍,加入500 μL含胰酶的DMEM培养基继续培养。
1.2.6 免疫荧光试验病毒感染细胞24 h后,以免疫荧光(Immunofluorescence assay,IFA)检测病毒的增殖情况。用体积分数为4%的多聚甲醛溶液固定细胞10 min,PBS缓冲液清洗3次,每次5 min。用体积分数为0.3%的Triton溶液透化细胞10 min,PBS缓冲液清洗3次,每次5 min。以3 g/mL BSA溶液室温封闭2 h,然后加入PEDV抗体于4 ℃条件下孵育过夜。次日弃去抗体液,用PBS缓冲液清洗3次,每次5 min,而后加入以PBS缓冲液按1∶500的体积比稀释的Alexa Fluor 594标记二抗,于室温孵育2 h,PBS缓冲液漂洗后于荧光显微镜下观察病毒的感染情况。
1.2.7 荧光定量PCR检测细胞上清病毒含量病毒感染24 h的细胞上清按照Magen病毒RNA抽提试剂盒抽提病毒RNA,抽提相同体积上清液的病毒以保证后续定量操作的一致性。以反转录试剂盒反转录成cDNA,反转录程序为:37 ℃15 min,85 ℃5 s。cDNA用无菌水稀释10倍后用于相对荧光定量PCR,根据Miller等[13]的报道设计PEDV检测用PCR引物:
PEDV-Forward:GAATTCCCAAGGGCGAAAAT;
PEDV-Reverse:TTTTCGACAAATTCCGCATCT。
定量PCR反应体系为:SYBR Green Ex Taq酶 10 μL,上下游引物(10 nmol/L)各0.8 μL,ROX 0.4 μL,cDNA 2 μL,无菌水补充至20 μL。定量PCR程序为:95 ℃30 s;95 ℃15 s,60 ℃1 min,40个循环。同时设置无菌水为阴性对照。定量结果用2−△△Ct法计算相对病毒RNA含量。
1.2.8 数据分析试验数据利用“SPSS for Windows 21.0”统计软件进行了数据差异性检验,采用Duncan’ s法进行组间的多重比较分析。
2 结果与分析 2.1 U6-crRNA载体构建根据病毒基因组设计的crRNA序列和位点见表2,其中靶点1和2靶向PEDV polI基因,靶点3和4靶向S基因。
|
|
表 2 CRISPR/Cas13b靶向PEDV病毒RNA的crRNA序列 Table 2 The crRNA sequences of CRISPR/Cas13b targeting PEDV RNA |
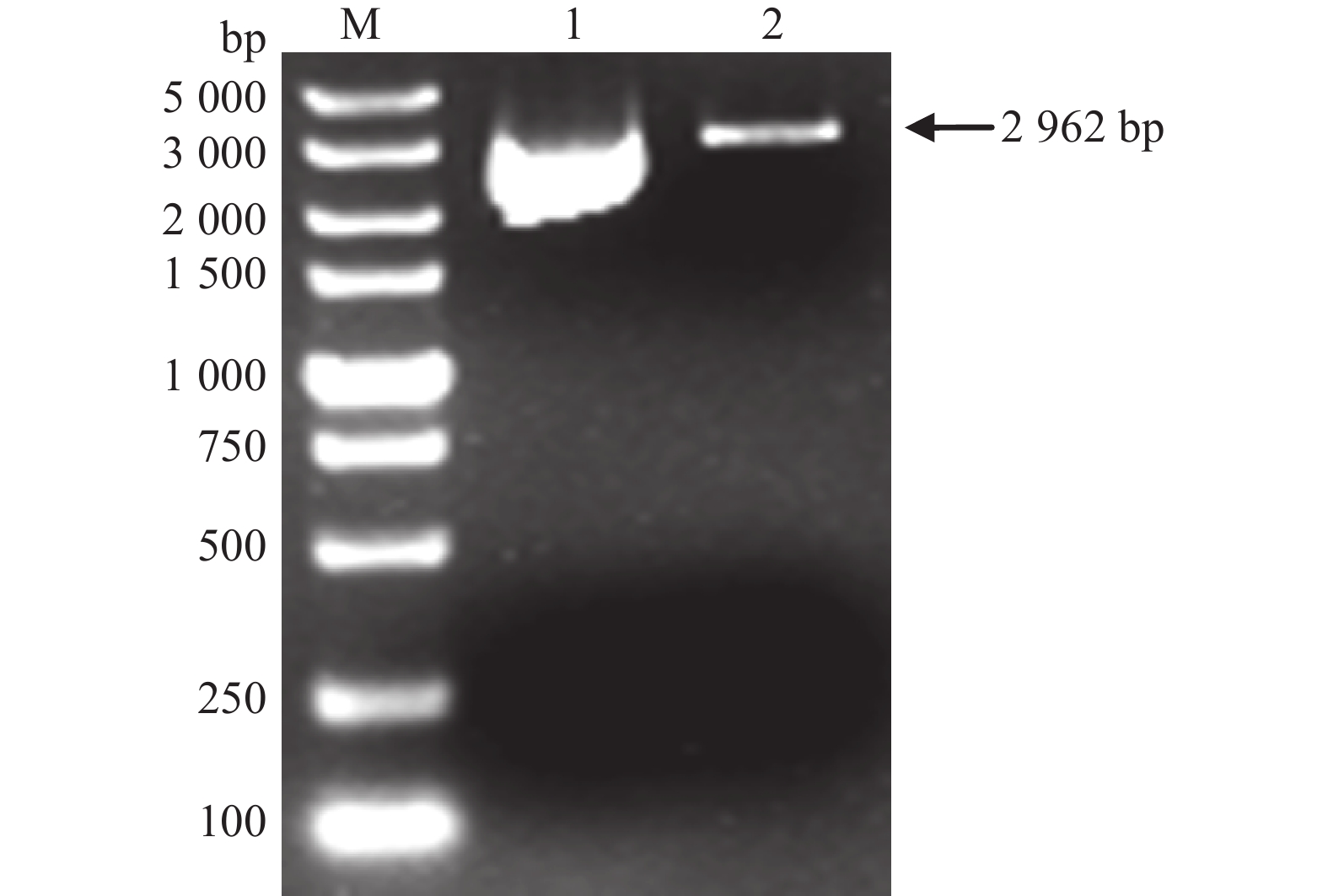
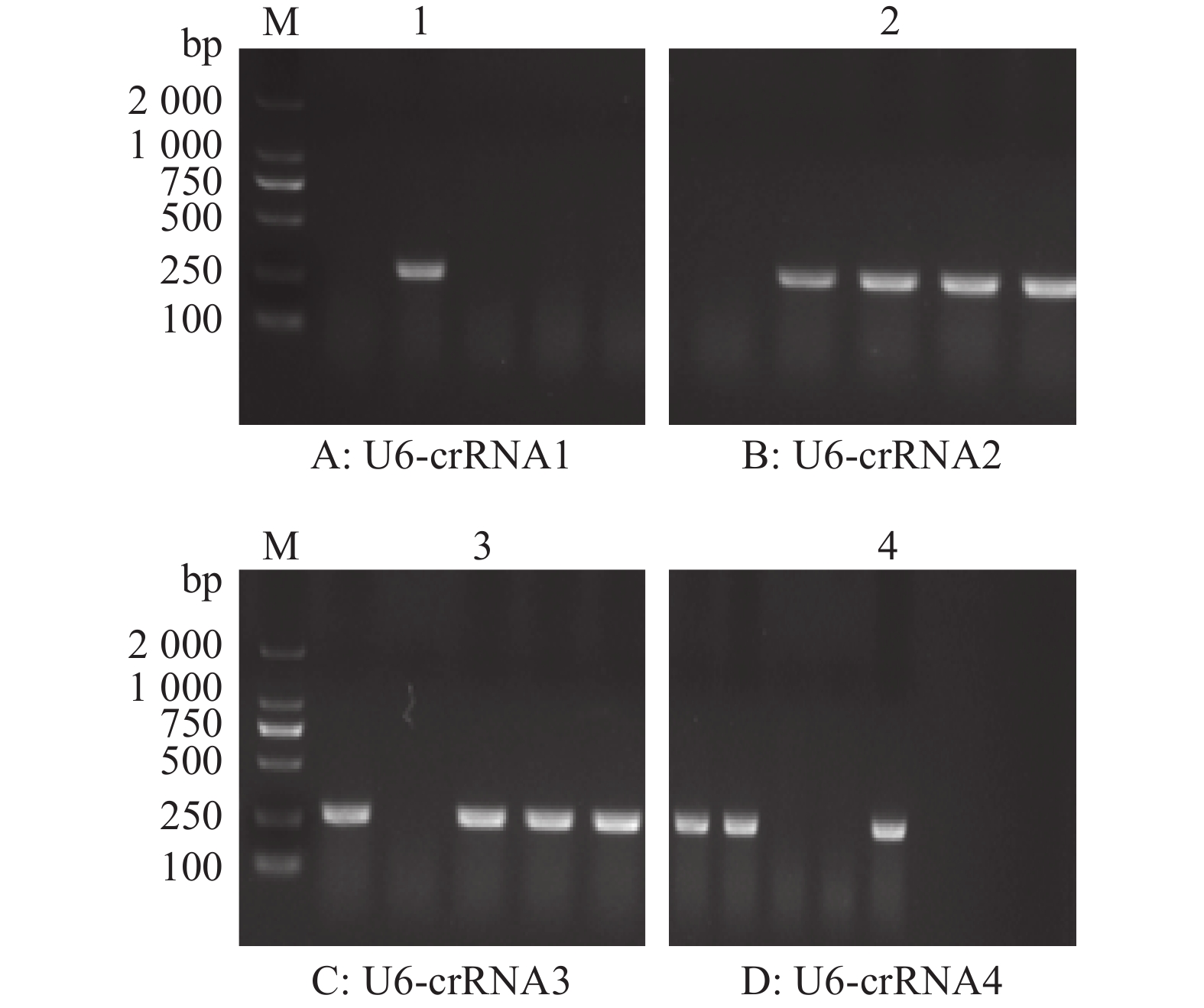
将crRNA序列经过复性后形成带2个黏性末端的双链 DNA,和Bbs Ⅰ酶切线性化后的U6-crRNA骨架质粒(图1)进行连接构建U6-crRNA1、U6-crRNA2、U6-crRNA3和U6-crRNA4载体(分别对应靶点1~4)。重构质粒经菌落PCR鉴定正确(图2)。测序结果如图3所示,显示插入crRNA序列正确。
|
图 1 BbsI酶切U6-crRNA骨架载体电泳图谱 Fig. 1 Electrophoresis of restriction digestion product of U6-crRNA backbone vector by BbsI M:DNA Marker 5000;1:U6-crRNA骨架质粒;2:BbsI酶切U6-crRNA线性化骨架质粒 M: DNA Marker 5000; 1: U6-crRNA backbone plasmid; 2: BbsI digestion of U6-crRNA backbone plasmid |
|
图 2 crRNA连接入骨架载体的阳性质粒PCR鉴定 Fig. 2 PCR result showing positive plasmids with crRNA insertions M:DNA Marker 2000;阳性PCR条带处代表目的crRNA成功构建入U6-crRNA载体;克隆1、2、3和4为被选择用作后续转染试验的U6-crRNA载体 M: DNA Marker 2000;Positive PCR band indicates U6-crRNA vector with successful crRNA insertion; Clone 1, 2, 3 and 4 are four crRNA vectors selected for subsequent cell experiments |

|
图 3 U6-crRNA靶向质粒测序结果 Fig. 3 Sequencing results of U6-crRNA targeting plasmids |
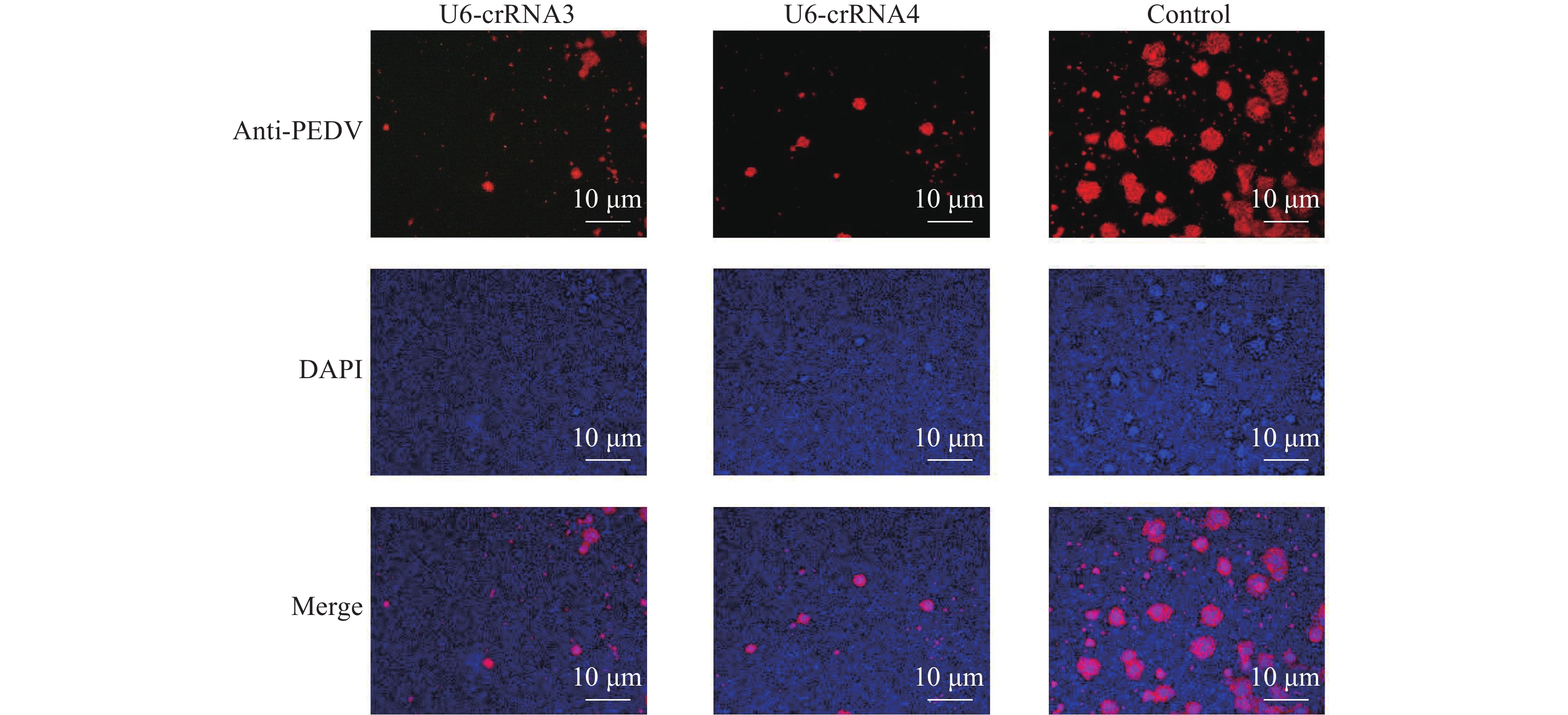
由于Vero细胞转染效率有限,我们在预试验中以相同大小的带荧光蛋白的质粒进行脂质体共转染,确定优化的转染参数。然后我们以相同的转染条件对U6-crRNA和Cas13b质粒进行Vero细胞共转染。将 PEDV ST2(10−3.6 TCID50/mL)以MOI=0.001的稀释度接种于转染48 h后的Vero细胞,24 h后用IFA检测细胞的病毒感染情况。结果显示在24 h后,转染U6-crRNA3和U6-crRNA4打靶质粒的细胞中红色荧光团(代表PEDV感染细胞)与对照组(转染空白crRNA和Cas13b质粒细胞)相比明显减少(图4),而U6-crRNA1和U6-crRNA2组的荧光强度则和对照组差异不大。
|
图 4 PEDV感染Vero细胞24 h的病毒免疫荧光检测 Fig. 4 Viral immunofluorescence assay of Vero cells infected with PEDV for 24 h Vero细胞与PEDV抗体和红色荧光标记的二抗反应(红色),并以DAPI进行细胞核复染(蓝色) Vero cells were labelled with anti-PEDV antibody and further incubated with red fluorescence conjugated secondary antibody (Red); Cell nuclei were counter stained with DAPI (Blue) |
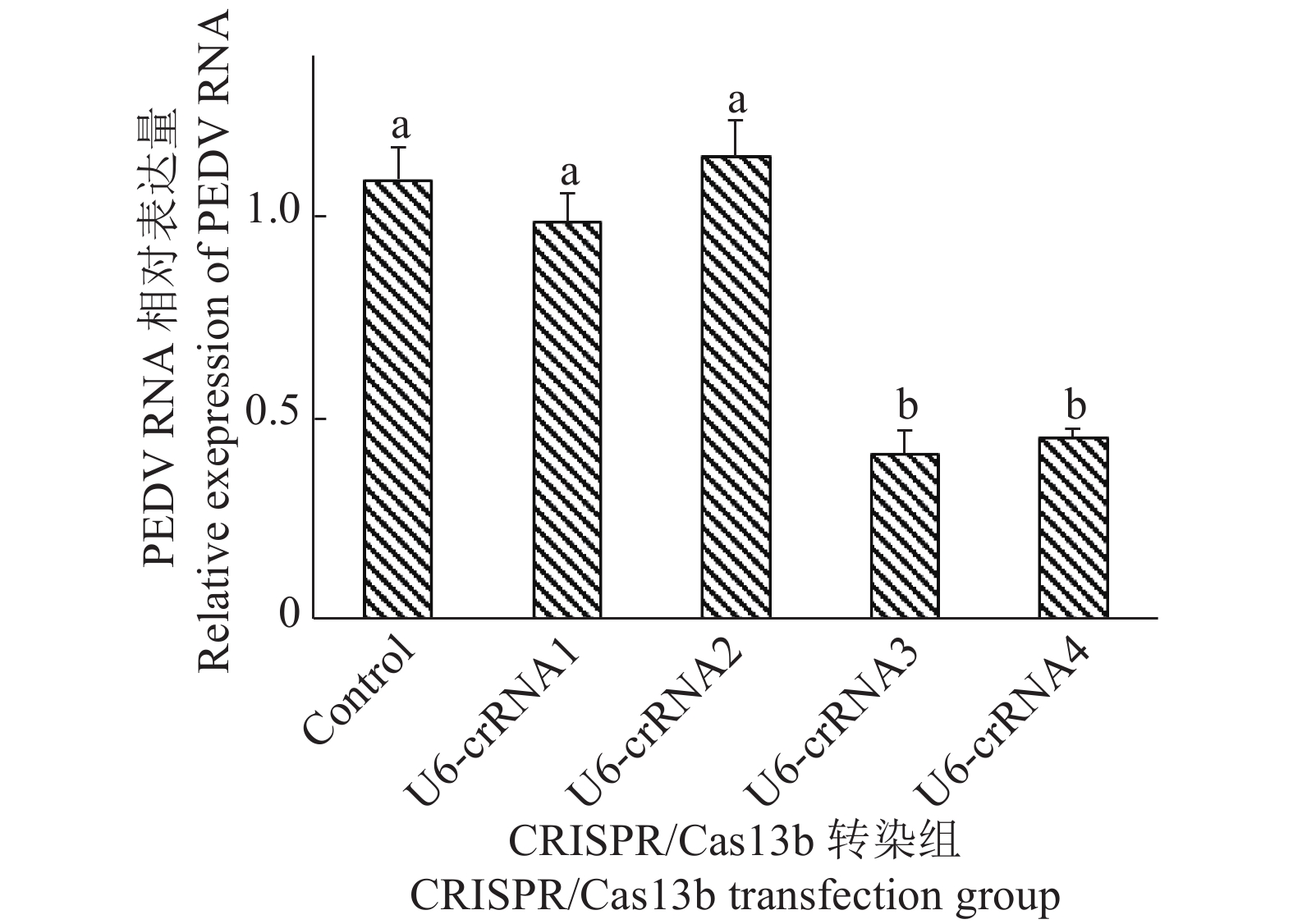
细胞转染48 h后以MOI=0.001的PEDV感染,24 h后收取上清用定量PCR检测细胞上清中的病毒增殖情况。定量PCR结果显示转染U6-crRNA3和U6-crRNA4打靶质粒的细胞组其PEDV Ct值明显大于对照组Ct值,显示了有效的PEDV抑制效果;而U6-crRNA1和U6-crRNA2组Ct值与对照组无明显差异。通过2−△△Ct计算,如图5所示,U6-crRNA3、U6-crRNA4组和对照组相比,PEDV病毒含量减少了约50%,差异显著(P<0.05);U6-crRNA1和U6-crRNA2组与对照组病毒含量差异不显著(P>0.05)。
|
图 5 病毒感染细胞上清中PEDV病毒RNA定量PCR检测 Fig. 5 Quantitative PCR result of PEDV RNA in the supernatant of cells infected by the virus 柱子上方的不同小写字母表示差异显著(P<0.05,Duncan’ s 法) Different lowercase letters on bars indicate significant difference (P<0.05,Duncan’ s test) |
在CRISPR/Cas13可编辑RNA的功能未被发现之前,RNAi被广泛使用在基因沉默中,即可以通过靶向同源mRNA实现真核生物靶基因沉默的方式[14],但是其脱靶率高一直被广为诟病,而Cas13b酶可以在高效敲低RNA表达量的同时实现零脱靶效率,Cox等[10]指出Cas13b与shRNA干扰相比,前者零脱靶,后者的脱靶位点近200个。Cas13酶与Cas9、Cpf1酶等相比,DNA编辑会造成细胞基因组彻底的改变,而Cas13酶则拥有可以敲低RNA而不改变基因组的特性[15],且相较于RNA干扰具有更特异更高效的优势,这为我们在研究各种RNA病毒引起的疾病时提供了新的技术选择。但Cox等[10]发现Cas13a的打靶效率不如Cas13b效果高,通过设计双荧光素酶报告基因法,将荧光素酶表达载体、Cas13a/crRNA和荧光素酶表达载体、Cas13b/crRNA分别转入293细胞中,探测Cas13a和Cas13b对荧光信号的敲低作用,结果表明,PspCas13b的平均敲低率为92.3%,LwaCas13a的平均敲低率为40.1%。在此基础上,为了了解Cas13a和Cas13b在靶向内源转录物上的差异,设计了靶向293细胞KRAS基因的crRNA,PspCas13b的敲低率在60%以上。Wolter等[16]认为是因为Cas13b在与核输出信号(Nuclear export signal,NES)结合后打靶效率更高,因为NES调节许多来自细胞核的蛋白质的输出[17]。近来也有Cas13a系统抑制植物RNA病毒的报道,针对TuMV设计crRNA证明了Cas13a拥有可以干扰TuMV的能力[10, 12, 18]。然而目前仍鲜见文章报道Cas13b在动物RNA病毒上的编辑。
3.2 影响CRISPR/Cas13b切割效率的原因本研究中我们以CRISPR/Cas13b系统对Vero细胞进行瞬时转染,验证其对PEDV病毒感染的敲低能力。但结果显示只有部分靶向crRNA能够有效抑制PEDV的增殖。例如U6-crRNA3和U6-crRNA4在PEDV增殖过程中起到了抑制作用,但U6-crRNA1和U6-crRNA2没有达到预期中的效果。研究认为CRISPR-Cas13b中crRNA-cas(crRNP)效应因子复合体工作时受到靶向RNA的邻间隔序列基序(Protospacer flanking sequence,PFS)序列调节[9, 19],但现在优化后的LwaCas13a、PspCas13b并不需要PFS调节,从结构上看Cas13b和Cas13a都有2个HEPN区用以切割靶向RNA,当Cas13a没有和靶标RNA结合时是失活状态,而在向导RNA和靶标RNA相互配对后,向导RNA和Cas13a蛋白构象发生变化引起Cas13a切割效应激活,所以我们猜测不同的crRNA在与Cas13b形成复合体时会影响Cas13b的活性,现行研究者们根据打靶位点会设置多个打靶crRNA以筛选有效的crRNA;又因为Cas13a在切割靶标RNA的同时也会切割非靶标的RNA[11, 20],基于Cas13b和Cas13a同源性和结构相似性,我们推测可能发生了一些非特异的切割导致不同crRNA的切割效率差异;另外,PEDV感染后的增殖速度极快,这也限制了CRISPR/Cas13b系统对PEDV基因组的高效切割进而抑制PEDV的增殖。因此CRISPR/Cas13b系统对RNA病毒的切割作用仍然需要进一步的优化以提升其靶向RNA的干扰效率。
3.3 CRISPR/Cas13b系统的应用不同于CRISPR/Cas9系统在打靶植物番茄黄叶病毒和人乳头瘤病毒等DNA病毒的应用[21-22],CRISPR/Cas13b系统提出一种新的抗RNA病毒的策略。本研究对Cas13b系统抑制动物RNA病毒进行了报道,其对PEDV的有效抵抗可以为开发新的PEDV治疗手段、制备PEDV抗性动物提供了新途径。同时本研究采用的RNA抑制策略可推广到其他多种RNA病毒的防控上,为Cas13b系统的实际应用提供了广阔的前景,具有广泛的适应性。
| [1] |
PENSAERT M B, DE BOUCKP. A new coronavirus-like particle associated with diarrhea in swine[J]. Arch Virol, 1978, 58(3): 243-247. DOI:10.1007/BF01317606 (  0) 0) |
| [2] |
WANG D L, FANG L R, XIAO S B. Porcine epidemic diarrhea in China[J]. Virus Res, 2016(226): 7-13. (  0) 0) |
| [3] |
HUANG Y W, DICKERMAN A W, PINEYRO P, et al. Origin, evolution, and genotyping of emergent porcine epidemic diarrhea virus strains in the United States[J]. MBio, 2013, 4(5): e713-e737. (  0) 0) |
| [4] |
HSU T, LIU H, CHIN C, et al. Detection, sequence analysis, and antibody prevalence of porcine deltacoronavirus in Taiwan[J]. Arch Virol, 2018, 163(11): 3113-3117. DOI:10.1007/s00705-018-3964-x (  0) 0) |
| [5] |
SONG D, MOON H, KANG B, et al. Porcine epidemic diarrhea: A review of current epidemiology and available vaccines[J]. Clin Exp Vaccine Res, 2015, 4(2): 166-176. DOI:10.7774/cevr.2015.4.2.166 (  0) 0) |
| [6] |
陈申秒, 牛成明, 何福庆, 等. 猪流行性腹泻病毒研究进展及疫苗应用前景[J]. 中国畜牧兽医, 2014(3): 223-229. (  0) 0) |
| [7] |
ABUDAYYEH O O, GOOTENBERG J S, KONERMANN S, et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector[J]. Science, 2016, 353(6299): aaf5573. DOI:10.1126/science.aaf5573 (  0) 0) |
| [8] |
MAKAROVA K S, WOLF Y I, ALKHNBASHI O S, et al. An updated evolutionary classification of CRISPR-Cas systems[J]. Nat Rev Microbiol, 2015, 13(11): 722-736. DOI:10.1038/nrmicro3569 (  0) 0) |
| [9] |
YAN W X, CHONG S, ZHANG H, et al. Cas13d is a compact RNA-targeting Type VI CRISPR effector positively modulated by a WYL-domain-containing accessory protein[J]. Mol Cell, 2018, 70(2): 327-339. DOI:10.1016/j.molcel.2018.02.028 (  0) 0) |
| [10] |
COX D B T, GOOTENBERG J S, ABUDAYYEH O O, et al. RNA editing with CRISPR-Cas13[J]. Science, 2017, 358(6366): 1019-1027. DOI:10.1126/science.aaq0180 (  0) 0) |
| [11] |
ABUDAYYEH O O, GOOTENBERG J S, ESSLETZBICHLER P, et al. RNA targeting with CRISPR-Cas13[J]. Nature, 2017, 550(7675): 280-284. DOI:10.1038/nature24049 (  0) 0) |
| [12] |
AMAN R, ALI Z, BUTT H, et al. RNA virus interference via CRISPR/Cas13a system in plants[J]. Genome Biol, 2018, 19(1): 1-9. DOI:10.1186/s13059-017-1381-1 (  0) 0) |
| [13] |
MILLER L C, CRAWFORD K K, LAGER K M, et al. Evaluation of two real-time polymerase chain reaction assays for porcine epidemic diarrhea virus (PEDV) to assess PEDV transmission in growing pigs[J]. J Vet Diagn Invest, 2016, 28(1): 20-29. DOI:10.1177/1040638715621949 (  0) 0) |
| [14] |
TIJSTERMAN M, PLASTERK R H. Dicers at RISC; The mechanism of RNAi[J]. Cell, 2004, 117(1): 1-3. DOI:10.1016/S0092-8674(04)00293-4 (  0) 0) |
| [15] |
肖婧, 张宗德. CRISPR技术: 一个新型基因编辑工具所引发的革命[J]. 华西医学, 2018, 33(8): 943-949. (  0) 0) |
| [16] |
WOLTER F, PUCHTA H. The CRISPR/Cas revolution reaches the RNA world: Cas13, a new Swiss army knife for plant biologists[J]. Plant J, 2018, 94(5): 767-775. DOI:10.1111/tpj.13899 (  0) 0) |
| [17] |
MCBRIDE K M. Nuclear export signal located within the DNA-binding domain of the STAT1 transcription factor[J]. EMBO J, 2000, 19(22): 6196-6206. DOI:10.1093/emboj/19.22.6196 (  0) 0) |
| [18] |
GOOTENBERG J S, ABUDAYYEH O O, LEE J W, et al. Nucleic acid detection with CRISPR-Cas13a/C2c2[J]. Science, 2017, 356(6336): 438-442. DOI:10.1126/science.aam9321 (  0) 0) |
| [19] |
TERNS M P. CRISPR-based technologies: Impact of RNA-targeting systems[J]. Mol Cell, 2018, 72(3): 404-412. DOI:10.1016/j.molcel.2018.09.018 (  0) 0) |
| [20] |
LIU L, LI X, MA J, et al. The molecular architecture for RNA-guided RNA cleavage by Cas13a[J]. Cell, 2017, 170(4): 714-726. DOI:10.1016/j.cell.2017.06.050 (  0) 0) |
| [21] |
ALI Z, ABULFARAJ A, IDRIS A, et al. CRISPR/Cas9-mediated viral interference in plants[J]. Genome Biol, 2015, 16(1): 238-249. DOI:10.1186/s13059-015-0799-6 (  0) 0) |
| [22] |
SCHINDELE P, WOLTER F, PUCHTA H. Transforming plant biology and breeding with CRISPR/Cas9, Cas12 and Cas13[J]. FEBS Lett, 2018, 592(12): 1954-1967. DOI:10.1002/1873-3468.13073 (  0) 0) |
 2019, Vol. 40
2019, Vol. 40


