犬瘟热病毒(Canine distemper virus, CDV)感染食肉动物可引起高发病率和死亡率的犬瘟热(Canine distemper, CD)。临床上,通常表现为发热、厌食、流鼻涕、结膜炎和腹泻,有的还引起角化过度和中枢神经系统症状,导致机体淋巴细胞减少和急性期淋巴细胞增殖抑制[1]。由CDV引起的疾病已有数百年的历史,其自然宿主已由传统的犬科Canidae、浣熊科Procyonidae和鼬科Mustelidae扩大到猫科Felidae、灵猫科Viverridae等所有陆地食肉动物,此外也有偶蹄目Artiodactyla、海豹和非人灵长类自然感染CDV的报道[2-3]。据历史记载,CD最早发现于南美洲大陆,在17世纪期间,CD从南美洲传播到西班牙,在18世纪传到英格兰、意大利和俄罗斯[4]。CD被认为是世界范围内威胁食肉动物健康的最主要疾病之一。
CDV属于副黏病毒科Paramyxoviride麻疹病毒属Morbillivirus,与人类麻疹病毒和牛瘟病毒密切相关[5-6]。CDV粒子呈多形性,多数为球形,病毒粒子的直径为110 ~550 nm,大多数在150 ~330 nm之间,亦有畸形和长丝状的病毒粒子。病毒的基因组由15 690个核苷酸组成,为不分节、非重叠的负链RNA,蛋白基因顺序依次为核蛋白(Nucleoprotein,N蛋白)、磷蛋白(Phophoprotein,P蛋白)和基质膜蛋白(Matrix protein,M蛋白)、融合蛋白(Fusion protein,F蛋白)、血凝素蛋白(Haemagglutinin protein,H蛋白)、大蛋白(Large protein,L蛋白)。P基因还编码2个非结构蛋白。H蛋白和F蛋白是表面糖蛋白,负责与宿主细胞膜的附着和融合,F蛋白是Ⅰ型膜蛋白,能在中性pH条件下使病毒囊膜和宿主细胞质膜融合来介导病毒穿透[7]。CDV H蛋白是由1 824 nt基因编码有607个氨基酸的蛋白,相对分子质量约为76 000~85 000。H蛋白作为CDV的关键蛋白,在感染的第1步与细胞受体结合,并介导宿主免疫反应[8]。H蛋白抗原变异性最高,并且可以反映CDV毒株的遗传变化进程。对不同动物物种和地理环境的CDV毒株的分析表明,地理隔离是影响CDV的H基因发生变异的重要因素[9-11]。因此,H基因可用来进行CDV毒株鉴定和进化树分析,CDV毒株的主要遗传谱系包括American Ⅰ、American Ⅱ、Asia Ⅰ、Asia Ⅱ、Europe和Arctic like[12-15]。
CDV分布广泛,家犬是CDV感染的主要宿主,野生动物以及野生动物区周围的家养犬通常没有接种疫苗,人员和物品的流动增加了CDV传播的风险[16]。尽管疫苗的广泛使用对CD的防控起到了重要作用,但近年来CDV的流行率迅速增长,对全世界毛皮经济动物养殖业和珍稀野生动物造成了极大损失和危害,而灵长类动物感染的不断出现也引起了人们对CDV具有潜在人畜共患病威胁的担忧[17]。本研究通过调查广东省部分地区CDV的流行情况,分析病毒H基因的遗传进化特性,为监测CDV的遗传进化特征提供基础数据。
1 材料与方法 1.1 病料2017—2018年收集了广州市和东莞市的宠物犬病例样本。其中,17份为CD疑似阳性样本(眼鼻口分泌物)。
1.2 引物设计和病毒鉴定下载GenBank上发表的中国广东、香港、吉林地区CDV H基因ORF序列,利用BioEdit软件比对后,使用NCBI的在线引物软件设计引物(CD-F:ATGCTCTCCTACCAAGACAAGG;CD-R:AACGGTTACATGAGAATCTTATACGG),引物由北京六合华大基因科技股份有限公司合成,PCR产物长度为1 824 bp。
用总RNA极速抽提试剂盒(上海飞捷生物科技有限公司)提取临床病料总RNA,获得总RNA后,利用PrimeScript Ⅱ RTase反转录试剂盒(宝日医生物技术有限公司),获得其cDNA。利用PrimeSTAR® Max DNA Polymerase试剂盒(宝日医生物技术有限公司)扩增H基因,扩增产物用10 g/L琼脂糖凝胶电泳鉴定。
1.3 H基因序列分析将所得的17个野毒株H基因ORF序列与传统疫苗株Onderstepoort(EU143737.1)、Lederle( DQ903854.1)、Convac(Z35493.1)以及不同地区的野毒参考株进行分析,运用DNAstar中Megalign软件计算H基因ORF核苷酸序列及其推导的氨基酸序列的相似性,使用MEGA7绘制基于CDV H基因的系统进化树,分析毒株之间的差异性。并利用NetNGlyc 1.0 Server在线软件(http://www.cbs.dtu.dk/services/NetOGlyc/)预测H蛋白的潜在N−糖基化位点。
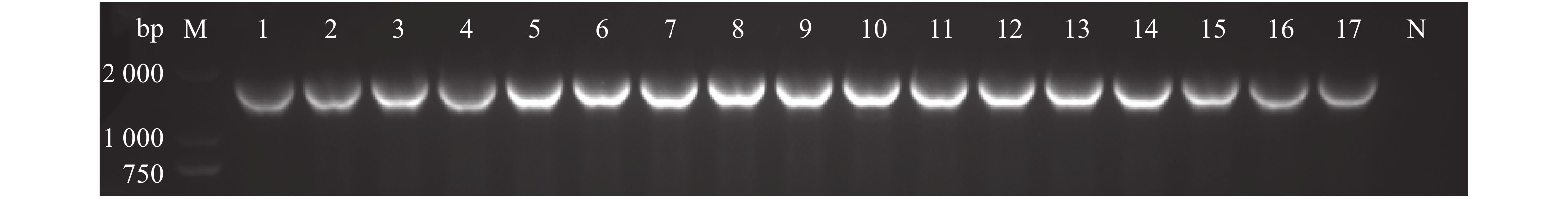
2 结果与分析 2.1 病毒鉴定对RT-PCR扩增产物进行琼脂糖凝胶电泳,结果如图1所示,17份样本均为阳性,条带位置正确。将图中目的片段用胶回收试剂盒分别回收,然后送北京六合华大基因科技股份有限公司进行测序,获得其序列。将获得的17株CDV命名为GD171、GD172、GD173、GD174、GD175、GD176、GD177、GD188、GD189、GD1810、GD1811、GD1812、GD1813、GD1814、GD1815、GD1816和GD1817。H基因测序结果显示,17株CDV H基因序列长度都为1 824 bp,对应编码607个氨基酸。上传其序列至GenBank,获得的登录号为MH091812、MH118274、MH118275、MH118276、MH118277、MH118278、MH118279、MH558134、MH558135、MH558136、MK275197、MK275198、MH558137、MH558138、MK275199、MK275200和MK275201。
|
图 1 17株CDV野毒株H基因的RT-PCR鉴定 Fig. 1 RT-PCR identification of H genes of 17 wild CDV strains M:DL2000 DNA marker;1~17分别为GD171、GD172、GD173、GD174、GD175、GD176、GD177、GD188、GD189、GD1810、GD1811、GD1812、GD1813、GD1814、GD1815、GD1816和GD1817;N:阴性对照 M:DL2000 DNA marker;1−17 represent GD171, GD172, GD173, GD174, GD175, GD176, GD177, GD188, GD189, GD1810, GD1811, GD1812, GD1813, GD1814, GD1815, GD1816 and GD1817 respectively;N:Negative control |
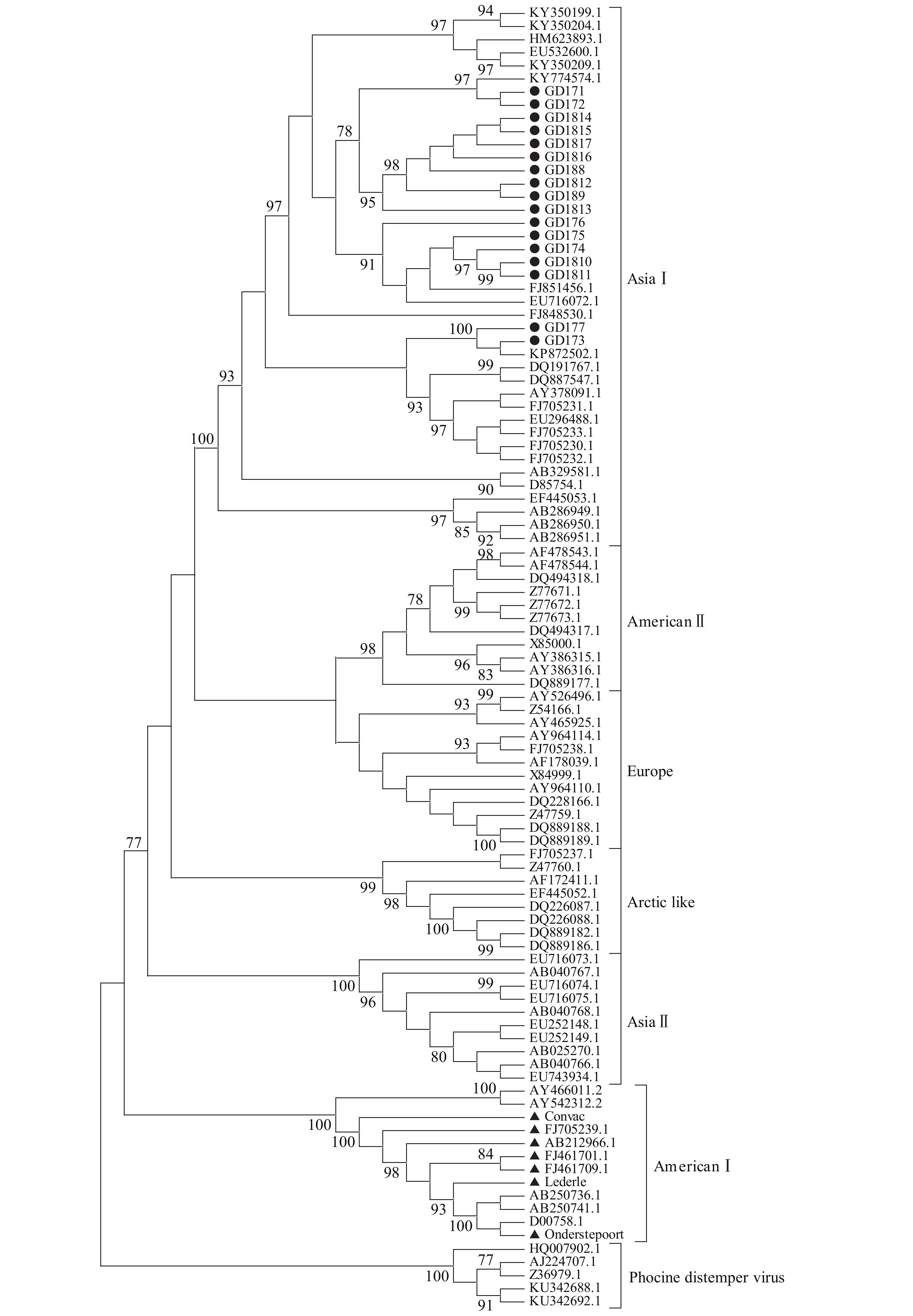
获得病毒H基因ORF序列后,利用MEGA7软件对所得的17个野毒株H基因与传统疫苗株Onderstepoort(EU143737.1)、Lederle( DQ903854.1)、Convac(Z35493.1)以及不同地区野毒株的H基因核苷酸序列进行进化树分析。由图2可知,从系统进化树上可分为两大主要谱系,疫苗株为一分支(American I),Asia II、Asia I等其他毒株为另一分支,并且本研究收集的17株CDV野毒株均为Asia I分支,与疫苗株不在同一分支。
|
图 2 CDV野毒株基于H基因序列的系统进化树 Fig. 2 Phylogenetic tree of CDV strains based on H gene sequences ●为本文分离野毒株;▲为疫苗株 ● Represents identified wild strain; ▲ Represents vaccine strain |
序列相似性分析结果表明(表1),17株CDV之间H基因核苷酸序列及其推导的氨基酸序列的相似性分别为97.4%~100.0%和97.5%~100.0%,而与疫苗株Onderstepoort、Lederle和Convac H基因核苷酸序列及其推导的氨基酸序列的相似性分别为90.3%~91.6%和89.3%~91.8%。可见,17株野毒株之间的序列相似性较高,但与3种传统疫苗株相比对,序列相似性相对较低。
|
|
表 1 CDV H基因的核苷酸和氨基酸序列的相似性1) Table 1 Similarities of nucleotide and amino acid sequences of CDV H genes |
将17株野毒株的氨基酸序列与参考株进行比对分析,发现GD171在344和601位氨基酸分别为赖氨酸和组氨酸,而Asia Ⅰ型毒株和其他分离株分别为精氨酸和丝氨酸;GD171和GD172的第419位氨基酸是苯丙氨酸,而Asia Ⅰ型毒株和其他分离株为亮氨酸;GD1810和GD1811的第65位氨基酸是异亮氨酸,而Asia Ⅰ型毒株和其他分离株为苏氨酸;GD188、GD189、GD1812、GD1813、GD1814、GD1815、GD1816和GD1817的第502位氨基酸是精氨酸,而Asia Ⅰ型毒株和其他分离株为赖氨酸。
2.5 H蛋白潜在的N−糖基化位点分析对分离到的野毒株和其他不同基因型CDV H基因的氨基酸进行潜在的N−糖基化位点预测,结果见表2。由表2可知,第19、149、422和587位是26株CDV都含有的潜在的N−糖基化位点;疫苗株Onderstepoort有4个潜在的N−糖基化位点,Convac和Lederle比其多了第391、456以及603位潜在的N−糖基化位点;本研究分离的17株野毒株(按“2.2”进化树分析证实都为Asia Ⅰ型)的潜在N−糖基化位点互为相同,并且与Asia Ⅰ型相同,都有9个N−糖基化位点,比其他野毒株多了第584位潜在的N−糖基化位点;本研究中涉及的其他野毒株都存在第19、149、309、391、422、456、587以及603位潜在的N−糖基化位点。
|
|
表 2 不同基因型CDV H基因潜在的N−糖基化位点统计 Table 2 Summary of protential N-glycosylation sites in H genes from different genotypes of CDV |
CDV是威胁全世界毛皮经济动物养殖业和珍稀野生动物健康最主要的病原之一。因此,持续监控CDV在犬群中的进化,掌握其遗传变异状况,具有重要意义。H基因编码CDV最重要的抗原蛋白,是研究该病毒遗传进化的主要基因。H蛋白的差异会导致抗原特异性,这使得用单克隆抗体区分野生型和疫苗株H蛋白成为可能[18]。Von Messling等[19]通过反向遗传系统重组的CDV病毒与亲本株比较病毒特性,发现病毒的融合性、生长特性和趋向性主要由H基因介导。不同种属的CDV H蛋白中的第549位氨基酸有显著差异,犬源为549Y,非犬源多为549H[14]。Nikolin等[20]和Sekulin等[21]的研究发现,把犬源CDV H蛋白的第549位氨基酸从Y替换成H后,增加了病毒对其他宿主(猫、师、狨猴)SLAM的适应性,这证明了第549位的突变有可能提高CDV对新宿主适应性。因此,通过研究临床上收集到的病料,分析CDV H基因的抗原性变异是研究CDV病毒基因变异的首选。
本研究根据H基因构建的CDV系统进化树显示,分离出的17株野毒株均位于Asia I分支,表明Asia I型CDV仍为该地区的流行毒株。序列相似性分析发现,分离的野毒株与疫苗株存在较明显的差异,这可能成为免疫失败的原因之一。尽管疫苗的临床应用表明它对预防CDV感染有效,但基于Asia I型的新疫苗是否更能保护犬免受Asia I型CDV的感染值得讨论[22]。
H蛋白β4、β5和β6折叠上第542~544中某些氨基酸位点与细胞受体SLAM或nectin-4结合,进而激活F蛋白,继而病毒囊膜和细胞膜融合,使病毒进入细胞内。研究证实H蛋白D526、I527、R529位点是依赖SLAM结合促发F蛋白构象变化从而引起病毒−细胞融合的关键位点[23-24]。除此之外,对CDV感染角质细胞的融合过程有重大影响的位点分别是H蛋白中的P454、L460、L482、F483、T496、L510、Y520、L522、Y541和Y543[25]。然而,本研究17株野毒株在该位置并无突变,其影响有待进一步探索。另外,17株野毒株之间也存在部分氨基酸位点差异,这些位点是否具备功能性影响也值得进一步研究。
N−聚糖链以共有序列N-X-S/T添加到内质网中的天冬酰胺残基上,其中“X”可以是除了脯氨酸以外的任何氨基酸。N−糖基化是抗原表位一种最重要的修饰,与病毒的毒力密切相关,它对副黏病毒F蛋白和H蛋白的正确折叠、转运和发挥功能方面有重要作用,F或H蛋白至少需要1个或2个N−聚糖才能正常发挥作用[26]。潜在的N−糖基化位点分析表明,本研究的17株毒株的H蛋白潜在N−糖基化位点有9个,比最早疫苗株Onderstepoort多5个,比其他疫苗株多2个。这说明犬瘟热病毒仍然保持着较高的变异率,动物饲主和兽医需要保持持续的关注。
| [1] |
刘雯, 夏志平, 靳朝. 犬瘟热研究进展[J]. 动物医学进展, 2010, 31(8): 83-87. DOI:10.3969/j.issn.1007-5038.2010.08.019 (  0) 0) |
| [2] |
刘玉秀. 犬瘟热病毒敏感细胞系和感染性克隆的构建与初步应用研究[D]. 长春: 吉林大学, 2014.
(  0) 0) |
| [3] |
TONG M, YI L, SUN N, et al. Quantitative analysis of cellular proteome alterations in CDV-infected mink lung epithelial cells[J]. Front Microbiol, 2017, 8: 2564. DOI:10.3389/fmicb.2017.02564 (  0) 0) |
| [4] |
BLANCOU J. Dog distemper: Imported into Europe from South America?[J]. Hist Med Vet, 2004, 29(2): 35-41. (  0) 0) |
| [5] |
DA F B R, STECK A F, NUNES W M, et al. Influence of vaccine strains on the evolution of canine distemper virus[J]. Infect Genet Evol, 2016, 41: 262-269. DOI:10.1016/j.meegid.2016.04.014 (  0) 0) |
| [6] |
DA F B R, VON MESSLING V. Morbillivirus experimental animal models: Measles virus pathogenesis insights from canine distemper virus[J]. Viruses, 2016, 8(10): 274. DOI:10.3390/v8100274 (  0) 0) |
| [7] |
KALBERMATTER D, SHRESTHA N, ADER-EBERT N, et al. Primary resistance mechanism of the canine distemper virus fusion protein against a small-molecule membrane fusion inhibitor[J]. Virus Res, 2019, 259: 28-37. DOI:10.1016/j.virusres.2018.10.003 (  0) 0) |
| [8] |
YUAN P, THOMPSON T B, WURZBURG B A, et al. Structural studies of the parainfluenza virus 5 hemagglutinin-neuraminidase tetramer in complex with its receptor, sialyllactose[J]. Structure, 2005, 13(5): 803-815. DOI:10.1016/j.str.2005.02.019 (  0) 0) |
| [9] |
MARTELLA V, PRATELLI A, CIRONE F, et al. Detection and genetic characterization of canine distemper virus (CDV) from free-ranging red foxes in Italy[J]. Mol Cell Probes, 2002, 16(1): 77-83. DOI:10.1006/mcpr.2001.0387 (  0) 0) |
| [10] |
LI W, LI T, LIU Y, et al. Genetic characterization of an isolate of canine distemper virus from a Tibetan Mastiff in China[J]. Virus Genes, 2014, 49(1): 45-57. DOI:10.1007/s11262-014-1062-z (  0) 0) |
| [11] |
CHEN M, XIN T, HOU S, et al. Genotyping and pathogenic characterization of canine distemper virus based on mutations in the hemagglutinin gene in Chinese domestic dogs[J]. Pol J Vet Sci, 2018, 21: 623-629. (  0) 0) |
| [12] |
LEDNICKY J A, DUBACH J, KINSEL M J, et al. Genetically distant American canine distemper virus lineages have recently caused epizootics with somewhat different characteristics in raccoons living around a large suburban zoo in the USA[J]. Virol J, 2004, 1: 2. DOI:10.1186/1743-422X-1-2 (  0) 0) |
| [13] |
MARTELLA V, CIRONE F, ELIA G, et al. Heterogeneity within the hemagglutinin genes of canine distemper virus (CDV) strains detected in Italy[J]. Vet Microbiol, 2006, 116(4): 301-309. DOI:10.1016/j.vetmic.2006.04.019 (  0) 0) |
| [14] |
MCCARTHY A J, SHAW M A, GOODMAN S J. Pathogen evolution and disease emergence in carnivores[J]. Proc Biol Sci, 2007, 274(1629): 3165-3174. DOI:10.1098/rspb.2007.0884 (  0) 0) |
| [15] |
TAN B, WEN Y J, WANG F X, et al. Pathogenesis and phylogenetic analyses of canine distemper virus strain ZJ7 isolate from domestic dogs in China[J]. Virol J, 2011, 8: 520. DOI:10.1186/1743-422X-8-520 (  0) 0) |
| [16] |
ALEXANDER K A, MCNUTT J W, BRIGGS M B, et al. Multi-host pathogens and carnivore management in southern Africa[J]. Comp Immunol Microbiol Infect Dis, 2010, 33(3): 249-265. DOI:10.1016/j.cimid.2008.10.005 (  0) 0) |
| [17] |
DEEM S L, SPELMEN L H, YATES R A, et al. Canine distemper in terrestrial carnivores: A review[J]. J Zoo Wildl Med, 2000, 31(4): 441-451. DOI:10.1638/1042-7260(2000)031[0441:CDITCA]2.0.CO;2 (  0) 0) |
| [18] |
HAMBURGER D, GRIOT C, ZURBRIGGEN A, et al. Loss of virulence of canine distemper virus is associated with a structural change recognized by a monoclonal antibody[J]. Experientia, 1991, 47(8): 842-845. DOI:10.1007/BF01922469 (  0) 0) |
| [19] |
VON MESSLING V, ZIMMER G, HERRLER G, et al. The hemagglutinin of canine distemper virus determines tropism and cytopathogenicity[J]. J Virol, 2001, 75(14): 6418-6427. DOI:10.1128/JVI.75.14.6418-6427.2001 (  0) 0) |
| [20] |
NIKOLIN V M, OSTERRIEDER K, VON MESSLING V, et al. Antagonistic pleiotropy and fitness trade-offs reveal specialist and generalist traits in strains of canine distemper virus[J]. PLoS One, 2012, 7(12): e50955. DOI:10.1371/journal.pone.0050955 (  0) 0) |
| [21] |
SEKULIN K, HAFNER-MARX A, KOLODZIEJEK J, et al. Emergence of canine distemper in Bavarian wildlife associated with a specific amino acid exchange in the haemagglutinin protein[J]. Vet J, 2011, 187(3): 399-401. DOI:10.1016/j.tvjl.2009.12.029 (  0) 0) |
| [22] |
LI W, CAI C, XUE M, et al. Phylogenetic analysis of canine distemper viruses isolated from vaccinated dogs in Wuhan[J]. J Vet Med Sci, 2018, 80(11): 1688-1690. DOI:10.1292/jvms.18-0116 (  0) 0) |
| [23] |
ZIPPERLE L, LANGEDIJK J P, ORVELL C, et al. Identification of key residues in virulent canine distemper virus hemagglutinin that control CD150/SLAM-binding activity[J]. J Virol, 2010, 84(18): 9618-9624. DOI:10.1128/JVI.01077-10 (  0) 0) |
| [24] |
苏凤艳, 温铁峰, 宗颖, 等. 犬瘟热病毒水貂株H与F蛋白基因原核表达重组质粒的构建及表达[J]. 华南农业大学学报, 2012, 33(3): 417-420. DOI:10.3969/j.issn.1001-411X.2012.03.029 (  0) 0) |
| [25] |
LANGEDIJK J P, JANDA J, ORIGGI F C, et al. Canine distemper virus infects canine keratinocytes and immune cells by using overlapping and distinct regions located on one side of the attachment protein[J]. J Virol, 2011, 85(21): 11242-11254. DOI:10.1128/JVI.05340-11 (  0) 0) |
| [26] |
VON MESSLING V, MILOSEVIC D, CATTANEO R. Tropism illuminated: Lymphocyte-based pathways blazed by lethal morbillivirus through the host immune system[J]. P Natl Acad Sci USA, 2004, 101(39): 14216-14221. DOI:10.1073/pnas.0403597101 (  0) 0) |
 2019, Vol. 40
2019, Vol. 40


