2. 温氏食品集团股份有限公司, 广东 新兴 527400
2. Guangdong Wens Pig Breeding Co., Ltd., Xinxing 527400, China
仔猪初生窝重是出生24 h内全部存活的猪仔体重之和。一般情况下,仔猪初生重越大,仔猪活力越强,抗病力越强[1]。仔猪初生窝重受环境因素的影响较大,尤其是母猪妊娠后期的营养措施[2]。在生产管理较为规范的猪场,仔猪初生窝重则主要受到品种、胎次等因素的影响。有研究表明:仔猪初生重为低遗传力性状,其遗传力在0.10左右[3];对猪繁殖性状的遗传改良,从结合系谱和表型信息的最佳线性无偏预测(Best linear unbiased prediction,BLUP)技术,到结合少数SNP标记信息的标记辅助选择(Marker assisted selection,MAS)技术,再到结合全基因组标记信息的BLUP(Genomic BLUP,GBLUP)技术,选择准确性逐步提高[3]。本研究采用简化基因组测序技术(Genotyping-by-sequencing,GBS)[5]和一步法BLUP(Single-step genomic BLUP,ssGBLUP)技术,对某一大白猪核心群母猪进行基因分型,并分别采用BLUP、GBLUP和ssGBLUP方法,对仔猪初生窝重的遗传估计育种值(Genomic estimated breeding value,GEBV)的选择准确性进行评估;此外,本文对初生窝重的遗传参数进行估计,旨在为仔猪初生窝重的选育提供参考。
1 材料与方法 1.1 材料和测定性状本研究以广东温氏种猪科技有限公司某核心场W64系大白猪为研究对象,并以该场为出生场,选取2010—2019年5月76 710条繁殖性能相关的测定记录,包括产仔数、产活仔数、健仔数、初生窝重、弱仔数、畸形仔数、死胎数和木乃伊数。其中弱差猪数为弱仔数、畸形仔数、死胎数和木乃伊数总和,用其来代表无效仔数,其与健仔数合并构成总产仔数。
基因分型试验样品来自于该场基础母猪核心群近2年来有繁殖记录的母猪,共2 344头,采集其耳样并用75%(φ)乙醇溶液保存。
1.2 简化基因组分型方法本研究采用的简化基因组分型方法参考文献[5]。该方法采用EcoRI和MspI双内切酶对基因组进行切割,并在两端加上能够对个体识别的标签序列,通过PCR扩增和磁珠纯化,来达到富集目的片段的目的,并采用高通量二代测序技术对目标片段进行双端测序。试验流程依次包括:基因组DNA的提取和质检、基因组稀释定量、GBS文库的构建、文库质控、上机测序和测序数据分析。
1.3 数据处理本研究利用Excel剔除缺失值和异常数据,并用Q-Q plot R程序包验证其是否服从正态分布,选取μ±3σ以内的表型数据,结合整理好的数据文件,利用DMUTrace软件追溯群体系谱,并按照DMU软件要求整理为数据文件和系谱文件;同时利用GVCBLUP和BLUPF90软件,分别用GBLUP和ssGBLUP方法计算基因组估计育种值。
DMU软件是一个全面的集合程序。此软件可用于估计正态分布和非正态分布性状的方差-协方差组分[5]。本研究采用的是AI和EM算法相结合的约束性最大似然(REML)方法估计方差组分。
1.4 统计模型 1.4.1 方差分析模型方差分析模型为:
| $ \mathrm{y}=\mathrm{Xb}+{{\mathrm{Z}}_{1}}\mathrm{a}+{{\mathrm{Z}}_{2}}\mathrm{Pe}+\mathrm{e} $ |
式中:y是个体观察值;b是固定效应向量,包括年季效应和胎次效应。a是动物个体加性效应;Pe是永久环境效应;e是残差效应。X、Z1、Z2分别是b、a、Pe的结构矩阵。
使用DMU软件估计性状间遗传相关性。
1.4.2 基因组育种值估计GBLUP模型:与传统的BLUP模型构建原理相似[7],区别在于利用基于SNP信息构建的基因组相关矩阵(G阵)替代常规的基于系谱关系的亲缘关系矩阵(A阵),从而提高GEBV的准确性,其模型如下:
| $ \begin{align} & \ \ \ \ \ \ \ \ \ \ \ \ \mathrm{y}=\mathrm{I }\!\!\mu\!\!\text{ }+\mathrm{Za}+\mathrm{e}, \\ & \mathrm{a}\tilde{\ }\mathrm{N}(0, \mathrm{G}{{\sigma }^{2}}_{a}), \mathrm{e}\tilde{\ }\mathrm{N}(0, \mathrm{w}{{\sigma }^{2}}_{e}), \\ \end{align} $ |
其中,μ表示反应变量y的平均值;a表示个体的加性遗传效应(即个体育种值);e是残差效应;I为单位矩阵;Z为a的关联矩阵。G矩阵按照Vanraden提出的方法构建[7];当反应变量为yc时,w=I,本研究通过GVCBLUP软件来利用GBLUP模型。
一步法GBLUP模型[8]:用H矩阵替代GBLUP中的G矩阵,从而将没有基因型的个体与有基因型的个体放在同一个模型中进行EBV的估计。H矩阵如下:
| $ \begin{align} & \mathrm{H}=\left[ \begin{matrix} {{G}_{\omega }} & {{G}_{\omega }}A_{11}^{-1}{{A}_{12}} \\ {{A}_{21}}A_{11}^{-1}{{G}_{\omega }} & {{A}_{21}}A_{11}^{-1}{{G}_{\omega }}A_{11}^{-1}{{A}_{12}}+{{A}_{22}}-{{A}_{21}}A_{11}^{-1}{{A}_{12}} \\ \end{matrix} \right]~, \text{ } \\ & \ \ \ \ \ \ \ \ \ \ \ \ \ \ \ \ \ \ {{\mathrm{H}}^{-1}}=\left[ \begin{matrix} G_{\omega }^{-1}-A_{11}^{-1} & 0 \\ 0 & 0 \\ \end{matrix} \right]+{{\mathrm{A}}^{-1}} \\ \end{align} $ |
式中,G为有基因型个体组成的矩阵,A为基于系谱的矩阵。
1.5 估计育种值的准确性根据每个个体GEBV的预测误差的方差(Predictor error variance, PEV),通过下列公式计算出对应的GEBV的理论准确性(Re):
| $ {{R}_{\text{e}}}=\sqrt{1-{\text{PEV}}/{\sigma _{a}^{2}}\;} $ |
式中,σa2为加性遗传方差。
2 结果与分析 2.1 初生窝重性状及方差组分估计初生窝重数据量达到76 710条,平均数为15.76 kg,标准差为4.64 kg,最小值和最大值分别为0.50和40.0 kg,另外根据偏度(-0.18)、峰度(0.54)和Q-Q plot图(图 1),可判断初生窝重基本符合正态分布。
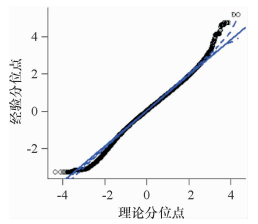
|
图 1 初生窝重的Q-Q plot图 |
初生窝重性状的加性方差、永久环境效应方差分别达到1.414和1.827,残差方差和表型方差分别为14.852和18.093,遗传力为0.08,说明该性状为低遗传力性状,永久环境效应方差占到表型方差的比例为0.10。
2.2 初生窝重与其他繁殖性状的遗传相关性初生窝重与总产仔数、产活仔数和健仔数遗传相关系数分别为0.59、0.68和0.88,为中等偏高的遗传正相关,与弱差猪数遗传相关系数为-0.17,为较低的遗传负相关。
2.3 验证群体基因组选择准确性比较将出生日期在2016年之后的母猪作为验证样本,共546头母猪。使用传统BLUP、GBLUP和ssGBLUP计算方法,在个体表型缺失的情况下,比较GEBV的准确性,并计算了不同方法下,546头母猪GEBV的秩相关系数。初生窝重的GEBV的准确性在BLUP、GBLUP和ssGBLUP计算方法下分别为0.32、0.36和0.38,相对于BLUP计算方法,GBLUP和ssGBLUP准确性分别提升了11.11%和15.79%。结果表明,ssGBLUP预测初生窝重育种值的准确性最高,ssGBLUP预测的初生窝重估计育种值与BLUP估计育种值秩相关达到0.63,相关性最高。
3 讨论与结论 3.1 初生窝重表型和遗传分析本文初生窝重性状记录达76 710条,数据近似服从正态分布,其遗传力估计值均在文献报道范围内[8];此外,遗传相关结果显示,初生窝重与健仔数遗传相关性最高,达到0.88,与相关报道结果较为接近[3]。
3.2 基因组选择准确性比较国际知名猪育种公司PIC和Norsvin,对出生窝重性状GEBV估计的准确性均进行过评估,其参考群均在1 000头以上,采用ssGBLUP方法的评估准确性为0.26~0.46[10-11]。本研究采用ssGBLUP方法,利用2 344头母猪构成基因组选择参考群,其育种值估计准确性为0.38,处于正常范围。但需要注意的是,基因组选择技术在各育种核心群中应用,其遗传基础、数据采集以及由基因分型方法不同导致标记位点数目和重量的差异,会对估计育种值的准确性和精确性造成较大的影响,实际应用中应具体问题具体分析。
3.3 结论本研究以出生窝重性状为研究对象,评估了某大白猪核心育种场出生窝重的遗传参数,估计了其与主要繁殖性状的遗传相关性,通过对该性状的选择,能够有效促进性状的遗传改良,尤其是出生健仔数;另外通过构建大白猪基因组选择参考群体,评价了ssGBLUP能够有效提高基因组选择估计育种值的准确性。通过本研究可以发现,基因组选择能够一定程度上提高初生窝重等低遗传力性状的选择准确性,但是如何将此准确性转化为遗传进展,或是提高生产表现,还需要结合和优化实际生产状况,开发更具性价比和准确性的分型方法,或是需要更加灵活、全面和有效的育种方案。总之,本研究为基因组选择的应用奠定了基础,有利于进一步提高繁殖效率。
| [1] |
刘志伟, 李东, 杨永平, 等. 精准饲养管理模式在母猪妊娠阶段的应用及效益分析[J]. 猪业科学, 2018, 35(2): 122-124. (  0) 0) |
| [2] |
王明. 影响仔猪初生重的因素及提高措施[J]. 现代畜牧科技, 2016(7): 37. (  0) 0) |
| [3] |
施辉毕, 王立刚, 梁晶, 等. 仔猪均匀度遗传参数估计及影响因素分析[J]. 畜牧兽医学报, 2015, 46(12): 2146-2152. (  0) 0) |
| [4] |
IBAÑEZESCRICHE N, GONZALEZRECIO O. Promises, pitfalls and challenges of genomic selection in breeding programs[J]. Spanish Journal of Agricultural Research, 2011, 9(2): 253-266. (  0) 0) |
| [5] |
TAN C, WU Z, REN J, et al. Genome-wide association study and accuracy of genomic prediction for teat number in Duroc pigs using genotyping-by-sequencing[J]. Genet Sel Evol, 2017, 49(1): 35. (  0) 0) |
| [6] |
MADSEN P, SØRENSEN P, SU G, et al. DMU: A package for analyzing multivariate mixed models[C]//8th World Congress on Genetics Applied to Livestock Production. Belo Horizonte: Research Gate, 2006: 247.
(  0) 0) |
| [7] |
VANRADEN P M. Efficient methods to compute genomic predictions[J]. J Dairy Sci, 2008, 91(11): 4414-4423. (  0) 0) |
| [8] |
CHRISTENSEN O F, MADSEN P, NIELSEN B, et al. Single-step methods for genomic evaluation in pigs[J]. Animal, 2012, 6(10): 1565-1571. (  0) 0) |
| [9] |
叶健, 傅金銮, 张锁宇, 等. 安徽省猪育种核心群场间联系性和遗传参数估计[J]. 中国畜牧杂志, 2015, 51(18): 62-67. (  0) 0) |
| [10] |
ANDERSEN-RANBERG I, GRINDFLEK E. Implementation of genomic selection in Norsvin genetic program: Genetic gain in production and maternal traits in Norsvin Landrace[C]//Proceedings of the 10th World Congress Genet. Vancouver, Canada: Appl Livest Prod, 2014: 508.
(  0) 0) |
| [11] |
DEEB N, CLEVELAND M, FORNI S. Genomic selection in pigs: Are we there yet?[C]//International Plant and Animal Genome XX Meeting, San Diego, CA: International Plant and Animal Genome, 2012: W698.
(  0) 0) |
 2019, Vol. 40
2019, Vol. 40


