2. 广东温氏种猪科技有限公司, 广东 新兴 527400
2. Guangdong Wens Pig Breeding Technology Co., Ltd., Xinxing 527400, China
最佳线性无偏预测(Best linear unbiased prediction, BLUP)法自提出到现在仍具有顽强的生命力。对于中等或高等遗传力性状,当系谱信息较完整、表型信息较准确时,BLUP法能准确估计育种值, 从而显著提高遗传进展。随着商业化高密度基因芯片的不断普及以及高通量测序价格的下降,Meuwissen等[1]于2001年首次提出基因组选择(Genomic selection, GS)方法。近年来,GS已逐渐应用于畜禽[2]、作物[3]和水产生物[4]生产中。GS可以实现早期选种,缩短世代间隔,且对低遗传力的性状具有优势。一般而言,基因组预测育种值(Genomic estimated breeding value,GEBV)的准确性高于利用系谱信息预测的EBV[5-6]。相比于传统BLUP法,GBLUP(Genomic BLUP, GBLUP)法通过构建基因组关系矩阵(G矩阵)来替换基于系谱信息构建的分子血缘关系矩阵(A矩阵),而一步法GBLUP(Single-step GBLUP,ssGBLUP)结合系谱与SNP标记信息共同构建H矩阵来替换A矩阵。在生产过程中,ssGBLUP可充分利用已有表型记录、基因组信息和系谱记录等历史数据,会显著提高GEBV的准确性。Fragomei等[7]与Christensen等[8]证明ssGBLUP法对估计育种值的准确性最高。因此,目前在GS应用中估计育种值的主流方法是ssGBLUP[9]。
在生产过程中由于种种原因会导致系谱错误,例如,精液的记录混乱、耳标的缺失、饲养员寄养问题等等。对于有错误的系谱,我们很难及时对其进行纠正,而系谱错误会严重影响ssGBLUP估计育种值的准确性,从而降低遗传进展和生产效益。因此,本研究的目的在于探讨系谱错误对ssGBLUP估计育种值的影响,当系谱错误率达到一定的比例时,可能ssGBLUP估计育种值的准确性会低于GBLUP的。因此,有必要对系谱错误率与EBV准确性的关系进行摸索,以期为今后GS育种提供一定的科学依据和参考。
1 研究方法 1.1 数据来源本研究利用QMSim软件进行数据模拟。QMSim被设计用来模拟家畜的多种遗传结构和种群结构,可以模拟大规模的基因分型数据和复杂的系谱。模拟数据分2步进行,第一,模拟历史世代来创建理想的LD(Linkage disequilibrium)水平,建立遗传突变、漂移等;第二,生成用于进行数据分析和验证的群体[10]。
1.1.1 历史群的模拟为了建立初始的LD以及实现突变-漂移平衡,进行历史种群的模拟,在历史种群无选择、无迁移、随机交配的前提下,通过突变产生新的变异,进而完成历史种群的模拟。历史群体模拟参数如表 1所示。
|
|
表 1 历史群体的模拟 |
在历史种群的最后一代中,随机挑选部分个体作为最近种群的创建者,然后以逐渐增大最后保持不变的速度进行扩群,详细参数见表 2。基因组参数的模拟详见表 3。
|
|
表 2 基础群体的模拟 |
|
|
表 3 基因组参数的模拟 |
对最终产生的20 444个基础群个体进行处理,用于产生参考群体与估计群体。选取最后15 000个个体,使其具有完整的系谱信息与表型信息,分别选取最后1 500、2 500、3 500、4 500和5 500个个体使其具有基因型信息,并将最后500个个体表型信息设置为缺失值,用于估计群体。
将15 000个个体的系谱信息分别按照4%,8%,12%,16%,20%,24%,28%,32%,36%,40%的错误率来将一些个体的系谱随机与附近个体的系谱进行交换,从而得到错误的系谱。
1.3 分析模型| $ \begin{align} & \ \ \ \ \ Y=\mathrm{Xb}+\mathrm{Za}+\mathrm{e}, \\ & \ \ \ \ \ \ \ \ E\left( Y \right)=\mathrm{Xb}, \\ & \ \ \ E\left( \mathrm{a} \right)=E\left( \mathrm{e} \right)=0, \\ & \ \ \ \ \ \ \ \ \ \text{var}\left( \mathrm{a} \right)=\mathrm{G}, \\ & \text{var}\left( \mathrm{e} \right)=\mathrm{R}, \text{cov}\left( \mathrm{a}, \mathrm{e} \right)=0. \\ \end{align} $ |
式中,Y是个体的表型值;b是固定效应向量,包括性别效应;a是动物所有随机效应(育种值)构成的向量;e是残差效应所构成的向量;X、Z分别为b、a的结构矩阵,G和R是已知的正定矩阵。
1.4 统计分析将处理过的数据进行整理,按照BLUPf90软件的要求将其整理为数据文件和系谱文件。
BLUPf90是一个全面的集合程序,主要用于动物育种,该程序可以使用多种方法进行方差组分和育种值估计[11],本研究利用AIREMLF90程序来估计育种值。
Henderson在1984年提出用理论可靠性(Theoretical Reliability, REL)来评价估计育种值的可靠性,其计算方法为:
| $ \text{REL}=1-\text{S}{{\text{E}}^{2}}/V\text{a, } $ |
式中,SE为个体EBV的标准误差;Va(加性遗传方差)由约束性最大似然(REML)方法进行估计。
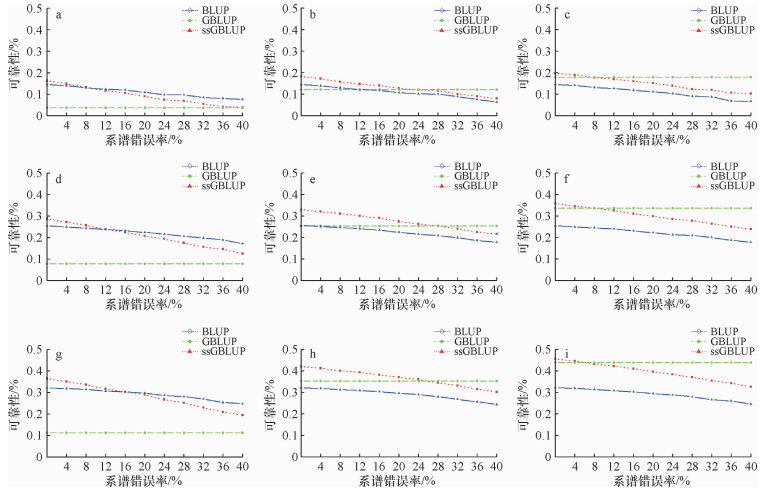
2 结果与分析 2.1 在不同参考群下,系谱错误对ssGBLUP估计育种值可靠性的影响由图 1a、图d和图 1g可知,当参考群个体数量为1 000时,GBLUP估计育种值的可靠性比较低,甚至低于BLUP预测的可靠性,ssGBLUP与BLUP估计育种值的可靠性随着系谱错误率的提高逐渐下降,且ssGBLUP下降的趋势更加明显;当参考群个体数量为3 000时(图 1b、图 1e、图 1h),GBLUP估计育种值的可靠性明显得到显著提高,BLUP估计育种值的可靠性相对于参考群为1 000时基本不变,且当系谱错误率达到24%左右时,ssGBLUP估计育种值的可靠性会低于GBLUP的;当参考群个体数量为5 000时(图 1c、图 1f、图 1i),GBLUP估计育种值的可靠性基本接近与ssGBLUP的可靠性,且当系谱错误率达到8%左右时,ssGBLUP估计育种值的可靠性会低于GBLUP的可靠性。
|
图 1 系谱错误对ssGBLUP估计育种值可靠性的影响 a~c H2=0.1, d~f H2=0.3, g~i H2=0.5;a、d、g参考群个体数量为1 000, b、e、h参考群个体数量为3 000, c、f、i参考群个体数量为5 000 |
由图 1可知,通过纵向对比,在3种遗传力(H2)作用下,随着系谱错误的不断增多,ssGBLUP估计育种值的可靠性(红色线条)与GBLUP估计育种值的可靠性(绿色线条)的交叉点基本一致。结果表明当参考群数量相等,遗传力的不同与系谱错误率对ssGBLUP估计育种值的趋势影响较小。
3 讨论与结论 3.1 参考群的大小与系谱错误率对ssGBLUP估计育种值的影响ssGBLUP估计育种值的可靠性随着系谱错误的增大逐渐降低,两者呈线性关系,GBLUP估计育种值的可靠性保持不变。由于GBLUP没有运用系谱信息,因此其不会因系谱错误而影响可靠性。当参考群较小时,GBLUP估计育种值效果极差[12],而ssGBLUP因为可以充分利用系谱,其预测的可靠性会略高于BLUP,但随着参考群数量的逐渐增多,GBLUP与ssGBLUP估计育种值的可靠性均有明显增加,但GBLUP增加的幅度更大,因此,当参考群逐渐增多时,ssGBLUP对系谱错误的容忍程度逐渐降低。由此得到的启发是,在生产过程中,当参考群足够大时,GBLUP估计育种值的可靠性基本接近于ssGBLUP的可靠性,所以我们可以考虑使用GBLUP来估计育种值以及进行多方面考虑分析。
3.2 遗传力的高低与系谱错误率对ssGBLUP估计育种值的影响随着遗传力的增高,BLUP、GBLUP和ssGBLUP估计育种值的可靠性均明显升高,但由于ssGBLUP与GBLUP增加的幅度基本一致,所以遗传力的高低并不会影响ssGBLUP对系谱错误的容忍程度。根据可靠性公式,遗传力会影响加性遗传方差(Va),对BLUP、GBLUP和ssGBLUP均有影响,影响程度可能不一致,故遗传力的高低与系谱错误对ssGBLUP估计育种值的可靠性的趋势基本没有影响。
3.3 系谱错误的处理杜绝系谱错误无疑是最好的,在现有条件下,可通过加强管理,严格做到先打耳标再调栏,使用电子耳标与普通耳标相结合来减少系谱错误的发生等。未来,随着猪脸识别技术的不断发展[13],发生系谱错误的概率将逐渐降低,甚至没有。当发生系谱错误的时候,尽管很难进行纠正,但依然可以找到一些办法,例如,在现在批次化生产过程中,每一批猪只的父本及母本是确定的,根据基因组信息可以进行亲子鉴定,从而纠正其部分系谱。
3.4 结论本研究分析显示,遗传力的高低与系谱错误对ssGBLUP估计育种值的趋势基本没有影响,当参考群逐渐增大时,ssGBLUP对系谱错误的容忍程度逐渐降低。当参考群个体为3 000时,系谱错误率达到24%左右时,ssGBLUP估计育种值的可靠性会低于GBLUP的;当参考群个体为5 000时,系谱错误率达到8%左右时,ssGBLUP估计育种值的可靠性会低于GBLUP的。
| [1] |
MEUWISSEN T H, HAYES B J, GODDARD M E. Prediction of total genetic value using genome-wide dense marker maps[J]. Genetics, 2001, 157(4): 1819-1829. (  0) 0) |
| [2] |
ROTHSCHILD M F, PLASTOW G S. Applications of genomics to improve livestock in the developing world[J]. Livest Sci, 2014, 166: 76-83. (  0) 0) |
| [3] |
HEFFNER E L, SORRELLS M E, JANNINK J L. Genomic selection for crop improvement[J]. Crop Sci, 2017, 49(1): 1-12. (  0) 0) |
| [4] |
TORO M A, SAURA J M, FERNANDEZ B, et al. Accuracy of genomic within-family selection in aquaculture breeding programmes[J]. J Anim Breed Genet, 2017, 134(3): 256-263. (  0) 0) |
| [5] |
OLSON K M, VANRADEN P M, TOOKER M E, et al. Differences among methods to validate genomic evaluations for dairy cattle[J]. J Dairy Sci, 2011, 94(5): 2613-2620. (  0) 0) |
| [6] |
PSZCZOLA M, CALUS M P L. Updating the reference population to achieve constant genomic prediction reliability across generations[J]. Animal, 2015, 10(6): 7. (  0) 0) |
| [7] |
FRAGOMENI B O, LOURENCO D A L, TSURUTA S, et al. Use of genomic recursions and algorithm for proven and young animals for single-step genomic BLUP analyses:a simulation study[J]. J Anim Breed Genet, 2015, 132(5): 340-345. (  0) 0) |
| [8] |
CHRISTENSEN O F, MADSEN P, NIELSEN B, et al. Single-step methods for genomic evaluation in pigs[J]. Animal, 2012, 10(6): 1565-1571. (  0) 0) |
| [9] |
LEGARRA A, AGUILAR I, MISZTAL I. A relationship matrix including full pedigree and genomic information[J]. J Dairy Sci, 2009, 92(9): 4656-4663. (  0) 0) |
| [10] |
SARGOLZAEI M, SCHENKEL F S. QMSim:A large-scale genome simulator for livestock[J]. Bioinformatics, 2009, 25(5): 680-681. (  0) 0) |
| [11] |
MISZTAL I, TSURUTA S, STRABEL T, et al. BLUPF90 and related programs (BGF90)[C]//Proceedings of the 7th world congress on genetics applied to livestock production. 2002, 33: 743-744.
(  0) 0) |
| [12] |
YAMAGUCHI N, MACDONALD D W. The burden of Co-Occupancy:Intraspecific resource competition and spacing patterns in American mink, mustela vison[J]. J Mammal, 2003, 84(4): 1341-1355. (  0) 0) |
| [13] |
赵静. 猪脸识别技术帮助金融机构获得牲畜"身份证"[J]. 农业知识, 2018(2): 37. (  0) 0) |
 2019, Vol. 40
2019, Vol. 40


