草地贪夜蛾Spodoptera frugiperda原产于美洲热带和亚热带地区,是联合国粮农组织全球预警的重大害虫,2016年开始横扫非洲、亚洲等50多个国家和地区[1-3]。2019年1月份入侵我国以来,已经蔓延到21个省份的1 160多个县(市、区),受害面积超过56万hm2[4]。基于分子标记对草地贪夜蛾的寄主型和遗传结构进行分析,开展指纹图谱数据库构建、区域种群监测、入侵虫源追踪、遗传多样性评价,是科学制定草地贪夜蛾防控技术、保障农业生产安全和保证农产品质量的前提。
目前,对草地贪夜蛾的分子鉴定主要基于CO I(线粒体细胞色素C氧化酶亚基I)和Tpi(Z染色体上编码磷酸丙糖异构酶)2个基因标记展开[5-7]。基于CO I基因片段序列,张磊等[7]对草地贪夜蛾进行了准确的物种鉴定;陈冬平等[8]发现了草地贪夜蛾与黏虫Mythimna separate的杂合子;徐丽娜等[9]发现安徽省草地贪夜蛾样本中86.23%是水稻型、13.77%是玉米型,而且安徽地区样品CO I序列特征与美国佛罗里达州种群高度一致。基于Tpi基因序列的单倍型分析发现,入侵我国云南的草地贪夜蛾均为玉米型,入侵广东省广州市和安徽省的均为玉米型,且在第126、127位碱基为GA/AT杂合[7-9]。CO I和Tpi这2种标记有机结合虽然能够提高鉴定的准确率,但是仍然无法全面了解草地贪夜蛾的群体多样性、群体遗传结构以及虫源动态。
SSR标记(Single sequence repeat marker)具有在基因组中的随机分布性以及高水平的等位多态性,因此广泛用于生物多样性研究、品系分类、遗传图谱构建、分子标记辅助选择和比较基因组研究等领域[10]。Arias等[11]基于来自3个地理群体的15个个体,利用焦磷酸测序法开发了192个SSR标记,用于美国草地贪夜蛾的群体遗传结构分析。Pavinato等[12]利用基因组富集的方法,开发了6个SSR标记,用于巴西草地贪夜蛾的群体遗传结构分析。Arias等[11]采用9个SSR标记对北美的草地贪夜蛾进行遗传分析,结果表明该地区草地贪夜蛾经历了虫口的扩张,虽然基因流与氟苯二胺和氟苯脲敏感性之间不存在相关性,但是发现具有较高LC50的群体扩散到其他的地区,并保持较高的抗性基因频率[13]。
本研究拟通过CO I和Tpi分子标记以及微卫星DNA标记,对入侵我国广东、湖南和广西3个省的6个市(区、县)的草地贪夜蛾进行分子鉴定,为监测虫源扩散规律、预测预警虫情和制定科学的防控方案提供参考。
1 材料与方法 1.1 供试昆虫采集本次供试草地贪夜蛾采集于广东省广州市花都区花山镇儒林村(GDHD)、湖南省张家界慈利县通津钱铺(HNZJJ)、广西南宁宾阳县古济(GXNN)、广东省东莞市(GDDG)、广东省清远市连山壮族瑶族自治县(GDQY)5个地区玉米地和广东省韶关市翁源县(GDSG)甘蔗地。研究样品共有42份,均为2~6龄幼虫。样品采集时间为2019年5月18日至6月19日,寄主为喇叭口期的春玉米。单头活体幼虫采集后放入含有新鲜玉米叶片的50 mL离心管,带回实验室进行形态学鉴定,编号拍照后,对3龄以上幼虫切取头胸部,3龄以下幼虫取整头虫体,放入2 mL离心管用液氮速冻,−80 ℃储存,用于DNA提取(表1)。
|
|
表 1 供试幼虫样本信息表 Table 1 Information of larva samples used in this study |
DNA提取采用微量样品基因组DNA提取试剂盒(TIANGEN,中国),用于PCR扩增的2对引物序列为CO-A和Tpi。PCR扩增按照陈冬平等[8]所建立的方法进行,采用PrimeSTAR® Max DNA Polymerase PCR(TaKaRa,日本)进行PCR扩增,反应体系包括PrimeSTAR® Max DNA Polymerase PCR(2×)10 μL,上游引物和下游引物各0.4 μL,模板1 μL,加ddH2O至20 μL。PCR反应为:98 ℃ 预变性2 min;98 ℃变性10 s,60 ℃退火15 s,72 ℃延伸 30 s,35个循环;72 ℃延伸5 min。使用15 g/L的琼脂糖凝胶电泳检测扩增片段特异性后,PCR产物由广州天一辉远基因科技有限公司进行切胶测序。
1.3 SSR标记的筛选参考Arias等[13]所采用的12对SSR标记, 随机选取5个样品进行引物筛选。选取其中扩增稳定、电泳条带清晰和多态性丰富的9对引物spf05、spf1502、spf1592、spf1706、spf343、spf670、spf789、spf918和spf997用于群体结构遗传分析。
SSR引物筛选的PCR体系包括TaKaRa Taq (5 U/μL) 0.125 μL, 10×PCR Buffer (Mg2+ Plus) 2.5 μL, dNTP Mixture (2.5 mmol/L) 2 μL,上游和下游引物(10 μmol/L)各0.4 μL,模板1 μL,加ddH2O至25 μL。PCR反应程序:94℃ 预变性2 min;94 ℃变性10 s,55 ℃退火30 s,72 ℃延伸 30 s,35个循环;72 ℃延伸5 min。PCR产物加入6 μL 6×Loading buffer后,采用6%(φ)变性聚丙烯酰胺胶电泳检测。电泳在DYCZ-30C电泳槽(北京六一)中进行。上样3 μL,以DL1000为DNA分子量标准,120 W恒功率电泳2 h后,银染显色。
1.4 SSR标记的基因分析仪检测本研究将M13 通用接头序列(TGTAAAACGACGGCC AGT)加到每对SSR标记的正向引物的 5′ 方向,并合成带不同荧光基团的M13接头序列。带荧光的PCR产物经DNA测序仪ABI 3730XL(Applied biosystems, USA)进行荧光电泳检测,并使用软件GeneMarker V2.2.0 对原始数据进行条带分型。PCR反应体系和扩增程序如下: 2×Taq PCR Master Mix 7.5 μL, 上游和下游引物(10 μmol/L)各1.0 μL, 模板1 μL,加ddH2O至10 μL。PCR扩增流程:96 ℃预变性3 min;96 ℃ 变性30 s,60 ℃ 退火30 s,72 ℃ 延伸1 min,30循环;72 ℃ 延伸10 min;12 ℃保存。取3 μL荧光PCR产物进行琼脂糖凝胶电泳鉴定,检测PCR条件是否单一、片段是否与预期一致,混样上机检测。混样体系为Hi−Di甲酰胺10 μL、内标 0.5 μL和PCR产物1.0 μL。
1.5 数据读取使用GeneMarker软件,导入基因分析仪上的原始结果文件,根据位点信息设置分析Panel;使用GeneScan 500 LIZ Size Standard进行数据分析。核查Size standard评分,剔除评分小于0.8的数据,导出Excel格式的等位基因信息。
1.6 数据分析利用Lastergene软件包对正反向测序序列进行人工拼接、峰型校正和序列比对。已经发布的草地贪夜蛾CO I和Tpi基因序列从GenBank数据库下载。针对已报道的基于CO I和Tpi基因序列的水稻型和玉米型单倍型特点,分别进行差异位点分析,确定本研究草地贪夜蛾的具体亚型。
用GeneAIEx软件计算各位点的观测等位基因数(NA)、有效等位基因数(NE)、Shannon信息指数(I)、观测杂合度(HO)和期望杂合度(HE)等参数;使用Phylip软件的UPGMA方法根据Nei’ s遗传距离矩阵绘制系统发育树;遗传结构分析在Structure软件完成后,用Harvester在线工具(
利用COI-A引物对所采集的42份样品扩增,得到预期大小为543 bp的单一特异性条带并进行双向测序。测序结果去除两端测序质量较低的序列后,对521 bp的序列进行了分析。结果(表2)发现,42份样品存在3种CO I序列类型,其中,HNZJJ-30#和GDHD-7#两者之间存在8个SNP位点,分别与草地贪夜蛾玉米型(HM136592)和水稻型(HM136602)相似性为100%;第3种类型是非草地贪夜蛾CO I序列,与陈冬平等[8]报道的HD3#-2序列完全一致,与黏虫Mythimna separate (KX863042) 对应的片段序列仅存在1个碱基的差异,说明此前观察到的草地贪夜蛾和黏虫的杂合型在本研究也能够检测到。在供试的样品中,湖南张家界群体有8个样品为水稻型,2个为玉米型,1个为草地贪夜蛾水稻型和黏虫的杂合型,其余群体均为水稻型单倍体型(表2)。
|
|
表 2 草地贪夜蛾2种单倍型CO I基因序列多态性位点比较 Table 2 Polymorphic site distribution of CO I gene sequence of haplotypes in Spodoptera frugiperda populations |
基于Tpi序列对42个草地贪夜蛾样品进行序列分析,结果见表3。由表3可见,检测样品的Tpi基因序列的差异位点主要发生在第126和127碱基处,该位点存在玉米型(AT)、水稻型(GA)和杂合型(AT/GA)3种类型。在广东花都群体中存在9个AT型、3个GA型和3个AT/GA型;广东东莞和广东清远2个群体均为AT型;湖南张家界、广西南宁和广东韶关3个群体存在AT型和AT/GA型。
|
|
表 3 CO I和Tpi标记鉴定的草地贪夜蛾42个样品单倍型样品数量 Table 3 The number of haplotype of 42 Spodoptera frugiperda samples identified by COI and Tpi markers |
9对SSR标记的多样性参数见表4。48头草地贪夜蛾样品(42头为本研究收集的样品、6头为陈冬平等[8]收集的样品)共检测到40个等位基因,平均每个位点检测到4.40个等位基因。其中,spf1592位点上拥有最多的有效等位基因(7个),spf1706位点上的等位基因仅有2个。各微卫星位点的表观杂合度平均值为0.639,预期杂合度平均值为0.546。各位点等位基因表观杂合度与预期基本一致,符合Hardy-Weinberg平衡。
|
|
表 4 9个SSR位点的遗传变异参数、F-statistics统计值和基因流1) Table 4 Indices of gene diversity,F-statistics values and gene flow deduced by nine SSR loci |
种内近交系数FIS平均值为−0.126,依据Nagylaki等[14]理论,FIS为负数表明被检测的种群在9个SSR位点杂交现象严重,尤其是在spf918、spf343和spf789位点更为明显。spf05和spf997位点存在较高的FIT值,分别为0.465和0.303,说明在该位点可能存在近交现象。种群间分化系数FST在0.094~0.244之间,平均值为0.151,表明仅有15.1%的遗传变异发生在种群间,发生在种群内的遗传变异高达84.9%。有研究表明,FST大于0.25时,种群间有极大的遗传分化;FST在0.15~0.25之间时,种群间明显分化;FST在0.05~0.15之间时,种群存在中等程度的分化;FST小于0.05时,种群间几乎无分化[14-16]。依据上述标准,在检测的9个位点处,spf05、spf1502、spf670、spf789和spf997存在明显分化,其余4个位点spf1592、spf1706、spf343和spf918为中度分化。
在种群的水平上,种群间的基因流参数NM范围在0.205~2.407之间,NM 平均值为1.594,NM>1表明基因流足以防止遗传分化发生[17],spf1502和spf997基因流水平非常低,种群在此位点非常容易因遗传漂变而分化。
2.3.2 群体多样性水平分析广东清远因样品数目仅有1头,不列入本项分析。由表5可见,各群体的观测等位基因数NA为2.333~8.444,平均为4.407; 有效等位基因数NE为2.071~4.374,平均为3.020;Shannon信息指数I为0.649~1.594,平均为1.067; 表观杂合度 HO为0.556~0.683,平均为0.640;预期杂合度HE为0.389~0.685,平均为 0. 546;固定指数F为−0.426~0.087,平均为−0.252。各种群表观杂合度和预期杂合度没有明显差异,说明各群体不存在连锁不平衡。除广东花都群体外,其余群体的F均为负值,表明广东花都群体的预期杂合度相对较高,其余群体预期杂合度较低。
|
|
表 5 草地贪夜蛾群体遗传多样性指数1) Table 5 The genetic diversity indexes of Spodoptera frugiperda populations |
各群体间的Nei’s标准遗传距离(D)和遗传相似性(I)见表6。广东清远因样品数目仅有1头,不列入本项分析。其余5个群体两两之间的I为0.670~0.895,均大于0.6,表明各种群之间遗传相似性较高。广东东莞群体和广西南宁群体之间的D最大(0.400),广东花都群体和湖南张家界群体之间的D最小(0.111)。其中,广东花都群体与其他地理群体的D为0.111~0.185,均低于其他各群体间遗传距离。UPGMA聚类分析显示5个群体聚合成为一个大的分支,广东花都群体与湖南张家界群体形成一个最近的分支(图1)。
|
|
表 6 草地贪夜蛾不同地理种群的Nei’ s遗传距离(D)和遗传相似性(I)1) Table 6 Genetic distance (D) and genetic identity (I) between different geo-populations of Spodoptera frugiperda |

|
图 1 草地贪夜蛾群体间Nei’ s标准遗传距离的UPGMA法聚类图 Fig. 1 UPGMA cluster analysis using the Nei’ s standard genetic distance indices for Spodoptera frugiperda |
PCoA分析表明,5个群体在二维平面上主要分为3个区域(图2)。第1个区域以湖南张家界群体为主,第2个区域以广东省的3个地区群体为主,混杂了湖南和广西的个体,第3个区域为广西南宁群体的3 个特殊个体(SF039、SF034和SF05)。从PCoA分析图可以看出,总体而言,湖南张家界群体和广西南宁群体含有的特异遗传个体较多,且后者的分布区域较为分散

|
图 2 基于SSR标记的草地贪夜蛾群体的主坐标分析 Fig. 2 Principal coordinate analysi based on SSR markers for Spodoptera frugiperda populations 1:湖南张家界群体为主;2:广东三地群体为主;3:广西南宁群体为主 1:The populations were mainly from Zhangjiajie of Hunan;2:The populations were mainly from three cities (district) of Guangdong;3:The populations were mainly from Nanning of Guangxi |
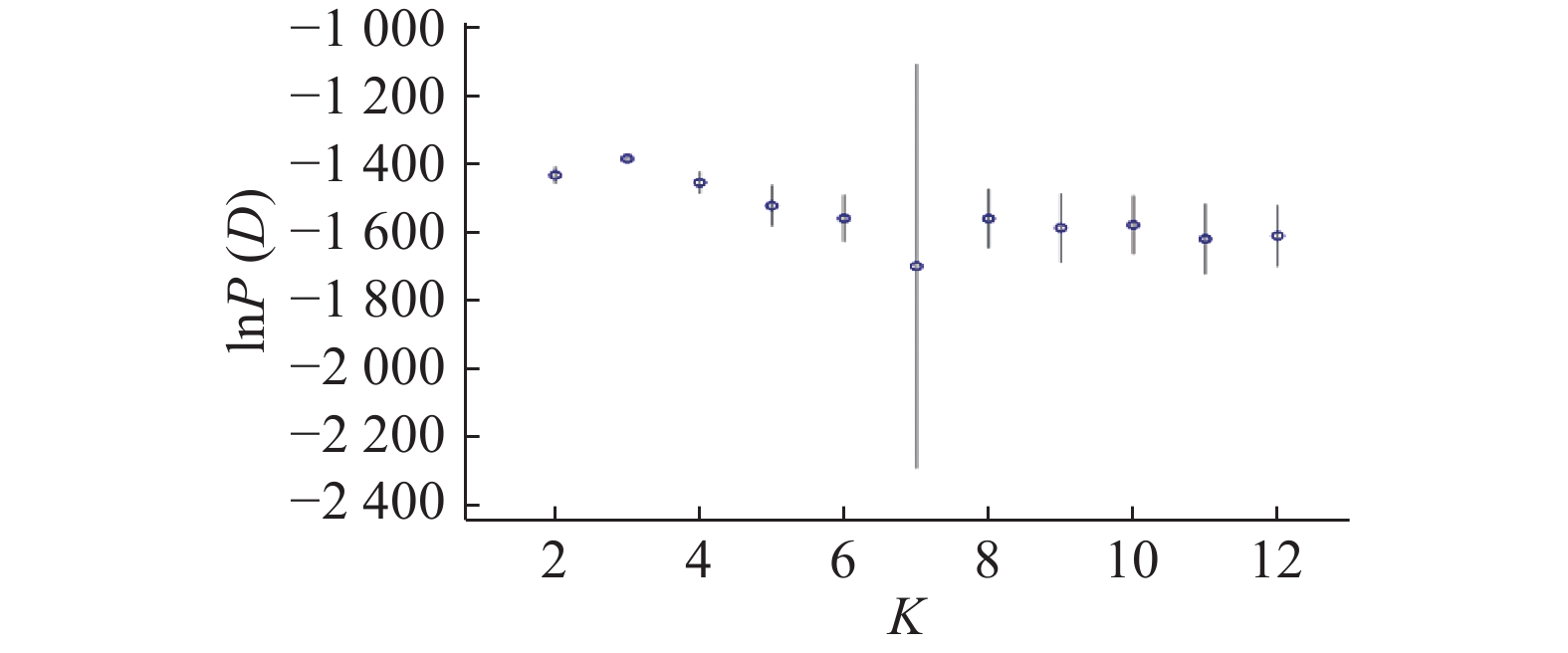
42个草地贪夜蛾的基因变异频率在K=3时出现拐点,此时ΔK值最大。当K=4时,lnP(D)取值较大且前后变化平缓,由此将供试材料分为3~4个类群较为合理(图3和图4)。群体遗传结构的分析结果(图5)表明,3种颜色表示所有样本可以分为3个群体以及每个样本种源基因所占的比例。3个类群均包含了来自不同地理群体个体的遗传特征,它们之间不存在显著的遗传分化。

|
图 3 K值与ΔK 值折线图 Fig. 3 The broken line graph of ΔK and K values |
|
图 4 K值与lnP(D)平均值 Fig. 4 The value of K and the mean of ln P(D) |

|
图 5 草地贪夜蛾群体遗传结构图(K=3) Fig. 5 Structure analysis for Spodoptera frugiperdasamples |
基于CO I基因片段的序列,对中南三省区的草地贪夜蛾单倍型的鉴定结果显示,被检测的42个样品中,39个样品为水稻型单倍体型,2个样品为玉米型。2个玉米型的个体均来自湖南张家界群体,说明入侵该地区的虫源可能不是单一的。在湖南张家界还检测到1个草地贪夜蛾/黏虫杂合型,说明入侵的草地贪夜蛾能够与本地的近缘种黏虫发生杂交[8]。
Tpi基因片段测序的结果则显示,成功测序的42个样品中,27个被鉴定为玉米型,3个为水稻型,12个为杂合型。与陈冬平等[8]报道的结果相比,广东花都出现了水稻型单倍型纯合个体,说明入侵群体在繁殖的过程中逐渐纯化。
由于CO I基因位于线粒体基因组,具有母系遗传特征,因此母本水稻型和父本玉米型产生杂交,后代获得了水稻型线粒体的 CO I基因序列,Tpi核基因组在繁衍扩散过程中更多地保留了玉米型的基因组[18]。大部分水稻型CO I单倍体型个体的存在说明了该类型的雌虫在生存和交配中具有绝对的优势。
3.2 群体的遗传多样性与遗传结构9 个SSR标记共检测到40个等位基因,平均每个位点检测到的等位基因数目为4.40,低于Arias [13]报道的平均每个位点的等位基因数目为6.49,暗示着群体的总体多样性比巴西群体低。
基因流是指生物个体从其发生地分散出去而导致不同种群间基因交流的过程,基因流的发生能够弱化群体间的遗传差异,使群体趋于一致。NM>1时能有效抑制由于遗传漂移引起的遗传分化;当NM<1时表明基因流不足以抵制种群内因遗传漂变引起的种群分化[17]。5个草地贪夜蛾群体间的NM平均值为1.594,说明各群体间的遗传分化不是很大。草地贪夜蛾入侵我国的时间较短,本次检测的样品大部分为当地初次发生的样品,因此未形成明显的遗传分化。
但是,广东东莞群体与广西南宁群体之间的遗传距离高于其与湖南张家界群体的遗传距离,且广东花都群体与湖南张家界群体聚类为一个最近的分支,说明可能受云贵高原的阻隔,从云南省入侵的草地贪夜蛾未能直线入侵湖南张家界,该地区的个体可能更多来自广东省群体的北迁,而广东群体可能由广西群体迁入。上述结果也说明,遗传距离与实际距离之间不一定呈正相关,天气背景场和地理隔离起更重要的作用。PcoA分析发现来自广西南宁的个体一部分位于广东个体所在区域,一部分位于独立的区域,说明入侵广西的草地贪夜蛾来源可能并不单一。
明确草地贪夜蛾入侵我国后如何传播、迁移、扩散和定殖的问题,是科学制定预防和控制策略的迫切要求。本研究基于线粒体标记和核基因标记的分析,探讨了入侵中南三省(区)6个地区的草地贪夜蛾的单倍型和遗传结构,为了解草地贪夜蛾的传播和扩散规律提供了遗传证据。后续研究将进一步提高样本的数量、地区和时间的代表性,为我国草地贪夜蛾群体的遗传分析积累基础性的数据。
| [1] |
TODD E L, POOLE R W. Keys and illustrations for the armyworm moths of the Noctuid genus Spodoptera Guenée from the Western Hemisphere
[J]. Ann Entomol Soc Am, 1980, 73(6): 722-738. DOI:10.1093/aesa/73.6.722 (  0) 0) |
| [2] |
联合国粮食及农业组织. 秋粘虫[Z/OL]. [2019-07-08]. http://www.fao.org/fall-armyworm/zh/.
(  0) 0) |
| [3] |
UZAYISENGA B, WAWRU B, KAJUGA J, et al. First record of the fall armyworm, Spodoptera frugiperda (Lepidoptera: Noctuidae), in Rwanda
[J]. Afr Entomol, 2018, 26(1): 244-246. DOI:10.4001/003.026.0244 (  0) 0) |
| [4] |
魏启文. 草地贪夜蛾入侵中国21个省份, 如何快速监测识别仍是防控痛点[Z/OL]. [2019-07-08]. http://finance.ifeng.com/c/7oNarkr1YVk.
(  0) 0) |
| [5] |
HAJIBABAEI M, JANZEN D H, BURNS J M, et al. DNA barcodes distinguish species of tropical Lepidoptera[J]. P Natl Acac Sci USA, 2006, 103(4): 968-971. DOI:10.1073/pnas.0510466103 (  0) 0) |
| [6] |
NAGOSHI R N, GOERGEN G, TOUNOU K A, et al. Analysis of strain distribution, migratory potential, and invasion history of fall armyworm populations in northern Sub-Saharan Africa[J]. Sci Rep, 2018, 8(1): 3710. DOI:10.1038/s41598-018-21954-1 (  0) 0) |
| [7] |
张磊, 靳明辉, 张丹丹, 等. 入侵云南草地贪夜蛾的分子鉴定[J]. 植物保护, 2019, 45(2): 19-24. (  0) 0) |
| [8] |
陈冬平, 陈志廷, 徐汉虹, 等. 不同分子标记对入侵广州草地贪夜蛾的寄主型鉴别能力比较[J]. 华南农业大学学报, 2020, 41(1): 17-21. (  0) 0) |
| [9] |
徐丽娜, 胡本进, 苏贤岩, 等. 入侵安徽省草地贪夜蛾的遗传分析[J/OL]. 植物保护, [2019-07-28]. https://doi.org/10.16688/j.zwbh.2019348.
(  0) 0) |
| [10] |
ELLEGREN H. Microsatellites: Simple sequences with complex evolution[J]. Nat Genet, 2004, 5(6): 435-445. DOI:10.1038/nrg1348 (  0) 0) |
| [11] |
ARIAS R S, BLANCO C A, PORTILLA M, et al. First microsatellites from Spodoptera frugiperda (Lepidoptera: Noctuidae) and their potential use for population genetics
[J]. Ann Entomol Soc Am, 2011, 104(3): 576-587. DOI:10.1603/AN10135 (  0) 0) |
| [12] |
PAVINATO V A C, MARTINELLI S, DE LIMA P F, et al. Methodology microsatellite markers for genetic studies of the fall armyworm, Spodoptera frugiperda
[J]. Gent Mol Res, 2013, 12(1): 370-380. DOI:10.4238/2013.February.8.1 (  0) 0) |
| [13] |
ARIAS O C E C. Population genetic structure and demographic history of Spodoptera frugiperda (Lepidoptera: Noctuidae): Implications for insect resistance management programs
[J]. Pest Manag Sci, 2019, 75(11): 2948-2957. DOI:10.1002/ps.5407 (  0) 0) |
| [14] |
NAGYLAKI T. Fixation indices in subdivided populations[J]. Genetics, 1998, 148(3): 1325-1332. (  0) 0) |
| [15] |
师沛琼, 杨茂发, 吕召云, 等. 贵州省烟青虫遗传多样性[J]. 中国农业科学, 2014, 47(9): 1836-1846. DOI:10.3864/j.issn.0578-1752.2014.09.019 (  0) 0) |
| [16] |
吕召云, 杨茂发, 师沛琼, 等. 贵州省烟蚜遗传多样性分析[J]. 中国农业科学, 2013, 46(13): 2685-2694. DOI:10.3864/j.issn.0578-1752.2013.13.007 (  0) 0) |
| [17] |
SLATKIN M, BARTON N H. A comparison of three indirect methods for estimating average levels of gene flow[J]. Evolution, 1989, 43(7): 1349-1368. DOI:10.1111/j.1558-5646.1989.tb02587.x (  0) 0) |
| [18] |
张磊, 柳贝, 姜玉英, 等. 中国不同地区草地贪夜蛾种群生物型分子特征分析[J/OL]. 植物保护, [2019-07-08]. https://doi.org/10.16688/j.zwbh.2019296.
(  0) 0) |
 2020, Vol. 41
2020, Vol. 41


