2. 广东省动物源性人兽共患病预防与控制重点实验室,广东 广州 510642;
3. 广东省兽医临床重大疾病综合防控重点实验室,广东 广州 510642
2. Key Laboratory of Zoonosis Prevention and Control of Guangdong Province, Guangzhou 510642, China;
3. Guangdong Provincial Key Laboratory of Prevention and Control for Severe Clinical Animal Diseases, Guangzhou 510642, China
猪繁殖与呼吸综合征(Porcine reproductive and respiratory syndrome,PRRS)是一种以母猪繁殖障碍和各种生长阶段猪的呼吸系统疾病为特征的病毒性传染病,俗称“猪蓝耳病”,其病原为猪繁殖与呼吸综合征病毒(Porcine reproductive and respiratory syndrome virus,PRRSV)[1-2]。PRRS于20世纪80年代末首先在美国被发现,临床上主要表现为母猪晚期繁殖障碍和新生仔猪的呼吸道疾病[3]。随后该病相继在北美、欧洲、亚洲的养猪国家广泛流行,并引发了空前的“流产风暴”[4],给世界养猪业造成了巨大的经济损失。
2006年,在我国南方部分省份出现的“猪无名高热”病,经鉴定其病原为以JXA1株为代表的高致病性PRRSV,引发了严重的PRRS疫情[5-6]。在PRRSV的遗传进化树中,HP-PRRSV和之前流行的经典毒株同属于Lineage 8,其重要特征是Nsp2编码区存在30个不连续氨基酸的缺失[7-8],这种新出现的毒株具有毒力强、致死率高、传播速度快等特点,至今依然是国内流行的优势毒株[9],给我国养猪业造成非常严重的损失。
反向遗传学技术是研究RNA病毒的有力工具,该技术通过对相关病毒基因进行直接改造后构建出病毒的感染性克隆,并经RNA-launched和DNA-launched 2种途径对病毒进行拯救[10-11]。RNA-launched途径是RNA病毒基因组先反转录成cDNA,接着克隆到合适的转录载体上,通过克隆到T7或者SP6启动子的下游,先在体外转录出病毒全长的基因组RNA,随后将这些体外的转录本转染宿主细胞,在宿主细胞内包装出活的病毒粒子[12-13]。DNA-launched途径则是将经反转录的病毒cDNA克隆到真核启动子如CMV启动子的下游,并将构建好的质粒不经体外转录,直接转染宿主细胞,利用宿主细胞的核功能转录出病毒基因组,进而完成病毒粒子的包装[14]。相比RNA-launched途径,DNA-launched途径避开了体外转录步骤,不仅操作简便,还降低了转染过程中RNA降解的风险,使转染的效率大大提高。目前,反向遗传技术在研究PRRSV的结构与功能、致病机制以及研发新型疫苗等方面有着广泛应用[15-17]。
本研究运用反向遗传操作技术将HP-PRRSV JXA1株的全基因组分段克隆至改造过的低拷贝载体pOKq上,成功构建了JXA1株的全长感染性克隆。并采取基于DNA-launched途径成功获得拯救的病毒,并对拯救病毒进行生物学特性的分析。HP-PRRSV代表株JXA1株反向遗传平台的构建,为HP-PRRSV后续的结构功能探索、致病机理研究以及新型疫苗研发奠定了基础。
1 材料与方法 1.1 材料 1.1.1 载体、毒株、细胞、抗体克隆载体pJET1.2购买自Thermo Fisher Scientific。由pOK12改造具有多克隆位点的低拷贝载体pOKq、XH-GD全长感染性克隆质粒、PRRSV易感细胞Marc-145细胞、转染用BHK-21细胞均由华南农业大学兽医学院传染病教研室保存。HP-PRRSV JXA1毒株由河南农业大学田克恭教授惠赠。PRRSV N蛋白单克隆抗体购买自韩国金诺公司。FITC标记的山羊抗小鼠IgG二抗购买自北京中杉金桥生物技术有限公司。
1.1.2 主要试剂RNA抽提试剂盒购自上海飞捷生物技术有限公司,反转录试剂盒、Taq酶、高保真DNA聚合酶购自宝日医生物技术(北京)有限公司。限制性核酸内切酶购自NEB公司。T4 DNA连接酶购自Thermo Fisher Scientific。DL2000 DNA marker、250 bp DNA ladder (Dye plus)、克隆菌感受态细胞Escherichia coli DH5α、克隆载体pMD18-T vector购自宝日医生物技术(北京)有限公司;DNA凝胶回收试剂盒、质粒抽提试剂盒购自OMEGA公司。DMEM高糖培养基、胰蛋白酶购自Biological Industries。胎牛血清购自Gibco公司。二甲基亚砜(DMSO)购自Amresco公司。
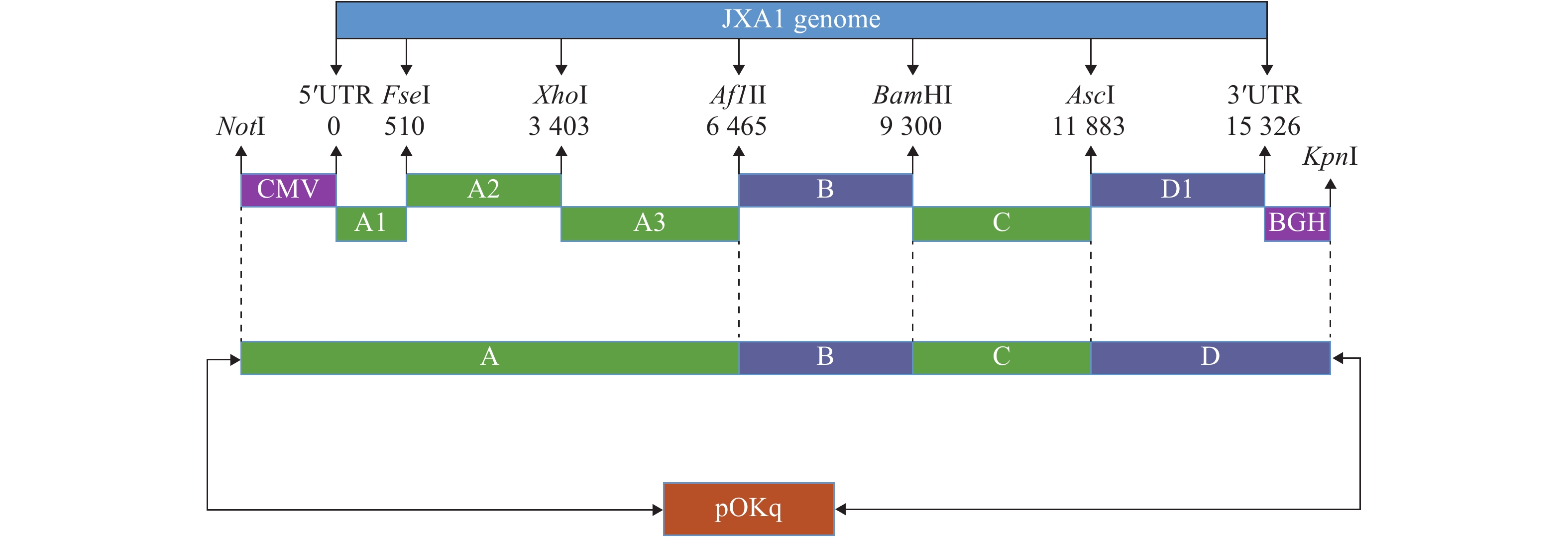
1.2 方法 1.2.1 全长cDNA构建方案与引物设计构建方案:JXA1毒株全长cDNA的构建见图1。先将CMV、A1、A2片段融合,融合产物再与A3片段连接,构成A片段。将D1片段和终止信号肽序列BGH片段融合构成D片段,然后将D、C、B、A共4个片段依次亚克隆到低拷贝载体pOKq上。
|
图 1 JXA1毒株全长cDNA克隆的构建策略 Fig. 1 The strategy for construction of the full-length cDNA clone of JXA1 strain |
根据GenBank中收录的PRRSV JXA1毒株全基因组序列,利用Oligo7.0软件 设计6对引物对病毒的全长基因进行分段克隆,获得A1、A2、A3、B、C和D1段。同时以PRRSV XH-GD全长感染性克隆质粒为模板,设计引物扩增CMV和BGH片段。同时在扩增A1片段时引入1个沉默突变,将全基因组第510位碱基由A突变为G,引入遗传标记FseI,以便与亲代病毒相区别。引物序列由上海英潍捷基生物技术有限公司合成。各段引物的序列、扩增片断大小及引物名称见表1。
|
|
表 1 构建JXA1株全长感染性克隆设计的引物序列 Table 1 Primer sequences used for construction of the full-length infectious clone of JXA1 strain |
以XH-GD全长感染性克隆质粒为模板,分别用引物(CMV-F、CMV-R)和(BGH-F、BGH-R)扩增CMV启动子序列和BGH终止信号肽序列;按上海飞捷生物技术有限公司的RNA抽提试剂盒操作说明书提取JXA1细胞病毒的总RNA,用适量RNase free水溶解,获得总RNA,用SuperScriptTM III逆转录酶和OligodT(20)(Invitrogen,USA),按照操作说明书进行cDNA合成,然后将获得的cDNA作为模板,用表1中扩增引物进行各片段的扩增,经过RT-PCR,获得A1、A2、A3、B、C和D1片段,参照DNA purification kit说明书纯化扩增的片段,利用融合PCR技术,用引物(A1-F、A2-R)融合A1和A2片段,构建点突变以引入全长感染性克隆的遗传标记FseI酶切位点,以回收的CMV片段和A1、A2融合片段为模板,用引物(CMV-F、A2-R)融合CMV、A1、A2片段并回收;以回收的BGH片段和D1片段为模板,用引物(D1-F、BGH-R)融合D1和BGH片段构成D片段并回收,PCR按常规方法进行,并设立空白对照。PCR产物用10 g/L琼脂糖凝胶(含0.5 μg/mL EB)电泳检测并回收纯化。另外,为了提高目的片段的保真性,构建感染性克隆过程中的所有PCR反应均使用宝日医生物技术(北京)有限公司的PrimeSTAR® Max高保真DNA聚合酶。
1.2.3 质粒pOK-A、pJET-B、pJET-C和pJET-D的构建将CMV、A1、A2的融合回收产物连接到pOKq空载体上形成载体pOK-CMV-A1-A2,再将扩增的A3片段连接到pOK-CMV-A1-A2载体上,构建出pOK-A中间载体。将D、C和B这3个片段的纯化产物分别与pJET1.2载体连接。取5 μL的连接产物转化克隆菌感受态细胞E. coli DH5α后,用灭菌枪头挑取可疑菌落于含氨苄青霉素的LB液体培养基中,于37 ℃条件下振荡培养4~6 h,取适量菌液进行PCR鉴定。将PCR鉴定为阳性的菌液送上海英潍捷基生物技术有限公司进行测序验证。测序正确的重组质粒分别命名为pJET-B、pJET-C和pJET-D,并按照OMEGA公司的质粒提取试剂盒说明书进行质粒抽提。
1.2.4 JXA1毒株全长cDNA的构建将pJET-D、pJET-C、pJET-B和pOK-A按照图1所示的顺序依次亚克隆到pOKq载体,构建的质粒经PCR鉴定为阳性的重组菌液送上海英潍捷基生物技术有限公司进行测序,将测序正确的克隆质粒命名为pOK-JXA1,并参照OMEGA公司去内毒素质粒提取试剂盒说明书进行去内毒素质粒抽提。
1.2.5 JXA1株病毒的拯救将JXA1株病毒全长cDNA的重组质粒pOK-JXA1转染BHK-21细胞,转染使用Lipofectamine 3000(后文简称Lipo 3000)脂质体,具体操作步骤参照说明书。转染72 h后,将6孔板反复冻融3次,收集细胞液,4 ℃、7 000 r/min离心7 min,取上清接种于单层Marc-145细胞,吸附1 h后补加含2%(φ)FBS的DMEM,置于37 ℃、CO2体积分数为5%的培养箱培养,观察细胞病变情况(Cytopathic effect,CPE),出现病变后冻融收毒,连续传12代。将拯救的病毒命名为rJXA1。
1.2.6 拯救病毒的鉴定为验证所看到的细胞病变是拯救病毒所致,排除亲本病毒污染的可能,利用引入的沉默突变FseI酶切位点进行鉴定。取第3代的病毒细胞培养悬液进行总RNA的提取,再取2 μg RNA进行反转录。以得到的cDNA为模板,以A1-F和A2-R为引物进行PCR扩增。PCR产物用10 g/L琼脂糖凝胶电泳检测并回收纯化,将纯化后的PCR产物送上海英潍捷基生物技术有限公司进行测序鉴定。
同时将拯救病毒的第3代rJXA1-P3和亲本病毒的第3代JXA1-P3接种至长满单层的Marc-145细胞6孔板中并设1孔阴性对照,置于37 ℃、CO2体积分数为5%培养箱中培养72 h后对拯救的病毒进行间接免疫荧光(Indirect immunofluoresence assay,IFA)试验鉴定。
1.2.7 拯救病毒的稳定性将拯救病毒rJXA1在Marc-145细胞上连续传至12代,观察每一代的CPE情况。同时从拯救获得的病毒中,选择第1、3、6、9和12代病毒进行TCID50测定。将Marc-145细胞接种到96孔细胞培养板上,待细胞汇合度达到90%以上时,将获得的相应代次的拯救病毒进行10倍梯度倍比稀释,即用DMEM进行10−1~10−8倍比稀释,分别接种于Marc-145细胞中,每个稀释度接种8孔,每孔100 μL。同时设未接毒的空白对照,将细胞板放置37 ℃、CO2体积分数为5%细胞培养箱作用90 min。弃去病毒液,加入含2%(φ)FBS的DMEM,放置于37 ℃、CO2体积分数为5%细胞培养箱。连续观察2~7 d,记录每个稀释度细胞病变的孔数,本试验重复3次求其平均值,并按照Reed-Muench法计算各个样品的TCID50。
1.2.8 拯救病毒的生长曲线测定rJXA1第3代病毒和亲本病毒JXA1第3代病毒在Marc-145细胞中的生长曲线。将rJXA1第3代病毒和亲本病毒JXA1第3代病毒用DMEM培养基稀释至感染复数(Multiplicity of infection,MOI)为0.1,接种于长成单层的Marc-145细胞,置于37 ℃、CO2体积分数为5%培养箱中孵育1 h,每个病毒各做3次重复。孵育结束后弃去上清,用PBS缓冲液洗涤细胞2次,加入5 mL含2%(φ)FBS的DMEM细胞维持液,置于37 ℃、CO2体积分数为5%的培养箱中培养。分别于接种病毒12、24、36、48、60、72、84和96 h后,从每瓶细胞中吸取200 μL上清,然后测定各时相上清中病毒的TCID50,绘制病毒的生长曲线。
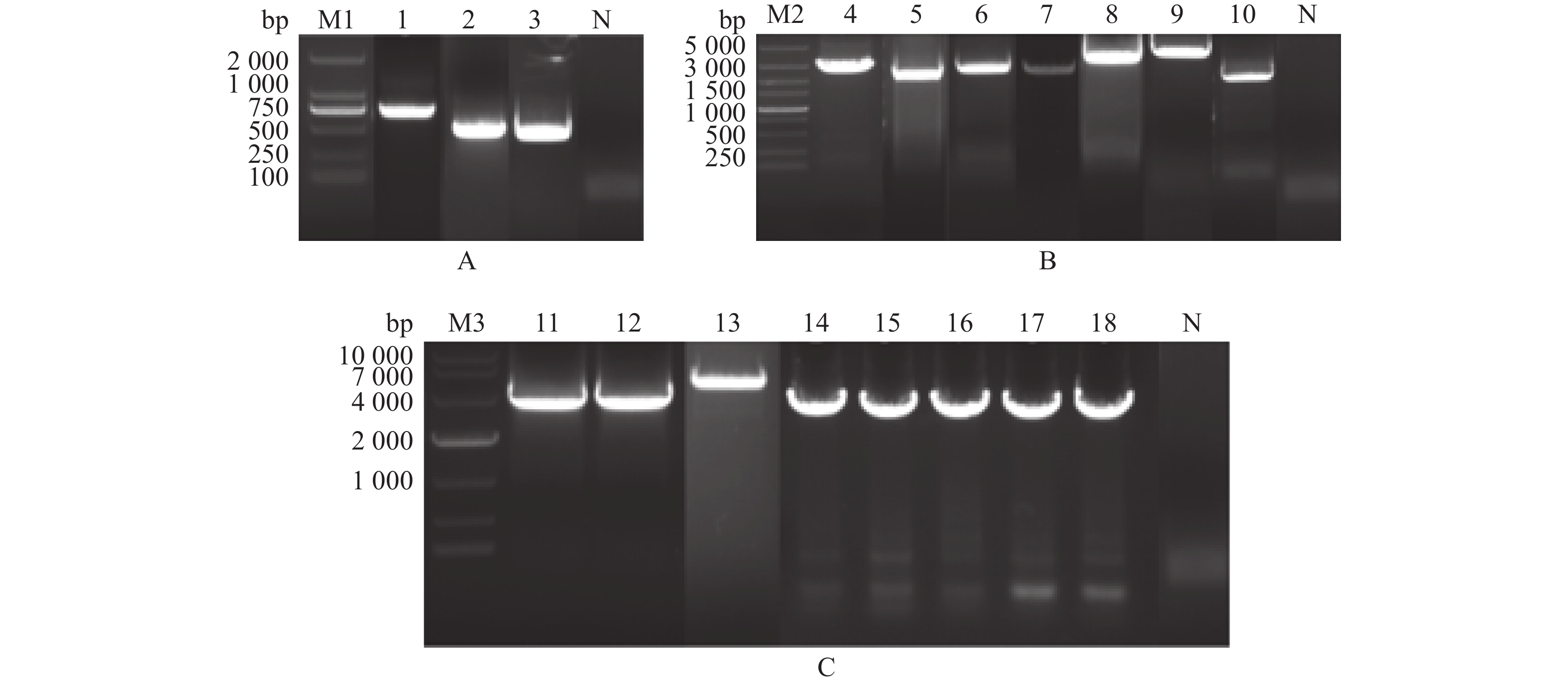
2 结果与分析 2.1 JXA1毒株全基因组各片段的扩增以XH-GD全长感染性克隆质粒为模板扩增CMV启动子序列和BGH终止信号肽序列,以JXA1反转录后的cDNA为模板扩增JAX1的A1、A2、A3、B、C和D1段。PCR产物经琼脂糖凝胶电泳后,可见相应的目的条带,其中CMV、BGH和A1片段的扩增产物大小分别约为750、500和500 bp,与预期大小相符(图2A)。A2、A3、B、C和D1片段的扩增产物大小分别约为2 200、3 000、3 000、2 700和3 300 bp,与预期大小相符(图2B)。
|
图 2 JXA1株各基因片段的PCR扩增与构建 Fig. 2 PCR amplification and construction of each gene fragment of JXA1 strain M1:DL2000 DNA marker;M2:DL5000 DNA marker;M3:DL10000 DNA marker;1:CMV片段;2:BGH片段;3:A1片段;4:B片段;5:C片段;6:A3片段;7:A3片段酶切产物;8:D1片段;9:D1和BGH融合片段;10:A2片段;11~12:CMV、A1和A2融合片段;13:pOK-CMV-A1-A2的酶切产物;14~18:菌液PCR鉴定;N:阴性对照 M1: DL2000 DNA marker; M2: DL5000 DNA marker;M3:DL10000 DNA marker; 1: CMV fragment; 2: BGH fragment; 3 A1 fragment; 4: B fragment; 5: C fragment; 6: A3 fragment; 7: Enzyme digestion of A3 fragment; 8: D1 fragment; 9: D1 and BGH fusion fragment; 10: A2 fragment; 11−12: CMV, A1 and A2 fusion fragment; 13: Enzyme digestion of pOK-CMV-A1-A2; 14−18: PCR identification of the bacterial liquid; N: Negative control |
利用融合PCR技术融合A1和A2片段,构建点突变来引入全长感染性克隆的遗传标记FseI酶切位点,以回收的CMV片段和A1、A2融合片段为模板,对CMV、A1、A2片段进行融合PCR扩增并回收,PCR产物经琼脂糖凝胶电泳后,可见约为3 800 bp扩增片段,与预期大小相符(图2C)。将CMV、A1、A2的融合回收产物连接到pOKq空载体上形成载体pOK-CMV-A1-A2,再将扩增的A3片段连接到pOK-CMV-A1-A2载体上,并进行菌液PCR鉴定(图2C),构建出pOK-A中间载体;以回收的BGH和D1段为模板,对D1和BGH片段进行融合PCR扩增并回收,PCR产物经琼脂糖凝胶电泳后,可见约为3 800 bp扩增片段,与预期大小相符(图2B),然后将D、C、B 片段依次克隆到pJET1.2载体上。
2.2 JXA1毒株全长cDNA的构建与鉴定将pJET-D、pJET-C、pJET-B和pOK-A按照图1所示的顺序依次连接到pOKq载体,构建全长cDNA的重组质粒pOK-JXA1,用引物CMV-F、A1-R对重组质粒pOK-JXA1进行PCR鉴定,PCR产物经琼脂糖凝胶电泳后,可见约为1 300 bp的扩增条带,与预期大小相符(图3),鉴定阳性的重组菌液送往上海英潍捷基生物技术有限公司进行测序验证,测序正确的重组质粒命名为pOK-JXA1。
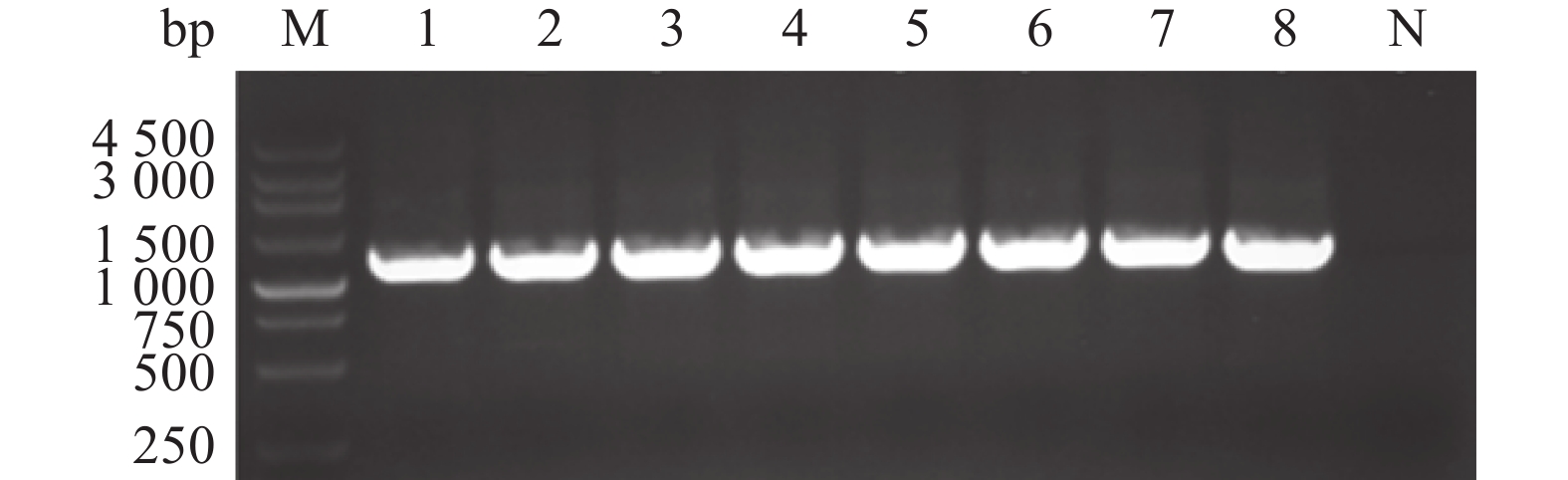
|
图 3 pOK-JXA1菌液PCR鉴定 Fig. 3 PCR identification of pOK-JXA1 bacterial liquid M:250 bp DNA ladder (Dye plus);1~8:pOK-JXA1菌液PCR扩增产物;N:阴性对照 M: 250 bp DNA ladder (Dye plus); 1−8: PCR amplification of pOK-JXA1 bacterial liquid; N: Negative control |
将质粒pOK-JXA1转染BHK-21细胞72 h后,冻融收获病毒,离心取上清后接种至Marc-145细胞,置于37 ℃、CO2体积分数为5%的培养箱中培养72 h,显微镜下可见与亲本病毒JXA1类似的CPE(图4),将拯救病毒命名为rJXA1。
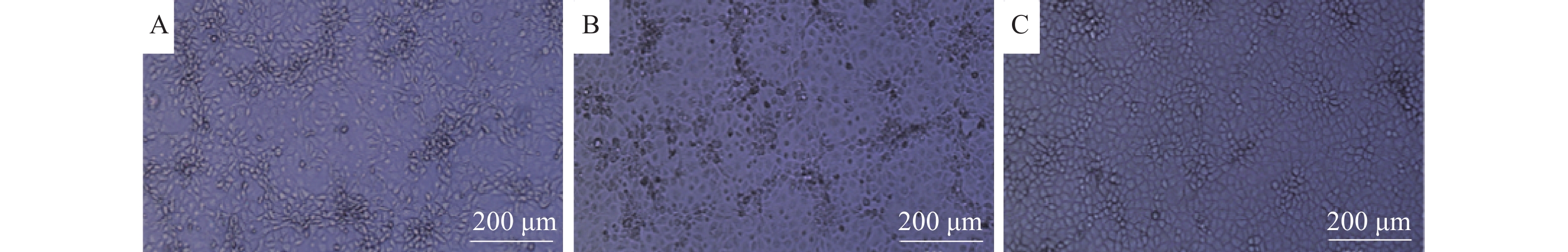
|
图 4 拯救病毒产生的细胞病变 Fig. 4 Cytopathic effect (CPE) produced in the rescued viruses A:拯救病毒rJXA1产生的CPE;B:亲本病毒JXA1产生的CPE;C:对照细胞 A: CPE produced in the rescued virus rJXA1; B: CPE produced in the parental virus JXA1; C: Control cell |
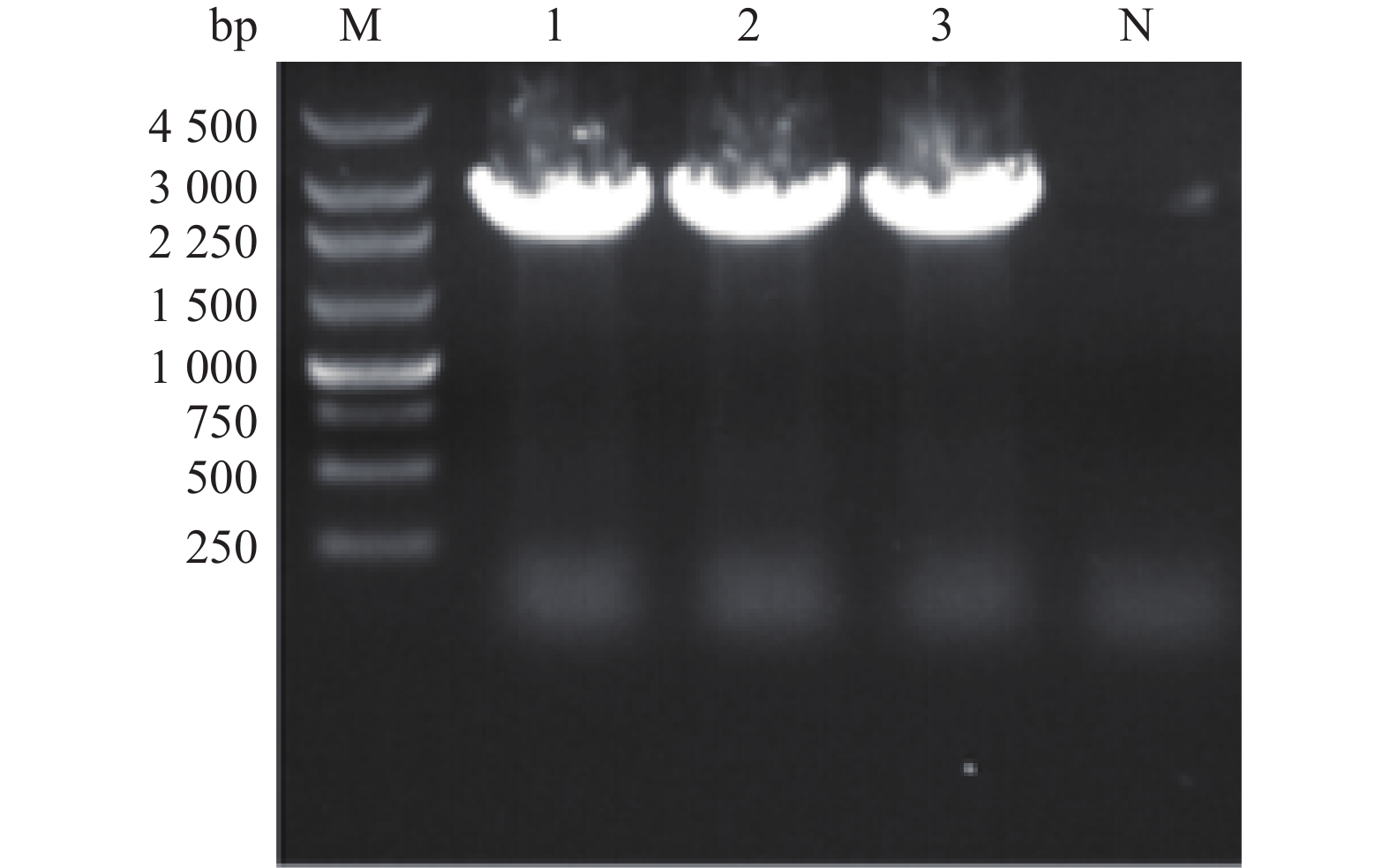
将拯救获得的rJXA1在Marc-145细胞中连续传代。取第3代病毒(rJXA1-P3)的培养液抽提RNA进行RT-PCR鉴定,PCR产物经琼脂糖凝胶电泳后可检测到强阳性条带,大小与预期相符(图5)。将纯化的PCR产物送测序,测序结果显示拯救病毒rJXA1全基因组的第510位核苷酸由A突变为G,对应的阅读框由AGA变为AGG,推导的氨基酸均为精氨酸,产生了新的酶切位点FseI(GGCCGGCC),与预期结果一致,表明拯救病毒的遗传标记被成功引入。
|
图 5 拯救病毒的遗传标记位点RT-PCR鉴定 Fig. 5 Identification of genetic marker of the rescued virus by RT-PCR M:250 bp DNA ladder (Dye plus);1~3:rJXA1-P3的3个重复的扩增产物;N:阴性对照 M: 250 bp DNA ladder (Dye plus); 1−3: Amplification products of three replicates of rJXA1-P3; N: Negative control |
用PRRSV N蛋白单克隆抗体对亲本病毒以及拯救病毒进行间接免疫荧光鉴定,结果显示:接种亲本病毒和拯救病毒的细胞在荧光显微镜下可观察到特异性的绿色荧光,而阴性对照细胞没有荧光产生(图6),证明病毒拯救成功。

|
图 6 拯救病毒的间接免疫荧光鉴定 Fig. 6 Identification of the rescued virus through indirect immunofluoresence assay A:亲本病毒;B:拯救病毒;C:空白对照 A: Parental virus; B: Rescued virus; C:Blank control |
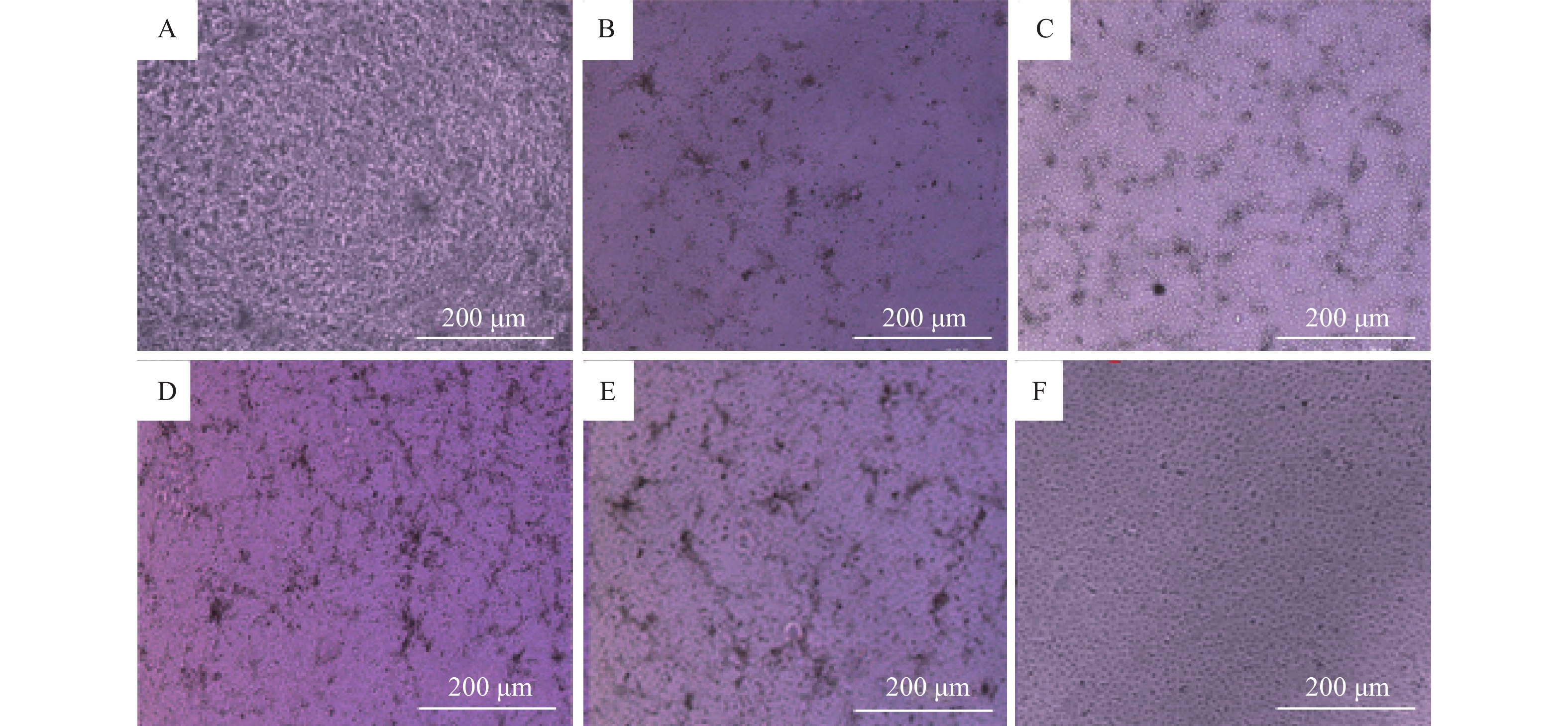
将拯救病毒rJXA1在Marc-145细胞上连续传至12代,每一代均可以产生明显CPE。图7为第1、3、6、9和12代的细胞病变情况。同时对第1、3、6、9和12代病毒进行TCID50的测定,结果表明,拯救病毒可在Marc-145细胞上稳定传代,病毒滴度介于106.0~107.3 TCID50/mL。
|
图 7 rJXA1在Marc-145细胞中引起的细胞病变 Fig. 7 Cytopathic effect (CPE) induced by rJXA1 in Marc-145 cells A:第1代;B:第3代;C:第6代;D:第9代;E:第12代;F:空白对照 A: The first generation; B: The third generation; C: The sixth generation; D: The ninth generation; E: The 12th generation; F: Blank control |
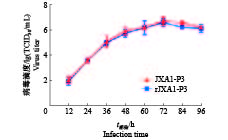
对拯救病毒rJXA1的第3代病毒和亲本病毒JXA1的第3代病毒进行生长曲线的测定,结果表明拯救病毒rJXA1第3代和亲本病毒JXA1第3代的生长曲线相似,二者达到最高滴度的时间相同,均为72 h(图8)。
|
图 8 拯救病毒和亲本病毒的生长曲线 Fig. 8 The growth curves of the rescued virus and parental virus. |
2006年以来,以感染猪高热、高发病率和高死亡率为特征的高致病性猪繁殖与呼吸综合征在我国爆发,并在全国范围流行。截至目前,该病依然在国内广泛流行,给我国养猪业带来巨大的经济损失[18]。
PRRSV基因组为不分节段的单股正链RNA,全长15 kb左右。构建其全长感染性克隆时需要避免基因突变、缺失等破坏保真性的情况出现。为克服这一难点,本研究在构建过程中全程使用PrimeSTAR® Max高保真聚合酶进行基因的扩增或融合PCR扩增。此外,为了避免长片段扩增导致的保真性降低,本研究还将整个病毒的基因组分为4个节段分段克隆,逐步连接到严谨性较高的低拷贝载体pOKq上,成功构建了JXA1株的全长感染性克隆。并采取基于DNA-launched途径进行病毒拯救,成功构建了HP-PRRSV JXA1株的反向遗传平台,获得了感染性拯救病毒rJXA1。该拯救病毒与亲本毒株JXA1具有相同的体外增殖能力。
为了区分亲本病毒和拯救病毒,需要在拯救病毒的基因中加入遗传标记。本研究选择了20株有代表性的基因II型PRRSV毒株,使用MegAlign软件对其进行全基因组序列比对。最终选择第510位碱基作为遗传标记位点。20株基因II型代表株在该位点的碱基均为A,对应的氨基酸密码子为AGA,编码精氨酸,将其突变为G后不影响编码的氨基酸,同时可以形成一个新的单酶切位点FseI(GGCCGGCC),便于对拯救病毒和亲本病毒的鉴定。
准确且完整的全长感染性克隆的构建是病毒拯救的关键[19]。本研究采取基于DNA-launched途径进行病毒拯救,避开了RNA体外转录的步骤,不仅减少了试验操作的影响,也简化了整个试验流程。另外,本研究还在JXA1病毒的基因组两端添加了核酶序列,全长质粒转染至BHK-21细胞后在细胞内转录出RNA,在核酶的作用下直接剪切,形成不含外源基因的病毒基因组。PRRSV基因组3′端存在1个ploy A尾,ploy A的个数对病毒转录的稳定性有重要作用,如果ploy A尾上A的个数过少,有可能导致转录失败,无法产生具有感染性的病毒粒子。基于广东省动物源性人兽共患病预防与控制重点实验室以前建立RNA-launched反向遗传平台时的经验,本研究选择了42个A构建病毒基因组的ploy A尾。
虽然国内已使用高致病性PRRSV弱毒活疫苗来控制HP-PRRSV的流行,但HP-PRRSV毒株依然在国内广泛流行,未呈现明显的减弱趋势[20],其他亚群(如NADC30-like亚群和GM2-like亚群)呈现上升趋势[21-22],所以很有必要对其原因进行研究。本研究成功构建的HP-PRRSV JXA1株的反向遗传平台将为进一步研究HP-PRRSV的致病机理、基因功能以及新型疫苗研发奠定了基础,同时可为PRRSV的防控提供技术参考。
致谢:感谢河南农业大学田克恭教授惠赠HP-PRRSV JXA1毒株,感谢华南农业大学兽医学院陈耀博士在本研究的选题上给予的指导与建议!
| [1] |
NEUMANN E J, KLIEBENSTEIN J B, JOHNSON C D, et al. Assessment of the economic impact of porcine reproductive and respiratory syndrome on swine production in the United States[J]. J Am Vet Med Assoc, 2005, 227(3): 385-392. DOI:10.2460/javma.2005.227.385 (  0) 0) |
| [2] |
LI B, LIU R F, LIU S Y, et al. The genomic diversity of Chinese porcine reproductive and respiratory syndrome virus isolates from 1996 to 2009[J]. Vet Microbiol, 2010, 146(3/4): 226-237. (  0) 0) |
| [3] |
童光志, 周艳君, 郝晓芳, 等. 高致病性猪繁殖与呼吸综合征病毒的分离鉴定及其分子流行病学分析[J]. 中国预防兽医学报, 2007, 25(5): 323-327. DOI:10.3969/j.issn.1008-0589.2007.05.001 (  0) 0) |
| [4] |
ALBINA E. Epidemiology of porcine reproductive and respiratory syndrome (PRRS): An overview[J]. Vet Microbiol, 1997, 55(1/2/3/4): 309-316. (  0) 0) |
| [5] |
TIAN K G, YU X L, ZHAO T Z, et al. Emergence of fatal PRRSV variants: Unparalleled outbreaks of atypical PRRS in China and molecular dissection of the unique hallmark[J]. PLoS One, 2007, 2(6): e526. DOI:10.1371/journal.pone.0000526 (  0) 0) |
| [6] |
ZHAO K, YE C, CHANG X B, et al. Importation and recombination are responsible for the latest emergence of highly pathogenic porcine reproductive and respiratory syndrome virus in China[J]. J Virol, 2015, 89(20): 10712-10716. DOI:10.1128/JVI.01446-15 (  0) 0) |
| [7] |
Li Y F, WANG X L, BO K T, et al. Emergence of a highly pathogenic porcine reproductive and respiratory syndrome virus in the mid-eastern region of China[J]. Vet J, 2007, 174(3): 577-584. DOI:10.1016/j.tvjl.2007.07.032 (  0) 0) |
| [8] |
ZHOU Z, LI X D, LIU Q, et al. Complete genome sequence of two novel Chinese virulent porcine reproductive and respiratory syndrome virus variants[J]. J Virol, 2012, 86(11): 6373-6374. DOI:10.1128/JVI.00663-12 (  0) 0) |
| [9] |
ZHAO K, GAO J C, XIONG J Y, et al. Two residues in NSP9 contribute to the enhanced replication and pathogenicity of highly pathogenic porcine reproductive and respiratory syndrome virus[J]. J Virol, 2018, 92(7): e02209-17. (  0) 0) |
| [10] |
TRUONG H M, LU Z, KUTISH G F, et al. A highly pathogenic porcine reproductive and respiratory syndrome virus generated from an infectious cDNA clone retains the in vivo virulence and transmissibility properties of the parental virus[J]. Virology, 2004, 325(2): 308-319. DOI:10.1016/j.virol.2004.04.046 (  0) 0) |
| [11] |
吴欣伟, 熊永忠, 秦宏阳, 等. 猪繁殖与呼吸综合征病毒XH株感染性克隆的构建[J]. 中国预防兽医学报, 2011, 33(2): 97-100. DOI:10.3969/j.issn.1008-0589.2011.02.04 (  0) 0) |
| [12] |
HUANG Y W, FANG Y, MENG X J. Identification and characterization of a porcine monocytic cell line supporting porcine reproductive and respiratory syndrome virus (PRRSV) replication and progeny virion production by using an improved DNA-launched PRRSV reverse genetics system[J]. Virus Res, 2009, 145(1): 1-8. DOI:10.1016/j.virusres.2009.05.013 (  0) 0) |
| [13] |
常亚飞, 刘永刚, 李爱东, 等. 高致病性猪繁殖与呼吸综合征病毒HuN4传代致弱株HuN4-F30感染性克隆的构建及拯救病毒致病性分析[J]. 中国预防兽医学报, 2016, 38(7): 532-536. (  0) 0) |
| [14] |
LEE C, CALVERT J G, WELCH S K, et al. A DNA-launched reverse genetics system for porcine reproductive and respiratory syndrome virus reveals that homodimerization of the nucleocapsid protein is essential for virus infectivity[J]. Virology, 2005, 331(1): 47-62. DOI:10.1016/j.virol.2004.10.026 (  0) 0) |
| [15] |
RENUKARADHYA G J, MENG X J, CALVERT J G, et al. Live porcine reproductive and respiratory syndrome virus vaccines: Current status and future direction[J]. Vaccine, 2015, 33(33): 4069-4080. DOI:10.1016/j.vaccine.2015.06.092 (  0) 0) |
| [16] |
SUHARDIMAN M, KRAMYU J, NARKPUK J, et al. Generation of porcine reproductive and respiratory syndrome virus by in vitro assembly of viral genomic cDNA fragments[J]. Virus Res, 2015, 195: 1-8. DOI:10.1016/j.virusres.2014.09.008 (  0) 0) |
| [17] |
翟新验, 韩焘, 周智. 反向遗传学在猪繁殖与呼吸综合征病毒研究中的应用[J]. 中国兽医杂志, 2014, 50(4): 52-53. DOI:10.3969/j.issn.0529-6005.2014.04.021 (  0) 0) |
| [18] |
ZHOU L, KANG R M, JI G S, et al. Molecular characterization and recombination analysis of porcine reproductive and respiratory syndrome virus emerged in southwestern China during 2012-2016[J]. Virus Genes, 2018, 54(1): 98-110. DOI:10.1007/s11262-017-1519-y (  0) 0) |
| [19] |
LV J, ZHANG J W, SUN Z, et al. An infectious cDNA clone of a highly pathogenic porcine reproductive and respiratory syndrome virus variant associated with porcine high fever syndrome[J]. J Gen Virol, 2008, 89(Pt 9): 2075-2079. (  0) 0) |
| [20] |
韩焘, 张硕, 吴佳俊, 等. 高致病性猪繁殖与呼吸综合征病毒疫苗株JXA1-R感染性克隆的构建与鉴定[J]. 中国兽医学报, 2016, 36(11): 1818-1822. (  0) 0) |
| [21] |
WANG L J, XIE W T, CHEN X X, et al. Molecular epidemiology of porcine reproductive and respiratory syndrome virus in central China since 2014: The prevalence of NADC30-like PRRSVs[J]. Microb Pathog, 2017, 109: 20-28. DOI:10.1016/j.micpath.2017.05.021 (  0) 0) |
| [22] |
ZHOU L, YANG B N, XU L, et al. Efficacy evaluation of three modified-live virus vaccines against a strain of porcine reproductive and respiratory syndrome virus NADC30-like[J]. Vet Microbiol, 2017, 207: 108-116. DOI:10.1016/j.vetmic.2017.05.031 (  0) 0) |
 2020, Vol. 41
2020, Vol. 41


