2. 华南农业大学 兽医学院,广东 广州 510642
2. College of Veterinary Medicine, South China Agricultural University, Guangzhou 510642, China
狂犬病是由狂犬病病毒(Rabies virus,RABV)引起的一种重要的高度嗜神经性人兽共患传染病,人类和所有温血动物均易感,感染后一旦出现临床症状,致死率几乎为100%[1]。目前尚无有效的治疗方法,主要依赖接种疫苗进行免疫预防。
狂犬病病毒属于单股负链RNA病毒目弹状病毒科狂犬病病毒属,基因组顺序高度保守,为3′-N-P-M-G-L-5′。狂犬病病毒基因组只编码5种结构蛋白:核蛋白(Nucleoprotein,N蛋白)、磷酸化蛋白(Phosphoprotein,P蛋白)、基质蛋白(Matrix protein,M蛋白)、糖蛋白(Glycoprotein,G蛋白)和RNA依赖的RNA聚合酶(RNA-dependent RNA polymerase,L蛋白),其中N、P、L蛋白与狂犬病病毒RNA共同组成核糖核蛋白体,核糖核蛋白体是狂犬病病毒转录和复制的核心成分[2]。基因重排是改变基因组中的基因顺序,从而改变病毒基因的表达和病毒表型。基因重排首先应用于水泡性口炎病毒(Vesicular stomatitis virus,VSV)的研究,该技术成功改变了VSV的表型[3-5]。陈凯云等[6]和文兆海等[7]利用基因重排技术将狂犬病病毒SRV9 G基因重排至基因顺序第2位,并对基因重排后病毒的遗传稳定性和免疫效果进行了评估。Mei等[8-9]对狂犬病病毒Flury高鸡胚传代株(High egg passage-Flury,HEP-Flury)的N和P基因进行了重排,并对基因重排后病毒在小鼠神经母细胞瘤(Neuroblastoma,NA)细胞和小鼠上的生物学特性进行了评估,探讨了基因重排与病毒表型的相关性。
狂犬病病毒基质蛋白M,是狂犬病病毒最小的结构蛋白,主要参与病毒粒子的装配和出芽,调节病毒的转录和复制平衡[10-12]。M蛋白被认为是决定狂犬病病毒致病性的一个重要因素[12-13]。在前期研究中,Yang等[14]成功拯救了M基因位于第2位和第4位的病毒,并发现这2个病毒在幼仓鼠肾细胞的传代细胞(Clone of baby hamster kidney-21 cell,BSR)上的生长劣于亲本毒株rHEP-Flury。由于狂犬病病毒为嗜神经病毒[15],本研究拟进一步对狂犬病病毒亲本毒株rHEP-Flury和M基因重排病毒在NA细胞上的基因转录、表达、生长和扩散进行评估和比较,进而探讨M基因重排对狂犬病病毒在NA细胞上的生物学特性的影响。
1 材料与方法 1.1 病毒和细胞狂犬病病毒亲本毒株rHEP-Flury基因结构顺序为3′-N-P-M-G-L-5′,由华南农业大学兽医学院郭霄峰教授课题组拯救保存。M基因位于第2位的基因重排病毒毒株M2(3′-N-M-P-G-L-5′)和M基因位于第4位的基因重排病毒毒株M4(3′-N-P-G-M-L-5′)由Yang等[14]前期通过反向遗传操作系统拯救获得。NA细胞购自武汉生物制品所基因工程室。
1.2 基因重排病毒的RT-PCR鉴定参照试剂盒说明书,利用Hipure universal RNA试剂盒(Magen)提取病毒RNA,然后利用HiScript®Ⅱ1st strand cDNA synthesis试剂盒(Vazyme)反转录酶进行cDNA第1链的合成,反转录引物为随机引物,最后利用3对引物D-F和P-R、D-F和N-R、D-F和M-R(表1)同时进行PCR扩增鉴定。根据PCR扩增产物大小区分亲本毒株rHEP-Flury和M基因重排病毒M2和M4,同时设置无菌双蒸水为阴性对照。回收PCR产物,测序并进一步验证。
|
|
表 1 用于M基因重排病毒基因顺序鉴定的引物 Table 1 Primers used for gene order verification of M gene rearranged virus |
为了检测M基因重排对病毒结构基因转录的影响,本研究将亲本毒株rHEP-Flury和基因重排病毒M2、M4分别感染NA细胞,感染指数(Multiplicity of infection,MOI)设为3.00,以确保每个细胞都能感染病毒。34 ℃条件下孵育12 h后,用PBS缓冲液将细胞漂洗1遍后收集细胞。利用HiPure Universal RNA试剂盒(Magen)提取细胞总RNA,参照Mei[8]前期建立的荧光定量PCR对感染细胞中的病毒Leader RNA(LeRNA)和N、P、M、G、L基因 mRNA的转录水平以及基因组RNA(vRNA)的复制水平进行半定量PCR,以β-actin作为内参基因。同时设置空白细胞对照组和病毒感染平行组,利用FITC标记的抗狂犬病病毒的免疫球蛋白(Fujirebio)进行标记,以确认每个细胞都感染了病毒。
1.4 Western blot试验检测病毒蛋白的表达将亲本毒株rHEP-Flury和M基因重排病毒M2、M4分别感染NA细胞,MOI=3.00,同时设未接毒的细胞作为空白对照。感染12 h后取出细胞板,每孔加入1 mL 4 ℃预冷的PBS缓冲液洗涤细胞1次,然后加入100 μL RIPA蛋白裂解液(1 mL RIPA蛋白裂解液加10 μL PMSF),混匀后置于冰上裂解30 min。12 000 r/min离心20 min,收集上清,冻存于−80 ℃冰箱。经Pierce BCA protein assay试剂盒进行蛋白定量后,调节成相同浓度,每份样品取15 μL进行100 g/L SDS聚丙烯酰胺凝胶电泳。电泳后将蛋白质转印到聚偏氟乙烯(PVDF)膜上,用5%(w)脱脂奶粉封闭1 h,然后用0.1%(w)的PBST缓冲液洗涤2次,最后分别加入抗狂犬病病毒N(按体积比1∶1 000稀释,浙江同点生物科技有限公司)、P(按体积比1∶500稀释,华南农业大学兽医学院郭霄峰课题组制备)、M(按体积比1:100稀释,华南农业大学兽医学院郭霄峰课题组制备)和G(按体积比1∶500稀释,华南农业大学兽医学院郭霄峰课题组制备)蛋白的单克隆抗体,4 ℃孵育过夜。设置β-actin(按体积比1∶1 000稀释,上海碧云天生物技术有限公司)为参考蛋白。第2天取出PVDF膜用0.1%(w)的PBST缓冲液洗涤4次,然后加入辣根过氧化物酶标记的二抗(体积比1∶50 000,Bioworld),37 ℃条件下孵育2 h。孵育后用0.1%(w)的PBST缓冲液洗涤4次,再用PBS缓冲液漂洗1次,然后进行曝光,拍照,最后利用软件Image-Pro Plus 6.0进行分析。
1.5 病毒在神经母细胞瘤细胞上的一步和多步生长曲线为了解亲本毒株rHEP-Flury和M基因重排病毒M2、M4在神经细胞上的生长动力学差异,本研究测定了它们在NA细胞上的一步(MOI=3.00)和多步(MOI=0.01)生长曲线。于第1天接种NA细胞至直径3.5 cm的细胞培养板中,每板8×105个细胞。第2天先将细胞用无血清的RPMI1640培养基洗1遍,然后分别以MOI=0.01和MOI=3.00接种病毒,37 ℃条件下孵育1 h后,弃上清,用RPMI1640培养基洗3遍,然后加入5 mL含5% (φ) FBS的RPMI1640,置于34 ℃、5% (φ) CO2环境中培养。分别于12、24、48、72和96 h后收集120 μL细胞培养上清液进行滴度测定[16],每个时间点的病毒滴度测2次取平均值,共做3次重复。
1.6 病毒在神经母细胞瘤细胞上的扩散慢生长和快扩散有利于狂犬病病毒逃避宿主免疫系统,增强病毒的致病性[17-18],因此本研究对病毒在NA细胞上的生长和扩散进行了评估。参照文献[19],于第1天在12孔板上接种细胞,每孔接种2.5×105个NA细胞,第2天以MOI=0.005分别接种M2、M4和rHEP-Flury。接种完将细胞板置于入34 ℃、5% (φ) CO2环境中培养,分别于12、24、36、48、60和72 h后取出,弃上清,用PBS缓冲液洗涤3遍。经80% (φ)预冷丙酮溶液固定后,用FITC标记的抗狂犬病病毒的免疫球蛋白进行免疫荧光染色,在荧光显微镜下观察和拍照,每孔分别选3个视野进行拍照。
2 结果与分析 2.1 基因顺序的确定利用3对引物对亲本毒株rHEP-Flury和重排病毒M2、M4的基因顺序进行确认,结果显示:利用引物D-F和P-R对rHEP-Flury、M2和M4进行扩增的产物大小分别为1 570 、2 380和1 570 bp(图1,泳道4、7、10);利用引物D-F和N-R对3种病毒进行扩增的产物大小均为677 bp(图1,泳道5、8、11);利用引物D-F和M-R对3种病毒进行扩增的产物大小分别为2 537、1 541和4 205 bp(图1,泳道6、9、12)。这些条带与预期大小一致,进一步测序比对确定为目的条带,说明病毒的基因顺序没有改变。
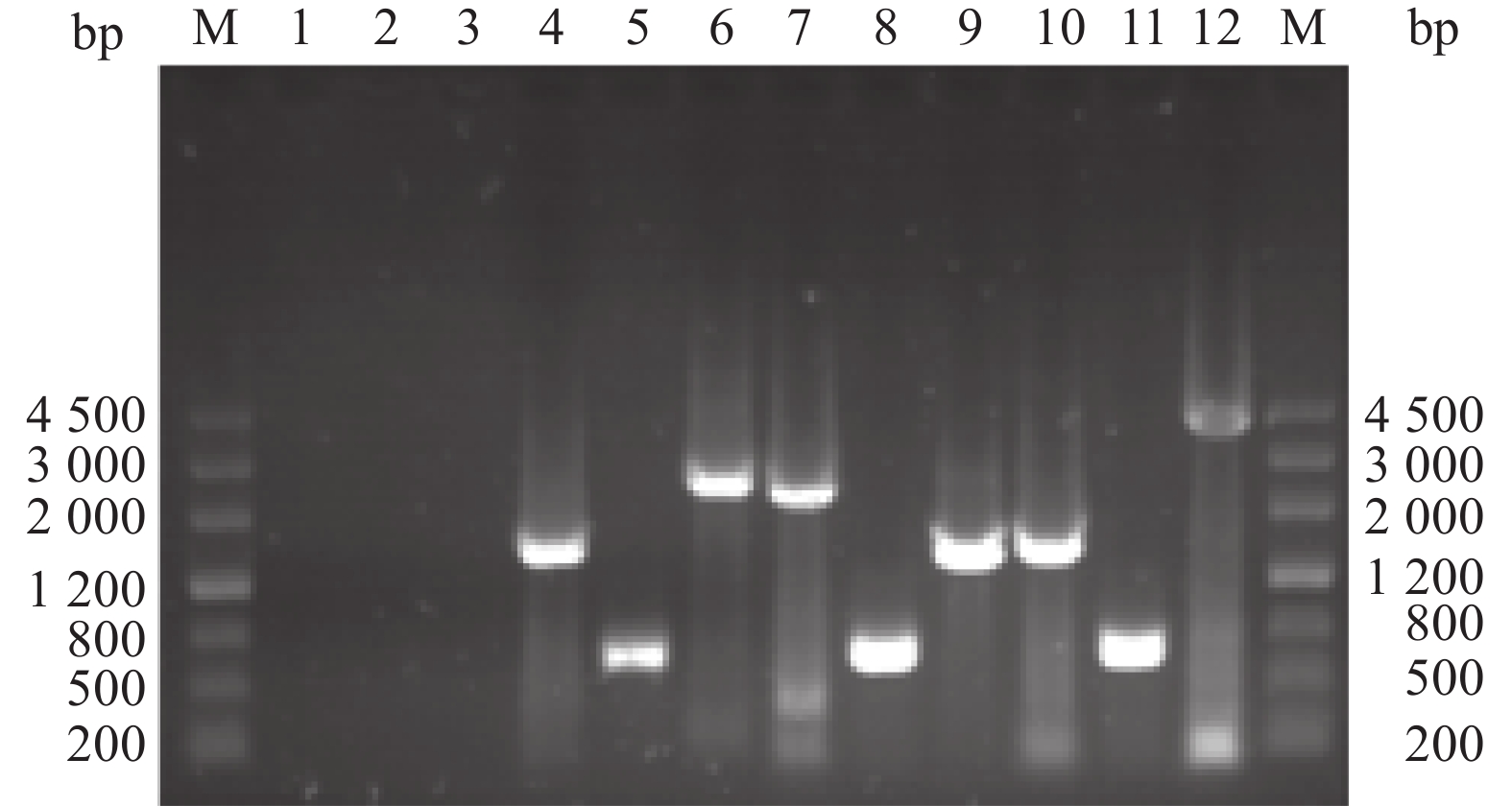
|
图 1 病毒基因顺序的鉴定 Fig. 1 Determination of gene order in virus genome M:DNA marker;泳道1、2、3:无菌双蒸水;泳道4、5、6:亲本毒株rHRP-Flury;泳道7、8、9:M基因重排病毒M2;泳道10、11、12:M基因重排病毒M4 M: DNA marker; Lane 1, 2, 3: Sterile double distilled water; Lane 4, 5, 6: Parent strain rHEP-Flury; Lane 7, 8, 9: M gene rearranged virus M2; Lane 10, 11, 12: M gene rearranged virus M4 |
将rHEP-Flury和M2、M4感染NA细胞后,对细胞内病毒基因合成转录和蛋白表达水平进行测定,以β-actin作为内参基因,利用软件GraphPad Prism 6.0进行作图和生物学统计分析。检测结果显示:病毒感染细胞12 h后,亲本毒株rHEP-Flury的病毒vRNA的合成水平和N、P、M、G mRNA的转录水平显著高于M2和M4。病毒N、P、M、G和L mRNA的转录水平与vRNA的合成水平变化趋势一致,均为rHEP-Flury > M4 > M2 ( 图2A)。进一步的相关性分析表明N、P、M、G和LmRNA的转录水平与病毒vRNA或LeRNA具有显著相关性(r>0.9,P<0.01),说明病毒结构基因的转录与病毒基因组vRNA的合成呈正相关。
|
图 2 不同病毒感染的神经母细胞瘤细胞中病毒基因的转录和表达 Fig. 2 Transcription and expression of virus gene in neuroblastoma cells infected by different viruses “**”和“****”分别表示在0.01和0.000 1水平差异显著(双因素方差分析法) “**” and “****” indicated significant differences at 0.01 and 0.000 1 levels respectively (Two-way anova method) |
为了进一步确定结构基因位置对结构基因mRNA转录的影响,本研究对各结构基因mRNA在一次完整的转录过程中的转录比例进行了分析。mRNA转录比例=该基因mRNA转录子数/(所有结构基因mRNA转录子总数+LeRNA数量)。结果显示,M基因位置距离基因组3′端越远,M mRNA的转录比例越低,即M2>rHEP-Flury>M4。M4的G基因被动向前移动一位,转录比例提高;M2的P基因被动向后移一位,转录比例降低。此外,这3个病毒的N基因和L基因,虽然基因位置相同,但是转录比例不完全一致,M基因重排病毒的L mRNA和LeRNA的转录比例均高于rHEP-Flury(图2B)。因此推测基因位置影响病毒结构基因的转录比例,但是还存在其他影响病毒结构基因转录的因素。
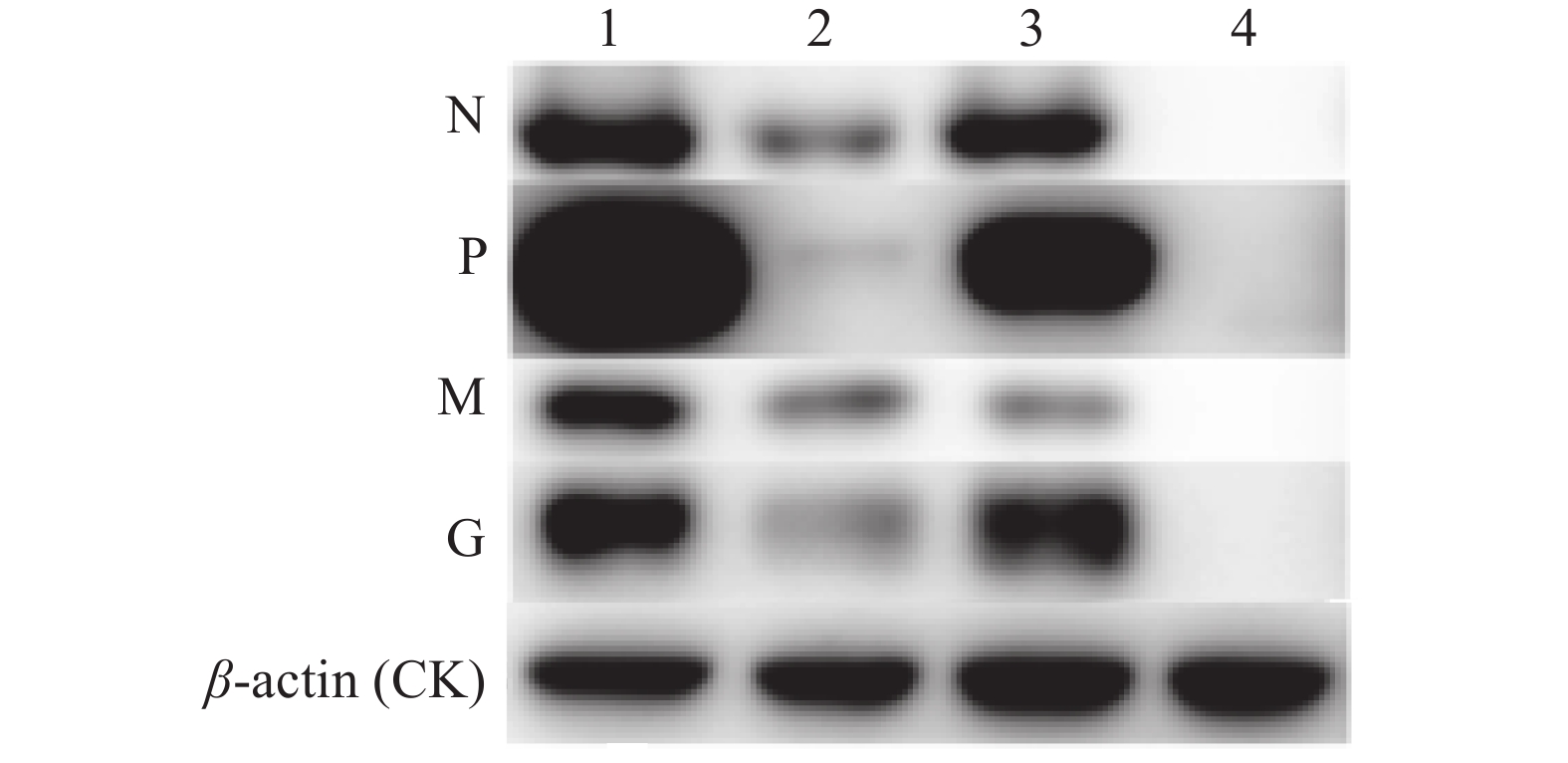
利用Western blot对病毒感染的NA细胞中的N、P、M和G蛋白表达的检测结果显示:病毒感染细胞12 h后,M2感染的NA细胞中的N、P、M和G蛋白表达水平明显低于亲本毒株,M4感染的NA细胞中的M蛋白的表达水平明显低于亲本毒株(图3)。以β-actin作为参考蛋白对细胞中的N、P、M和G蛋白进行半定量,结果显示:N、P、M和G这4种结构蛋白的表达水平与mRNA的表达水平基本一致(图4),说明病毒结构蛋白的表达受到结构基因mRNA的转录水平调节。
|
图 3 不同病毒感染的神经母细胞瘤细胞细胞中的蛋白表达 Fig. 3 Protein expression in neuroblastoma cells infected by different viruses 1:rHEP-Flury感染;2:M2感染;3:M4感染;4:无病毒感染 1: rHEP-Flury infection; 2: M2 infection; 3:M4 infection; 4: No virus infection |
|
图 4 不同病毒感染的神经母细胞瘤细胞中的蛋白表达 Fig. 4 Protein expression in neuroblastoma cells infected by different viruses |
当MOI=0.01时,病毒在NA细胞中的多步生长曲线显示:rHEP-Flury、M2和M4在NA细胞上的生长没有显著差别,rHEP-Flury的生长和最大滴度优于M2和M4;感染48 h前,M4在NA细胞中的生长速度快于M2,48 h后,M2的生长速度快于M4,且M2最大滴度高于M4(图5A)。当MOI=3.00时,病毒在NA细胞中的一步生长曲线显示:rHEP-Flury、M2和M4在NA细胞上的一步生长曲线没有明显规律,都是在感染48 h左右达到最高峰(图5B)。进一步对MOI=0.01和MOI=3.00时病毒的最大滴度进行比较,结果显示:当MOI=0.01时,M2、rHEP-Flury和M4在NA细胞上的生长都优于MOI=3.00,其中亲本毒株rHEP-Flury差异最显著。当MOI=0.01时,M2、M4在NA细胞中的最大滴度与rHEP-Flury无显著性差异;当MOI=3.00时,M2在NA细胞中的最大滴度显著高于rHEP-Flury(图5C)。
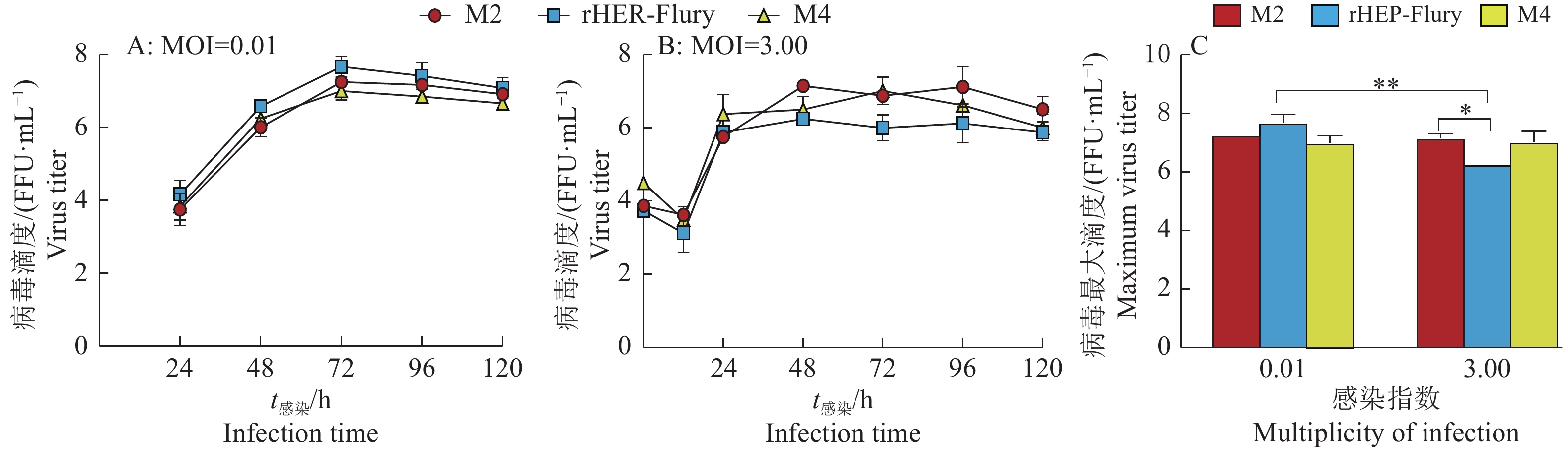
|
图 5 狂犬病病毒在神经母细胞瘤细胞中的生长 Fig. 5 Growth of rabies virus in neuroblastoma cells “*”和“**”分别表示在0.05和0.01水平差异显著(双因素方差分析法) “*” and “**” indicated significant differences at 0.05 and 0.01 levels respectively (Two-way anova method) |
病毒在NA细胞中的扩散试验结果(图6)显示:病毒感染细胞12 h后,利用FITC标记的抗狂犬病病毒的免疫球蛋白进行标记,所有细胞均为阴性;当病毒感染细胞24 h后,所有处理均有阳性细胞呈现,但数量上没有明显差别;当病毒感染36 h后,亲本毒株rHEP-Flury阳性细胞数量略多;当病毒感染48 h后,rHEP-Flury和M4几乎感染了全部细胞,M2此时未能感染全部细胞,这与病毒在NA细胞上的多步生长曲线显示的感染48 h前,M4在NA细胞中的生长速度快于M2一致;当病毒感染60 h后,rHEP-Flury、M2和M4接种的细胞均显示全部被感染。这表明狂犬病病毒M基因的重排只能降低病毒在细胞间的扩散速度,并不能限制病毒在细胞间的扩散。

|
图 6 狂犬病病毒在神经母细胞瘤细胞中的扩散 Fig. 6 Spread of rabies virus in neuroblastoma cells |
前期研究表明单股负链RNA病毒缺少同源重组的能力,基因顺序改变后不会发生回复突变[20],利用RT-PCR方法对本研究M基因重排病毒M2和M4的基因顺序进行验证,结果表明狂犬病病毒HEP-Flury作为单股负链RNA病毒,其基因重排后不会发生回复突变。
M蛋白对狂犬病病毒的转录和复制调节主要是刺激复制抑制转录。本研究显示当病毒感染NA细胞12 h后,病毒N、P、M、G和L mRNA的转录水平与vRNA的合成呈正相关,这可能是因为狂犬病病毒只表达N、P、M、G、L这5种蛋白,它们不仅是狂犬病病毒的功能蛋白还是组成狂犬病病毒粒子的结构蛋白,因此表达受到病毒复制的二级影响[21]。亲本毒株rHEP-Flury具有野生型的基因顺序,最适合病毒的生长和基因表达,因此最先达到病毒复制的需求,细胞内vRNA水平显著高于M基因重排病毒[22]。基因重排病毒M2的M基因从3位移至2位,提高了M基因的转录比例,但是在病毒感染细胞的早期,其病毒基因组RNA的复制水平仍低于亲本毒株rHEP-Flury和M4,这可能是因为狂犬病病毒的RNA、N、P和L蛋白共同形成核糖核蛋白体,核糖核蛋白体是狂犬病病毒转录和复制的核心成分[2]。此外,Pattnaik等[23]发现最佳的N、P、L比例能产生最佳的病毒复制,亲本毒株rHEP-Flury和基因重排病毒M4的N、P、L位置没有发生改变,因此对N、P、L比例影响较小,病毒复制速度较快,而M2的M基因从第3位移至第2位,P基因被动从第2位移至第3位,对N、P、L比例影响较大,因此病毒复制速度较慢。狂犬病病毒LeRNA能够与宿主的La蛋白结合,抑制细胞RNA的合成[24]。近期研究表明致病性狂犬病病毒的LeRNA能够干扰狂犬病病毒N蛋白与vRNA的结合,从而抑制病毒狂犬病病毒的复制[25]。本研究中M基因重排病毒的LeRNA的转录比例明显高于亲本毒株rHEP-Flury,重排后病毒增殖速度降低是否与此相关还有待进一步验证。
Yang等[14]前期研究显示:在低感染指数(MOI=0.1)和高感染指数(MOI=5.0)时M基因重排病毒在BSR细胞上的生长都劣于亲本毒株rHEP-Flury,但M4的生长优于病毒M2,这与本研究病毒感染细胞12 h后病毒在细胞中的增殖速度一致。但是病毒在NA细胞中生长的最大滴度结果显示:当MOI=3.00时,亲本毒株rHEP-Flury感染的NA细胞上清中病毒滴度显著低于MOI=0.01时,同时也低于相同感染指数时的M基因重排病毒,这可能是因为狂犬病病毒HEP-Flury虽然不能诱导NA细胞产生经典的细胞病变,但是能够诱导NA细胞发生凋亡[26-27],且在高感染指数时,诱导细胞凋亡的能力更强[8, 28]。因此,高感染指数时,亲本毒株rHEP-Flury在NA细胞中的最大滴度显著下调,而狂犬病病毒HEP-Flury不能诱导BSR细胞凋亡,因此病毒的最大滴度与感染指数无关。
基因重排病毒M2在NA细胞中生长的最大滴度高于M4,可能与M2在NA细胞中的增殖速度减慢有关,因为狂犬病病毒的缓慢增殖能够维持细胞结构的稳定性,从而有利于病毒的复制。在病毒感染细胞48 h前,M4生长速度快于M2,在细胞间的扩散更快,细胞上清中的病毒滴度高于M2;感染细胞48 h后,几乎所有的细胞被感染,此时M2的慢生长更利于细胞结构的稳定,因此后期细胞上清中的病毒滴度高于M4。病毒M2的M基因移至第2位,转录比例增加可能刺激病毒在单个细胞中的复制,使病毒滴度升高。此外,G蛋白是狂犬病病毒诱导NA细胞凋亡的主要蛋白[29],病毒M4在M基因后移一位时G基因被动前移一位,增强了病毒诱导细胞凋亡的能力,导致病毒在NA细胞上的最大滴度降低。
狂犬病病毒弱毒株的G蛋白能够诱导中枢神经系统凋亡,从而限制病毒扩散[30-31]。虽然HEP-Flury的G蛋白能够诱导神经细胞凋亡,但在低感染指数时,病毒诱导细胞凋亡的能力不足以限制病毒的扩散,因此病毒的扩散速度与病毒增殖速度一致,M4的扩散速度快于M2。
本研究比较分析了狂犬病亲本毒株rHEP-Flury和M基因重排病毒M2、M4在NA细胞中的基因转录、蛋白表达、生长及扩散,推测出狂犬病病毒HEP-Flury的基因顺序通过影响结构基因在一次基因转录中的比例,改变病毒在NA细胞中的生长和扩散特性,这为进一步了解病毒结构基因与体外生物学特性的相关性奠定了基础。
| [1] |
MRAK R E, YOUNG L. Rabies encephalitis in humans: Pathology, pathogenesis and pathophysiology[J]. J Neuropathol Exp Neurol, 1994, 53(1): 1-10. DOI:10.1097/00005072-199401000-00001 (  0) 0) |
| [2] |
ALBERTINI A A, RUIGROK R W, BLONDEL D. Rabies virus transcription and replication[J]. Adv Virus Res, 2011, 79: 1-22. DOI:10.1016/B978-0-12-387040-7.00001-9 (  0) 0) |
| [3] |
BALL L A, PRINGLE C R, FLANAGAN B, et al. Phenotypic consequences of rearranging the P, M, and G genes of vesicular stomatitis virus[J]. J Virol, 1999, 73(6): 4705-4712. DOI:10.1128/JVI.73.6.4705-4712.1999 (  0) 0) |
| [4] |
WERTZ G W, PEREPELITSA V P, BALL L A. Gene rearrangement attenuates expression and lethality of a nonsegmented negative strand RNA virus[J]. Proc Natl Acad Sci USA, 1998, 95(7): 3501-3506. DOI:10.1073/pnas.95.7.3501 (  0) 0) |
| [5] |
FLANAGAN E B, BALL L A, WERTZ G W. Moving the glycoprotein gene of vesicular stomatitis virus to promoter-proximal positions accelerates and enhances the protective immune response[J]. J Virol, 2000, 74(17): 7895-7902. DOI:10.1128/JVI.74.17.7895-7902.2000 (  0) 0) |
| [6] |
陈凯云, 文兆海, 翟少华, 等. 基因重排狂犬病病毒疫苗株免疫效果的初步研究[J]. 中国畜牧兽医, 2019, 46(1): 279-286. (  0) 0) |
| [7] |
文兆海, 毛丽萍, 陈凯云, 等. 基因重排减毒狂犬病病毒的拯救及遗传稳定性的研究[J]. 中国兽医科学, 2018, 48(10): 1235-1241. (  0) 0) |
| [8] |
MEI M, LONG T, ZHANG Q, et al. Phenotypic Consequences in vivo and in vitro of rearranging the P gene of RABV HEP-Flury[J]. Front Microbiol, 2017, 8. doi: 10.3389/fmicb.2017.00120.
(  0) 0) |
| [9] |
MEI M, LONG T, ZHANG Q, et al. Phenotypic consequence of rearranging the N gene of RABV HEP-Flury[J]. Viruses, 2019, 11(5). doi: 10.3390/v11050402.
(  0) 0) |
| [10] |
WIRBLICH C, TAN G S, PAPANERI A, et al. PPEY motif within the rabies virus (RV) matrix protein is essential for efficient virion release and RV pathogenicity[J]. J Virol, 2008, 82(19): 9730-9738. DOI:10.1128/JVI.00889-08 (  0) 0) |
| [11] |
FINKE S, MUELLER-WALDECK R, CONZELMANN K K. Rabies virus matrix protein regulates the balance of virus transcription and replication[J]. J Gen Virol, 2003, 84(6): 1613-1621. DOI:10.1099/vir.0.19128-0 (  0) 0) |
| [12] |
PULMANAUSAHAKUL R, LI J, SCHNELL M J, et al. The glycoprotein and the matrix protein of rabies virus affect pathogenicity by regulating viral replication and facilitating cell-to-cell spread[J]. J Virol, 2008, 82(5): 2330-2338. DOI:10.1128/JVI.02327-07 (  0) 0) |
| [13] |
王朝, 周明, 傅振芳, 等. 狂犬病病毒逃逸宿主天然免疫反应的研究进展[J]. 生命科学, 2017, 29(3): 237-244. (  0) 0) |
| [14] |
YANG X F, PENG J J, LIANG H R, et al. Gene order rearrangement of the M gene in the rabies virus leads to slower replication[J]. Virusdisease, 2014, 25(3): 365-371. DOI:10.1007/s13337-014-0220-1 (  0) 0) |
| [15] |
UGOLINI G. Rabies virus as a transneuronal tracer of neuronal connections[J]. Adv Virus Res, 2011, 79: 165-202. DOI:10.1016/B978-0-12-387040-7.00010-X (  0) 0) |
| [16] |
ZHAO L, TORIUMI H, KUANG Y, et al. The roles of chemokines in rabies virus infection: Overexpression may not always be beneficial[J]. J Virol, 2009, 83(22): 11808-11818. DOI:10.1128/JVI.01346-09 (  0) 0) |
| [17] |
FABER M, FABER M L, PAPANERI A, et al. A single amino acid change in rabies virus glycoprotein increases virus spread and enhances virus pathogenicity[J]. J Virol, 2005, 79(22): 14141-14148. DOI:10.1128/JVI.79.22.14141-14148.2005 (  0) 0) |
| [18] |
DAVIS B M, RALL G F, SCHNELL M J. Everything you always wanted to know about rabies virus (but were afraid to ask)[J]. Annu Rev Virol, 2015, 2(1): 451-471. DOI:10.1146/annurev-virology-100114-055157 (  0) 0) |
| [19] |
WIRBLICH C, SCHNELL M J. Rabies virus (RV) glycoprotein expression levels are not critical for pathogenicity of RV[J]. J Virol, 2011, 85(2): 697-704. DOI:10.1128/JVI.01309-10 (  0) 0) |
| [20] |
PRINGLE C R. The genetics of vesiculoviruses[J]. Arch Virol, 1982, 72(1/2): 1-34. (  0) 0) |
| [21] |
OKUMURA A, HARTY R N. Rabies virus assembly and budding[J]. Adv Virus Res, 2011, 79: 23-32. DOI:10.1016/B978-0-12-387040-7.00002-0 (  0) 0) |
| [22] |
FINKE S, CONZELMANN K K. Replication strategies of rabies virus[J]. Virus Res, 2005, 111(2): 120-131. DOI:10.1016/j.virusres.2005.04.004 (  0) 0) |
| [23] |
PATTNAIK A K, WERTZ G W. Replication and amplification of defective interfering particle RNAs of vesicular stomatitis virus in cells expressing viral proteins from vectors containing cloned cDNAs[J]. J Virol, 1990, 64(6): 2948-2957. DOI:10.1128/JVI.64.6.2948-2957.1990 (  0) 0) |
| [24] |
KURILLA M G, CABRADILLA C D, HOLLOWAY B P, et al. Nucleotide sequence and host La protein interactions of rabies virus leader RNA[J]. J Virol, 1984, 50(3): 773-778. DOI:10.1128/JVI.50.3.773-778.1984 (  0) 0) |
| [25] |
ZHANG R, LIU C, CAO Y, et al. Rabies viruses leader RNA interacts with host Hsc70 and inhibits virus replication[J]. Oncotarget, 2017, 8(27): 43822-43837. (  0) 0) |
| [26] |
PREHAUD C, LAY S, DIETZSCHOLD B, et al. Glycoprotein of nonpathogenic rabies viruses is a key determinant of human cell apoptosis[J]. J Virol, 2003, 77(19): 10537-10547. DOI:10.1128/JVI.77.19.10537-10547.2003 (  0) 0) |
| [27] |
THOULOUZE M I, LAFAGE M, YUSTE V J, et al. High level of Bcl-2 counteracts apoptosis mediated by a live rabies virus vaccine strain and induces long-term infection[J]. Virology, 2003, 314(2): 549-561. DOI:10.1016/S0042-6822(03)00491-4 (  0) 0) |
| [28] |
PENG J, ZHU S, HU L, et al. Wild-type rabies virus induces autophagy in human and mouse neuroblastoma cell lines[J]. Autophagy, 2016, 12(10): 1704-1720. DOI:10.1080/15548627.2016.1196315 (  0) 0) |
| [29] |
LIU X, YANG Y, SUN Z, et al. A recombinant rabies virus encoding two copies of the glycoprotein gene confers protection in dogs against a virulent challenge[J]. PLoS One, 2014, 9(2): e87105. DOI:10.1371/journal.pone.0087105 (  0) 0) |
| [30] |
LAY S, PREHAUD C, DIETZSCHOLD B, et al. Glycoprotein of nonpathogenic rabies viruses is a major inducer of apoptosis in human jurkat T cells[J]. Ann N Y Acad Sci, 2003, 1010: 577-581. DOI:10.1196/annals.1299.108 (  0) 0) |
| [31] |
SARMENTO L, TSEGGAI T, DHINGRA V, et al. Rabies virus-induced apoptosis involves caspase-dependent and caspase-independent pathways[J]. Virus Res, 2006, 121(2): 144-151. DOI:10.1016/j.virusres.2006.05.002 (  0) 0) |
 2020, Vol. 41
2020, Vol. 41


