2. 吉林大学 动物医学学院,吉林 长春 130062
2. College of Veterinary Medicine, Jilin University, Changchun 130062, China
动物模型是科学研究不可缺少的工具,受到取材和伦理道德的限制,生物医学必须依靠动物模型进行广泛的试验和评估。小鼠等啮齿类作为经典的模式动物,一些致病突变在小鼠上引起的症状与在人类引起的症状截然相反,这让啮齿类动物模型无法满足人类疾病相关的研究。家兔是标准的生物医学研究动物模型,它繁殖快,体型适中,比小鼠能更真实地模拟人类疾病的发病机制,目前已经成为多种人类疾病模型和发育机制研究模型的首选动物。家兔疾病模型已被开发用于动脉粥样硬化、糖尿病、脂蛋白代谢和心血管疾病[1]、骨关节炎[2]、眼病[3]、病毒感染以及癌症等多种人类疾病的研究。家兔因其胎盘与人的相似性也经常被用作发育生物学的研究模型[4],比如发育毒性测试和孕期糖尿病等的相关研究[5]。
转录激活样效应因子核酸酶(Transcription activator-like effector nuclease, TALEN)技术已经取代锌指核酸酶(Zinc-finger nuclease, ZFN) 技术成为基因组定点靶向修饰的主流技术,并广泛应用于真核细胞以及多种模式生物的科学研究中。2010 年,Sun 等[6]将全序列合成的 Tale 蛋白与 Fok I 核酸内切酶融合组成TALEN,在人细胞系 HEK293 中对 NTF3 和 CCR5这2个内源基因分别进行了定点敲除,敲除效率达到了39.9%;Tong 等[7]利用 TALEN 技术在大鼠胚胎干细胞中实现了对 BMPR2 基因的敲除;Carlson 等[8]在牛和猪中实现了TALEN 介导的多基因敲除;2013 年赖良学团队将 TALEN 技术应用于兔基因敲除研究,建立了世界首例免疫缺陷家兔疾病模型[9];2014 年,Liu 等[10]通过将 3 对 TALEN 的 mRNA 同时注射到食蟹猴胚胎中,成功获得1只Rett 综合症的雌性猴模型。TALEN技术的出现极大地促进了基因定点修饰动物的研究,为这些克隆效率低下又缺少全能胚胎干细胞物种的基因修饰开辟了新渠道。
Tiki1基因在无翅基因 (Wingless/Int1,Wnt)信号通路中作为抑制因子而发挥作用[11]。在蛙上进行的试验证实了Tiki1在头部诱导过程中起着决定性的作用,Tiki1基因的表达模式与Dickkopf1(DKK1)基因和Goosecoid(Gsc)基因最为接近[12]。Tiki1基因在小鼠上缺失,而在家兔上表达,Tiki1基因的错误调控还与人类的肿瘤等疾病相关[13]。Tiki1基因是Wnt信号通路中新的抑制因子,Tiki1、DKK-1、Gsc 这3个基因之间,是否存在协同的相互作用,它们非重叠的那一部分又在生物发育中扮演着什么样的角色,这些问题都需要借助合适的动物模型进行更加深入和完整的研究。本研究拟利用TALEN技术建立Tiki1基因敲除兔模型,来研究Tiki1基因在家兔发育过程中的作用,以填补Tiki1基因在哺乳动物胚胎发育功能研究上的空白。
1 材料与方法 1.1 材料和试剂分子试验相关试剂无特殊说明均购自TaKaRa 公司,细胞培养相关试剂无特殊说明均购自 Gibco公司,化学试剂无特殊说明均购自Sigma 公司。兔子为新西兰白兔Oryctolagus cuniculus,普通级,雌性6~8 月龄,体质量2.0~4.5 kg,购自南方医科大学实验动物中心,试验动物生产许可证为[SCXK(粤)2011—0015]。所有的动物试验均按照试验动物使用操作规程进行操作, 试验动物使用许可证为[SYXK(粤)2010—0063]。
1.2 TALEN质粒设计与构建 1.2.1 TALEN质粒设计家兔Tiki1基因的mRNA由哈佛大学医学院神经学实验室提供,mRNA长度为1 518 bp,有7个外显子。根据Cermak等[14]给出的靶点设计原则,我们在Tiki1基因上设计了4对TALEN。
1.2.2 TALEN的合成与组装按照Golden Gate TALEN试剂盒说明书组装如下:比照TALEN靶点序列,分别把Golden Gate TALEN试剂盒前10 个碱基相应的重复可变双残基(Repeat-variable di-residue, RVD)以及载体pFUS_A加入到1个反应体系中;把Golden Gate TALEN试剂盒11~15的碱基相应的RVD以及载体pFUS_B#15加入到1个反应体系中;下游靶点序列按照同样方式操作。
为了减少未连接成环状的dsDNA片段的影响,在连接后向每个体系中加入Plasmid-Safe nuclease,将DNA片段消化掉。
1.2.3 转化感受态细胞悬液解冻后立即置于冰上。加入上述连接质粒DNA溶液10 μL,42 ℃水浴中热击90 s,热击后迅速置于冰上冷却3~5 min。向管中加入500 μL LB液体培养基(不含Amp),混匀后37 ℃条件下振荡培养30 min。将上述菌液摇匀后取100 μL涂布于含Amp的筛选平板上,37 ℃条件下培养16~24 h。每个平板挑选1~3 个白斑,进行PCR检测,挑选正确的克隆过夜培养,小量提取质粒鉴定。
1.2.4 体外转录获得的TALEN质粒需要转录为mRNA并在末端加上Ploy (A),采用mMESSAGE kit、mMACHINE T7 和Poly(A) polymerase。具体转录步骤根据mMESSAGE kit说明书操作。
1.2.5 RNA加尾修饰按以下顺序加入用于加尾的试剂:20 μL mMESSAGE mMACHINE T7 Ultra reaction、36 μL Nuclease-free water、20 μL 5×E-PAP缓冲液、10 μL 25 mmol/L MnCl2溶液、10 μL ATP 溶液。
加入4 μL E-PAP酶,轻轻混匀,37 ℃条件下孵育30~45 min,之后置于冰上。在加入E-PAP酶前先预留出2.5 μL的反应混合液,作为电泳检测是否加尾成功的对照。
1.2.6 RNA产物回收采用氯化铝沉淀的方式回收RNA。具体步骤如下:加入50 μL的LiCl沉淀液体中止加尾反应和沉淀RNA。4 ℃、12 000 r/min离心15 min,收集RNA颗粒。小心去除上清,用1 mL体积分数为70%的乙醇溶液清洗1次,4 ℃、12 000 r/min离心15 min。小心移去乙醇溶液,重悬于RNAse-free 0.1×TE缓冲液中。利用NanoDrop-1000检测质量浓度,将mRNA稀释到50 ng/μL,每10 μL分装1管储存于−80 ℃冰箱。
1.2.7 TALEN的打靶效率检测为了验证TALEN的打靶效率,分别以设计的4对TALEN转染家兔胎儿成纤维细胞,转染48 h后,收取该细胞并提取基因组,然后扩增包含靶位点在内的序列,并用T7EⅠ内切酶酶切。
1.3 兔受精卵的显微注射 1.3.1 母兔的超排和受精卵的收集对供体母兔注射100 IU的孕马血清促性腺激素(Pregnant mare serum gonadotropin,PMSG),注射72~120 h后与公兔合笼配种,对于配种成功的母兔进行耳缘静脉注射100 IU的人绒毛膜促性腺激素(Human chorionic gonadotropin,HCG),注射18~20 h后对母兔实施安乐死。取出卵巢和输卵管置于预热的1×PBS缓冲液中,然后用冲卵液从输卵管冲出受精卵,挑选原核期前后的受精卵置于胚胎培养液中,38.5 ℃、CO2体积分数为5%的饱和湿度的培养箱内孵育待用。
1.3.2 显微操作针的制备固定针的制备:固定针要求外径为0.12 ~0.18 mm,内径为0.02~0.03 mm。具体操作:在酒精灯上加热玻璃管中部,变软后离开火焰并迅速拉开,在外径0.12~0.18 mm处用砂轮切断玻璃针;然后将针安装到锻针仪上,锻针端口受热后逐渐收缩至0.02~0.03 mm,停止加热;在距离针尖0.3~0.4 mm处靠近加热的玻璃球,使玻璃针受热后弯向玻璃球,弯至30°时停止加热。
注射针的制备:胞质内注射针选择带有玻璃芯的玻璃管,利用程序拉针仪拉制,其针尖端封口。与固定管一样,在400 μm左右的位置烤成约30°的弯,便于操作。
1.3.3 受精卵的RNA显微注射把原核期的受精卵放置在1个操作液滴中,用移液枪将约1 μL的Tiki1-TALEN mRNA沿注射针玻璃管内壁注射到底部,待气泡排出后,将注射针的针尖在固定针上撞开1个开口,调节mRNA流出的流速,可在透明带下调节。将注射针刺入受精卵的胞质内,注入适量的Tiki1-TALEN mRNA,可以看到胞质略有膨胀即可拔出注射针。
1.4 囊胚基因型打靶结果的鉴定每个囊胚用5 μL的 NP40裂解液在 PCR 仪中进行裂解,然后进行PCR或者巢式PCR来扩增目的片段,并测序鉴定各基因是否打靶。测序结果显示有2套或2套以上峰图的产物克隆到T载体后测序,以确定最终的基因突变情况。
1.5 Tiki1基因敲除兔的制备为了进一步获得Tiki1基因敲除兔,将50 ng/μL的Tiki1-TALEN mRNA注射到17个原核期的家兔受精卵胞质中,并将受精卵分别移植到2只受体兔体内,分别编号为1#和2#。统计仔兔出生情况,并检测仔兔的基因突变。
2 结果与分析 2.1 Tiki1基因TALEN靶位点的设计根据哈佛大学医学院神经学实验室提供的人Tiki1基因序列,在家兔基因组序列中进行比对,找出家兔上的Tiki1基因,选择其最保守的第2外显子进行TALEN靶位点的设计(图1),根据GOLD GATE TALEN试剂盒说明书合成了4条TALEN,分别命名为L1、L2、R1和R2,这4条TALEN可以组合成4 对TALEN,TALEN组合命名为L1R1、L1R2、L2R1和L2R2。以此4对TALEN转染家兔胎儿成纤维细胞,T7EⅠ内切酶酶切图如图2所示,从图2中可以看出以L1R1组合转染家兔胎儿成纤维细胞提取的基因组扩增的PCR产物可以经T7EⅠ内切酶切出2条带,其他组合的未见明显条带,说明在这4种TALEN组合中,L1R1打靶效率最高。因此,选择L1R1对家兔Tiki1基因进行打靶研究。
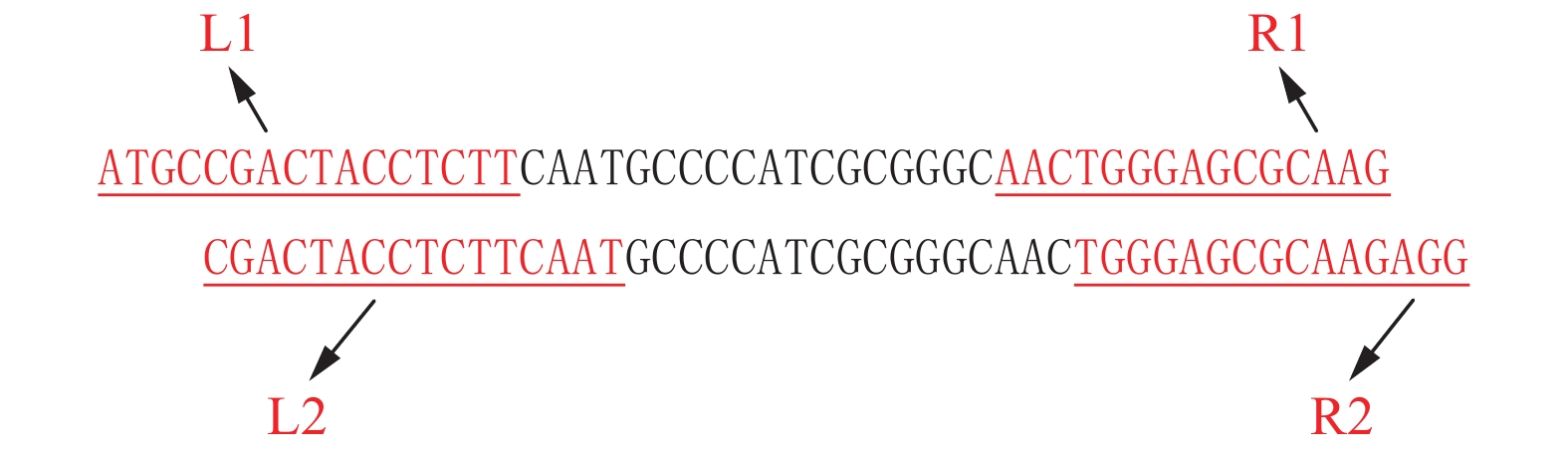
|
图 1 Tiki1基因TALEN靶位点 Fig. 1 Target sites for TALEN of Tiki1 gene 加下划线的序列表示靶位点 The underlined sequences represent the target sites |
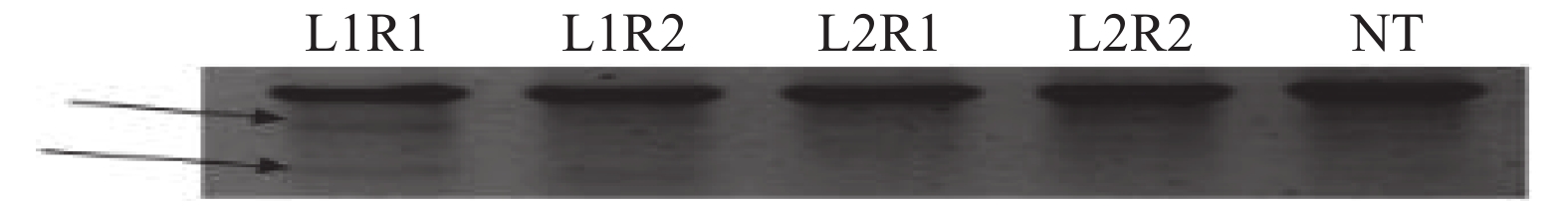
|
图 2 4 对TALEN打靶效率酶切检测电泳图 Fig. 2 Electrophoregram detecting targeting efficiency of four pairs of TALEN by enzyme digestion NT为阴性对照;箭头所指为T7EⅠ内切酶切开的2个片段条带 NT is negative control; The arrows point at two fragments generated by the T7E I endonuclease |
选取原核期受精胚胎,分别以10和50 ng/μL的质量浓度将Tiki1-TALEN mRNA注射到处于原核期的家兔受精卵的胞质内,统计发育到囊胚期的受精卵,计算囊胚率,检测注射的RNA对胚胎发育是否存在影响。结果见表1,当以10 ng/μL的质量浓度注射时,囊胚率为64%;当以50 ng/μL的质量浓度注射时,囊胚率为57%;对照组以相同量的H2O注射,囊胚率为82%。结果表明,试验组与对照组相比囊胚发育率稍有降低,但统计学差异均不显著,说明注射的mRNA对囊胚的发育会有影响但可以忽略,而且注射mRNA组的囊胚率都在50%以上,所以并不会影响基因修饰兔的建立。
|
|
表 1 注射Tiki1-TALEN mRNA后家兔胚胎的体外发育结果 Table 1 Results of in vitro development of rabbit embryos after injection with Tiki1-TALEN mRNA |
基因打靶效率检测结果如表2所示。注射10 ng/μL Tiki1-TALENs mRNA后所获得的7 个囊胚中,有1个囊胚(14.3%)发生了基因打靶,进一步进行基因型的深度分析,发现该囊胚除了检测到该基因存在碱基缺失外,还能检测到野生型(WT)序列,为单等位基因敲除;注射50 ng/μL Tiki1-TALENs mRNA后所获得的8个囊胚中,100%发生了基因打靶,进一步进行基因型的深度分析,全部都只发生了单等位基因敲除,没有检测到双等位基因敲除。在以上9个发生基因打靶的囊胚中,存在5种基因突变方式,分别为缺失1、6、10、15和28 bp碱基,具体突变位置见图3。注射50 ng/μL Tiki1-TALEN mRNA试验组的囊胚基因修饰效率显著高于10 ng/μL注射组(P=0.001 4,Fisher’s精确检验)。综上,结合囊胚率和打靶效率考虑,我们选择以50 ng/μL质量浓度的Tiki1-TALEN mRNA注射家兔受精卵制备基因打靶兔。
|
|
表 2 注射Tiki1-TALEN mRNA后家兔受精卵Tiki1基因修饰结果 Table 2 Modified results of Tiki1 gene in fertilized eggs of rabbits after injection with Tiki1-TALEN mRNA |
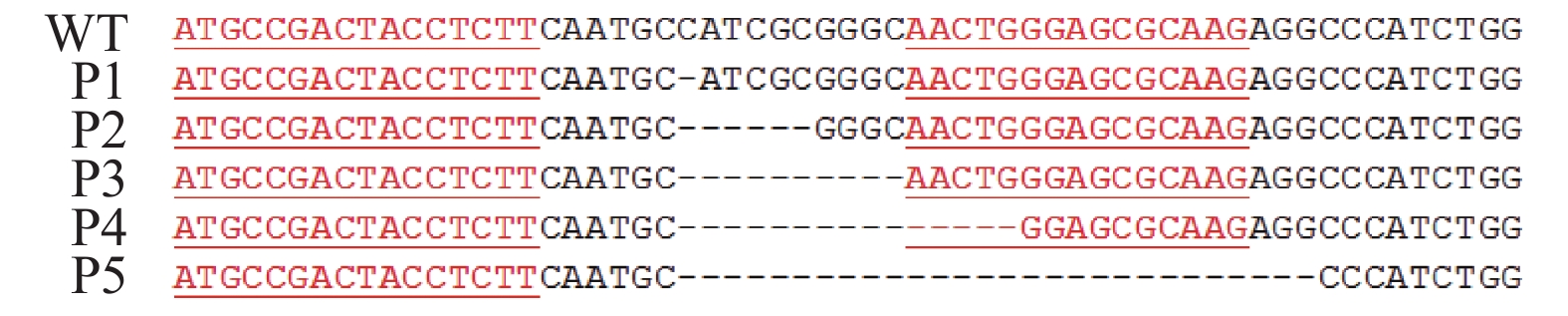
|
图 3 注射Tiki1-TALEN mRNA后家兔受精卵Tiki1基因的碱基突变 Fig. 3 Base mutation of Tiki1 gene in fertilized eggs of rabbits after injection with Tiki1-TALEN mRNA WT为野生型,P1~P5代表5种不同的基因突变型;短横线表示缺失的碱基,加下划线的序列表示靶位点 WT is wildtype, P1-P5 represent five different types of gene mutation; The short dashes represent the missing bases, and the underlined sequences represent the target sites |
将50 ng/μL的Tiki1-TALEN mRNA注射到17枚原核期的家兔受精卵胞质中,分别移植到2只受体兔体内,共计出生3个活的仔兔,编号为1#-1、2#-1和2#-2(表3)。测序分析其基因型发现1#-1和2#-2仔兔在靶位点处出现双峰(图4),说明发生了基因打靶;2#-1仔兔测序结果为单峰(图4),进一步比对该测序结果发现其为WT型,说明未发生基因打靶。回收测序结果在靶位点附近存在套峰的2只仔兔(1#-1和2#-2)的PCR 产物,与T载体连接测序,再与WT序列进行比对。结果表明,对于仔兔1#-1,Tiki1基因除了检测到WT型序列外,还在靶位点发生了基因敲除,具体突变方式为插入1 bp的同时删减了9 bp;对于仔兔2#-2,Tiki1基因除了检测到WT型序列外,还在靶位点发生了基因敲除,具体突变方式为删减22 bp(图5)。以上结果表明,我们建立了Tiki1基因敲除兔模型,但是这些基因敲除兔仍存在正常的Tiki1基因序列,说明这些打靶兔可能是单基因敲除或者是嵌合体。
|
|
表 3 基因打靶仔兔出生统计 Table 3 Birth summary of gene targeted baby rabbits |
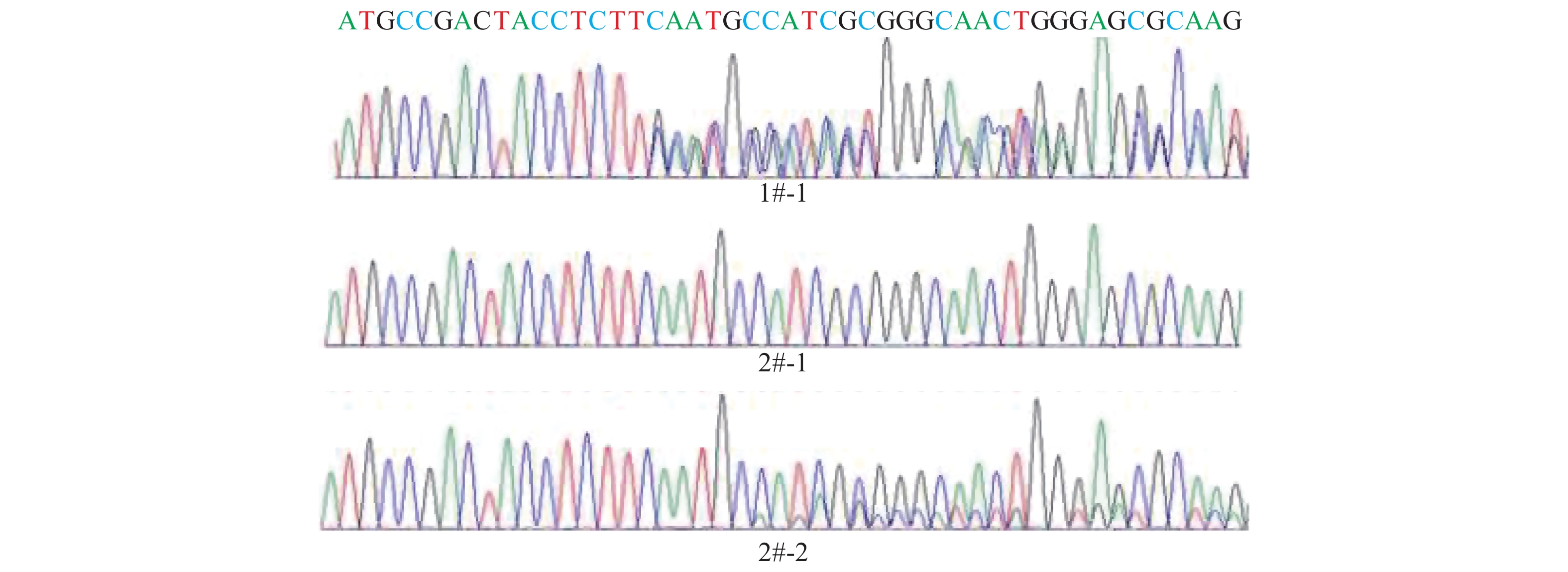
|
图 4 仔兔Tiki1基因测序峰图 Fig. 4 Sequencing peak map of Tiki1 gene in baby rabbit |
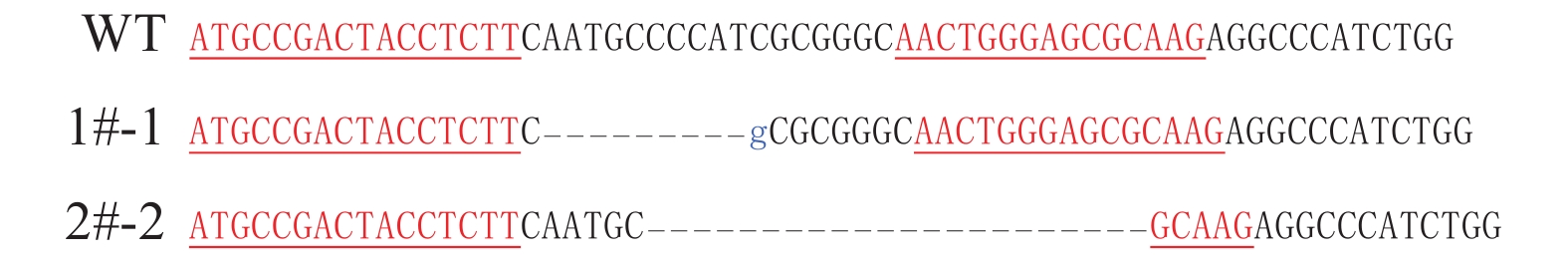
|
图 5 仔兔Tiki1基因的碱基突变 Fig. 5 Base mutations in Tiki1 genes of baby rabbits WT为野生型,1#-1和2#-2为仔兔编号;短横线表示缺失的碱基,蓝色小写字母表示插入的碱基,加下划线的序列表示靶位点 WT is wildtype, 1#-1 and 2#-2 are indentifying numbers for baby rabbits; The short dashes represents the missing base, the blue lowercase letter represents the inserted base, and the underlined sequences represent the target sites |
Tiki1基因是哈佛大学儿童医学院贺熹教授团队发现在Wnt信号通路中作为抑制因子而发挥作用的一个基因,抑制其表达会导致蛙胚中头部发育的缺失[11]。Tiki1基因在小鼠体内缺失,因而无法在小鼠体内研究其功能,而家兔中该基因能够表达[15]。基于Tiki1基因的特殊性,即Tiki1基因在啮齿类动物,如小鼠、大鼠体内缺失,使用小鼠作为研究人类 Tiki1基因缺失的疾病模型这一传统途径被阻断,而兔由于心血管系统与人类非常相似,因此成为研究 Tiki1 基因极佳的哺乳动物。
本研究利用自身常年做基因编辑兔的技术优势试图探索Tiki1基因对兔早期胚胎发育的影响。我们首先基于TALEN系统设计了靶向兔Tiki1基因的打靶载体,然后分别将10和50 ng/μL的Tiki1-TALEN mRNA注射到原核期的家兔受精卵胞质中,并进行囊胚率和基因修饰效率的统计,检测所设计的Tiki1-TALEN mRNA对胚胎发育是否存在影响以及能否高效地对胚胎进行基因修饰。结果表明,当以10 ng/μL的质量浓度注射时,囊胚率为64%,对照组以相同量的H2O注射,囊胚率为82%,试验组与对照组相比囊胚发育稍差但无明显统计学差异。当以50 ng/μL的质量浓度注射时,囊胚率为57%,该试验组与对照组相比囊胚发育稍差,但无明显统计学差异,说明注射的mRNA对囊胚的发育没有明显影响,而且后续只要在胚胎移植时多移植几枚胚胎不影响获得想要的基因修饰兔。注射50 ng/μL mRNA试验组的囊胚率(57%)与注射10 ng/μL mRNA试验组的囊胚率(64%)相比影响不大,但注射50 ng/μL mRNA试验组的囊胚基因修饰效率(100%)与注射10 ng/μL mRNA试验组的囊胚基因修饰效率(14.3%)相比显著提高,这有助于提高后续获得阳性的Tiki1基因修饰兔子的效率,所以后续试验选择了以50 ng/μL的质量浓度注射来高效获得基因修饰兔,后续将进一步获得纯合子以检测其是否影响胚胎的后期发育或者成体发育。
| [1] |
NIIMI M, YANG D, KITAJIMA S, et al. ApoE knockout rabbits: A novel model for the study of human hyperlipidemia
[J]. Atherosclerosis, 2016, 245: 187-193. DOI:10.1016/j.atherosclerosis.2015.12.002 (  0) 0) |
| [2] |
MARTINEZ-CALATRAVA M J, LARGO R, HERRERO-BEAUMONT G. Improvement of experimental accelerated atherosclerosis by chondroitin sulphate[J]. Osteoarthr Cartilage, 2010, 18(S1): S12-S16. (  0) 0) |
| [3] |
ZERNII E Y, BAKSHEEVA V, IOMDINA E N, et al. Rabbit models of ocular diseases: New relevance for classical approaches[J]. CNS Neurol Disord-Dr, 2016, 15(3): 267-291. DOI:10.2174/1871527315666151110124957 (  0) 0) |
| [4] |
OKAMOTO I, PATRAT C, THEPOT D, et al. Eutherian mammals use diverse strategies to initiate X-chromosome inactivation during development[J]. Nature, 2011, 472(7343): 370-374. DOI:10.1038/nature09872 (  0) 0) |
| [5] |
BOURDON M, TORRES-ROVIRA L, MONNIAUX D, et al. Impact of a gestational exposure to diesel exhaust on offspring gonadal development: Experimental study in the rabbit[J]. J Dev Orig Health Dis, 2018, 9(5): 519-529. DOI:10.1017/S2040174418000351 (  0) 0) |
| [6] |
SUN N, LIANG J, ABIL Z, et al. Optimized TAL effector nucleases (TALENs) for use in treatment of sickle cell disease[J]. Mol Bio Syst, 2012, 8(4): 1255-1263. (  0) 0) |
| [7] |
TONG C, HUANG G, ASHTON C, et al. Rapid and cost-effective gene targeting in rat embryonic stem cells by TALENs[J]. J Genet Genom, 2012, 39(6): 275-280. DOI:10.1016/j.jgg.2012.04.004 (  0) 0) |
| [8] |
CARLSON D F, TAN W, LILLICO S G, et al. Efficient TALEN-mediated gene knockout in livestock[J]. P Natl Acad Sci USA, 2012, 109(43): 17382-17387. DOI:10.1073/pnas.1211446109 (  0) 0) |
| [9] |
SONG J, YANG D, XU J, et al. RS-1 enhances CRISPR/Cas9- and TALEN-mediated knock-in efficiency[J]. Nat Commun, 2016, 7: 10548. DOI:10.1038/ncomms10548 (  0) 0) |
| [10] |
LIU H, CHEN Y, NIU Y, et al. TALEN-mediated gene mutagenesis in rhesus and cynomolgus monkeys[J]. Cell Stem Cell, 2014, 14(3): 323-328. DOI:10.1016/j.stem.2014.01.018 (  0) 0) |
| [11] |
ZHANG X, MACDONALD B T, GAO H, et al. Characterization of tiki, a new family of wnt-specific metalloproteases[J]. J Biol Chem, 2016, 291(5): 2435-2443. DOI:10.1074/jbc.M115.677807 (  0) 0) |
| [12] |
ZHANG X, ABREU J G, YOKOTA C, et al. Tiki1 is required for head formation via Wnt cleavage-oxidation and inactivation[J]. Cell, 2012, 149(7): 1565-1577. DOI:10.1016/j.cell.2012.04.039 (  0) 0) |
| [13] |
REIS A H, MACDONALD B T, FEISTEL K, et al. Expression and evolution of the Tiki1 and Tiki2 genes in vertebrates
[J]. Int J Dev Biol, 2014, 58(5): 355-362. DOI:10.1387/ijdb.140106ja (  0) 0) |
| [14] |
CERMAK T, DOYLE E L, CHRISTIAN M, et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting[J]. Nucleic Acids Res, 2011, 39(12): e82. DOI:10.1093/nar/gkr218 (  0) 0) |
| [15] |
ZHANG X, HE X. Methods for studying Wnt protein modifications/inactivations by extracellular enzymes, Tiki and Notum[M]//BARRETT Q, LUM L. Wnt Signaling, New York: Humana Press, 2016: 29-38.
(  0) 0) |
 2020, Vol. 41
2020, Vol. 41


