2. 广东省动物源性人兽共患病预防与控制重点实验室/华南农业大学 兽医学院,广东 广州 510642
2. Key Laboratory of Zoonosis Prevention and Control of Guangdong Province/College of Veterinary Medicine, South China Agricultural University, Guangzhou 510642, China
H9N2禽流感在1994年首次暴发于我国广东省,引起蛋鸡产蛋率降低(14%~75%)和肉鸡死亡(10%~40%)[1]。在过去20多年中,H9N2禽流感病毒(Avian influenza virus,AIV)迅速蔓延传播,成为我国流行最为广泛的AIV亚型[2]。H9N2 AIV与细菌、病毒等病原体共感染时,可造成家禽死亡率大幅升高,对家禽养殖业构成严重威胁。同时,H9N2亚型禽流感已出现跨种传播的现象,广东、香港、湖南和安徽均出现人感染H9N2 AIV的病例[2]。
同义密码子的使用具有某种倾向性,并非是一个随机的过程。在蛋白质翻译过程中,部分同义密码子会比其他密码子使用度更高,这被称为密码子使用偏好性(Codon usage bias)[3]。若病毒在同一宿主长时间循环,则该病毒的密码子使用模式将趋同于宿主的密码子使用模式(即病毒使用与宿主相同的同义密码子以翻译某一氨基酸),以便更好地利用宿主细胞资源进行病毒复制、繁殖[4]。研究密码子使用模式有助于解释病毒在基因进化过程中所发生的适应性变化,进一步认识病毒的基因表达调控,有助于病毒疫苗的研制[5-6]。H9N2 AIV对家禽养殖和人类健康的威胁日益加剧,但目前对H9N2 AIV密码子使用模式的研究较少。
为了探讨近年来H9N2 AIV的密码子使用偏好性及其影响因素,本研究选取90株2010—2018年H9N2 AIV的全基因组为研究对象,分析其碱基组成特性、最优密码子、密码子使用偏好性的影响因素以及病毒对宿主密码子使用模式的适应性,以期为H9N2 AIV基因进化、已有疫苗密码子的优化和新型疫苗(密码子去优化疫苗)研制提供理论依据。
1 材料与方法 1.1 序列来源利用NCBI数据库(
利用CodonW 1.4.4软件计算H9N2 AIV基因组的密码子使用偏好性参数,包括同义密码子第3位各碱基(A3S、U3S、C3S和G3S)含量,同义密码子第3位的GC(GC3S)含量,有效密码子数(Effective number of codons,ENC),总平均亲水性(General average hydropathicity)和芳香性(Aromaticity)。用Puigbò等[7]的方法计算各碱基(A、U、C和G)含量,GC含量,密码子第3位各碱基(A3、U3、C3和G3)含量,密码子第3位的GC(GC3)含量,密码子第1、2位的GC(GC12)平均含量,同义密码子相对使用度(Relative synonymous codon usage)和密码子适应指数(Codon adaptation index)。宿主鸡Gallus gallus和人Homo sapiens的密码子使用模式获取自密码子使用数据库(
ENC为20~60,与密码子使用偏好性呈负相关,ENC≤35说明密码子使用偏好性高,ENC>50说明密码子使用偏好性低[9]。ENC绘图是以ENC为纵坐标、GC3S含量为横坐标绘制的二维散点图,若基因组位于标准曲线(即ENC期望与GC3S含量的函数关系)上或其附近,表明密码子使用偏好性仅受突变压力影响;若基因组位于标准曲线下方较远的位置,表明密码子使用偏好性受其他因素(如自然选择)影响。ENC期望与GC3S含量的函数关系为:
| ${\rm{ENC}}_{\text{期望}}=2+s+\frac{29}{{{s}^{2}}+{{\left( 1-s \right)}^{2}}}{\text{,}}$ |
式中,s为GC3S含量[9]。
1.4 中性绘图分析采用中性绘图[w(GC3)为横坐标、w(GC12)为纵坐标]分析突变压力和自然选择对密码子使用偏好性的影响程度。若回归曲线斜率接近1,即基因组几乎沿对角线分布,表示密码子使用偏好性仅受突变压力影响;随着斜率逐渐减小,甚至减小为0,自然选择对密码子使用偏好性的作用逐渐增大[5]。
1.5 PR2绘图分析以w(A3)/w(A3+U3)为纵坐标、w(G3)/w(G3+C3)为横坐标绘制二维散点图,若w(A3)=w(U3)且w(C3)=w(G3),基因组均匀分布于图中心(横、纵坐标均为0.5),表明突变压力和选择率(替代率)对密码子使用的作用相等;反之,两者作用存在差异[5]。
1.6 密码子适应指数分析密码子适应指数为0~1,值越高,病毒对宿主的适应性越强[10]。密码子适应指数和ENC之间的相关性分析可确定突变压力和自然选择对密码子使用偏好性的相对影响。若自然选择优于突变压力,则密码子适应指数和ENC之间的相关性高,相关系数(r)趋向于−1;相反,若突变压力更为重要,则r应接近0,密码子适应指数和ENC之间无相关性[11]。
1.7 对应性分析同义密码子相对使用度是其实际使用频率与期望频率(某一特定氨基酸的所有同义密码子能被均等使用的情况下该密码子的使用频率)的比率。同义密码子相对使用度等于1代表对应氨基酸无密码子使用偏好性,大于1代表对应氨基酸对该密码子的使用较多,小于1代表对该密码子的使用较少。对应性分析(Correspondence analysis)是常用于研究密码子使用模式的多元统计分析方法,基于各基因组的同义密码子相对使用度,将每个基因组依密码子使用频率在1个59维的向量空间内表示[12]。
1.8 最优密码子分析各基因组的ENC从高到低依次排序,从上限区域和下限区域各取10%的基因组作为高、低表达组,使用t检验分析两组之间密码子使用的差异显著性。比较两组的同义密码子相对使用度,若差异≥0.08,并且高表达组显著高于低表达组(P<0.01),则该密码子被定义为最佳密码子[9, 13]。
1.9 统计学分析使用CodonW 1.4.4软件对同义密码子相对使用度进行对应性分析,通过SPSS 23.0软件完成t检验和Spearman相关性分析,使用SigmaPlot 12.5软件绘图。
2 结果与分析 2.1 碱基组成特性由2010—2018年H9N2 AIV基因组的碱基组成分析结果(表1)可知,各碱基含量为A(32.92%~33.03%)>G(24.65%~24.72%)>U(22.88%~22.90%)>C(19.41%~19.49%)。2010—2018年GC含量整体呈下降趋势,且均少于50%,提示H9N2 AIV基因组中AU含量多于GC含量,碱基组成更倾向于使用AU,随着时间推移,该倾向性逐渐加大。同义密码子第3位碱基含量为A3S(41.42%~41.80%)>U3S(30.76%~30.95%)>G3S(27.74%~28.11%)>C3S(26.51%~26.73%),GC3S含量变化与GC相同,说明以A或U作为末位碱基的同义密码子的使用度较大,随着时间推移,其使用度将逐渐增大。
|
|
表 1 H9N2禽流感病毒基因组的碱基组成分析1) Table 1 Base composition analysis of H9N2 avian influenza virus genomes |
以ENC为依据形成的高、低表达组数据如表2所示,大部分同义密码子相对使用度差异较小且无显著性差异,仅有10个密码子(UUU、GUU、GUA、GCU、GCC、GAU、CGU、CGA、GGU和GGC)满足相对使用度差异≥0.08且高表达组相对使用度显著高于低表达组(P<0.01)的条件,因此这10个密码子可判定为最优密码子。其中有8个最优密码子均以A或U结尾,仅有2个以C结尾,说明最优密码子的形成可能主要受到碱基组成限制,可能存在突变压力影响密码子使用偏好性。
|
|
表 2 H9N2禽流感病毒基因组的最优密码子分析 Table 2 Optimal codon analysis of H9N2 avian influenza virus genomes |
2010—2018年H9N2 AIV的ENC为51.90~55.01,平均值52.86,标准差0.64,说明H9N2 AIV的密码子使用偏好性较低。在ENC绘图(图1)分析中,所有点均聚集于标准曲线的下方,说明H9N2 AIV密码子使用偏好性的主导因素并非突变压力,而是其他影响因素如自然选择。
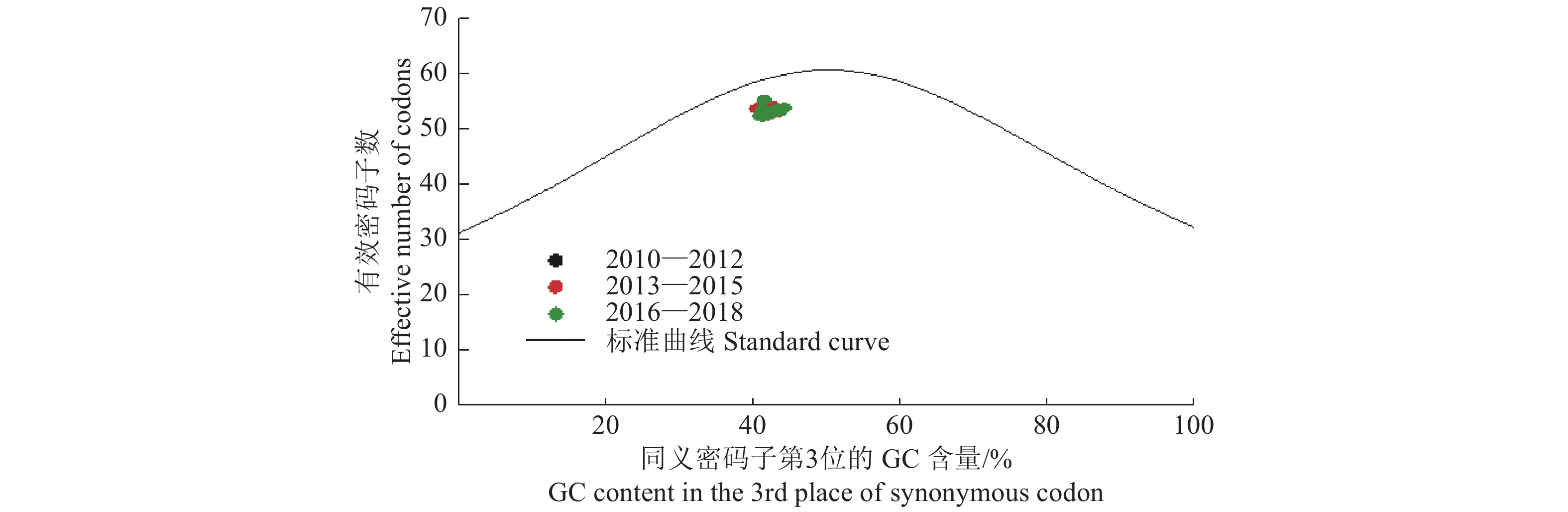
|
图 1 不同年份H9N2禽流感病毒的有效密码子数绘图分析 Fig. 1 Plot analysis of effective number of codons of H9N2 avian influenza virus in different years |
PR2绘图分析结果(图2)显示,2010—2018年H9N2 AIV基因组主要聚集于图中心(横、纵坐标均为0.5)的右上方,基因组对AU的使用度大于GC,其中w(A3)>w(U3)且w(G3)>w(C3),这表明密码子使用偏好性可能由突变压力和其他因素(如自然选择)共同决定,但两者作用存在差异。
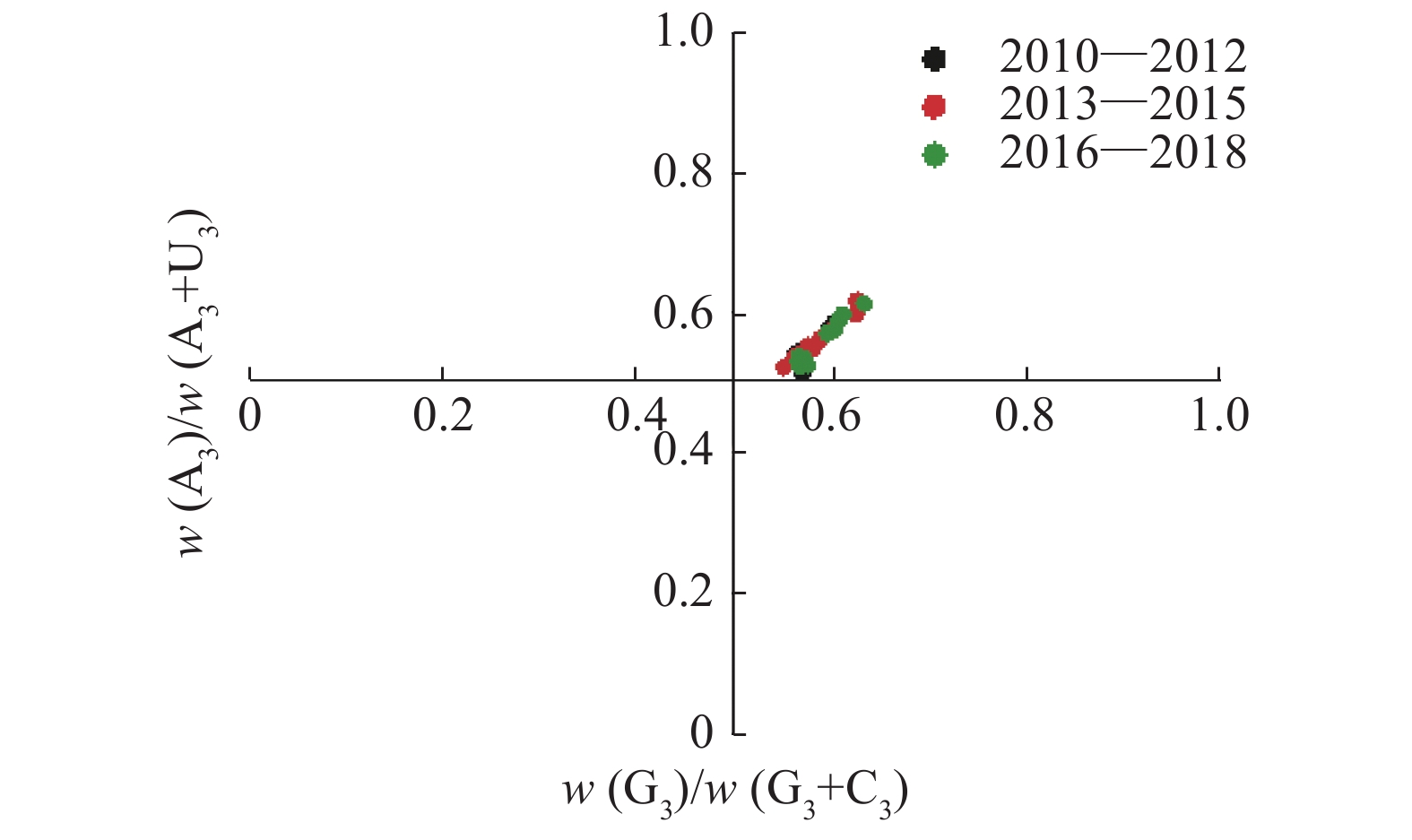
|
图 2 不同年份H9N2禽流感病毒的PR2绘图分析 Fig. 2 PR2 plot analysis of H9N2 avian influenza virus in different years 下标“3”代表密码子第3位 The subscripted “3” indicates the 3rd place of codon |
对2010—2018年H9N2 AIV进行对应性分析(图3),结果显示第1、2轴分别解释34.65%和21.34%的变异,大部分基因组都聚集在原点(横、纵坐标均为0)周围,彼此之间不存在太大差异,这提示各基因组间的密码子使用偏好性差异相对较小。第1、2轴与碱基组成指数进行相关性分析(表3),结果显示第1、2轴与ENC均呈极显著正相关(r=0.633、0.216,P<0.001);第1、2轴与大部分碱基组成指数呈显著相关,其中第1轴与GC含量呈极显著正相关(r=0.510,P<0.001),第2轴与G3S含量呈极显著负相关(r=−0.717,P<0.001)。这提示碱基组成对密码子使用偏好性产生影响。
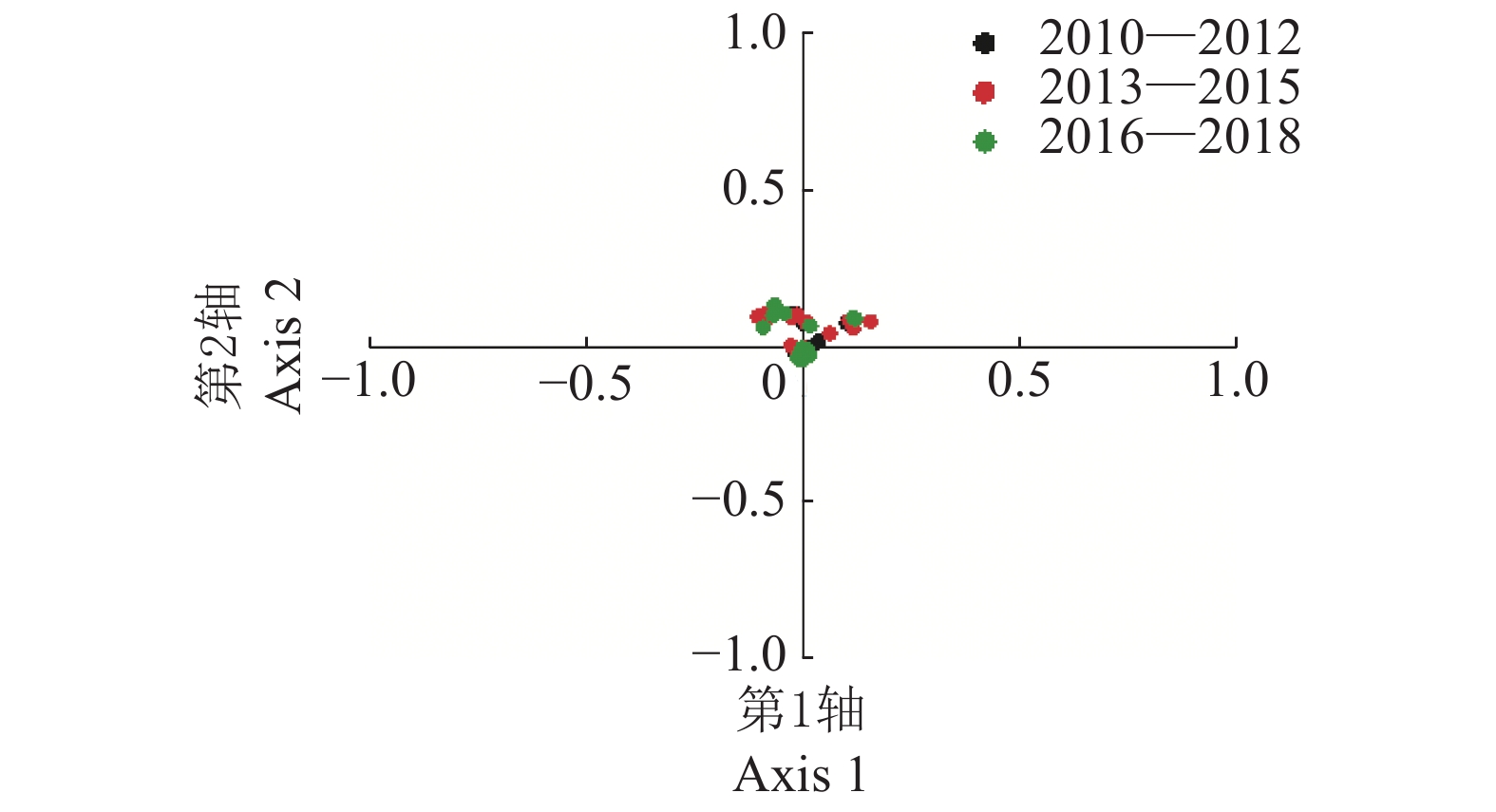
|
图 3 不同年份H9N2禽流感病毒的对应性分析 Fig. 3 Correspondence analysis of H9N2 avian influenza virus in different years |
|
|
表 3 前两主轴对应性分析及与密码子使用偏好性主要参数的相关性分析 Table 3 Correspondence analyses of the first two principle axes and correlation analyses between the first two principle axes and main parameters of codon usage bias |
第1、2轴与总平均亲水性、芳香性进行相关性分析(表3),结果显示第1、2轴与总平均亲水性、芳香性均存在显著相关性,这说明氨基酸的疏水性和芳香性均显著影响密码子使用偏好性。此外,对2010—2018年H9N2 AIV进行密码子适应指数分析(表4),结果显示H9N2对宿主的密码子适应指数平均值随时间变化不大,但H9N2 AIV对鸡的密码子适应指数平均值(0.766~0.768)均高于对人的(0.739~0.741)。同时,密码子适应指数与ENC相关性分析发现,除了2013—2015年H9N2 AIV对人的密码子适应指数与ENC相关性不显著(r=−0.223,P=0.237),其余密码子适应指数均与ENC呈显著负相关,说明自然选择对H9N2 AIV的密码子使用偏好性产生重要影响,但r≠−1,所以也存在突变压力产生影响。
|
|
表 4 H9N2 禽流感病毒基因组的密码子适应指数分析1) Table 4 Codon adaptation index analysis of H9N2 avian influenza virus genomes |
由中性绘图分析和GC12含量与GC3含量相关性分析(图4)可知,2010—2012、2013—2015和2016—2018年的GC12含量与GC3含量均呈极显著负相关(r=−0.720、−0.826、−0.595,P<0.001),提示密码子的这3个位置均受到突变压力的影响。对2010—2012、2013—2015和2016—2018年的GC12含量与GC3含量进行线性回归分析发现,斜率分别为−0.238 5、−0.382 1和−0.346 4,表明突变压力对H9N2 AIV密码子使用偏好性的影响分别为23.85%(2010—2012年)、38.21%(2013—2015年)和34.64%(2016—2018年),所以自然选择对密码子使用偏好性的影响较大,分别为76.15%(2010—2012年)、61.79%(2013—2015年)和65.36%(2016—2018年)。
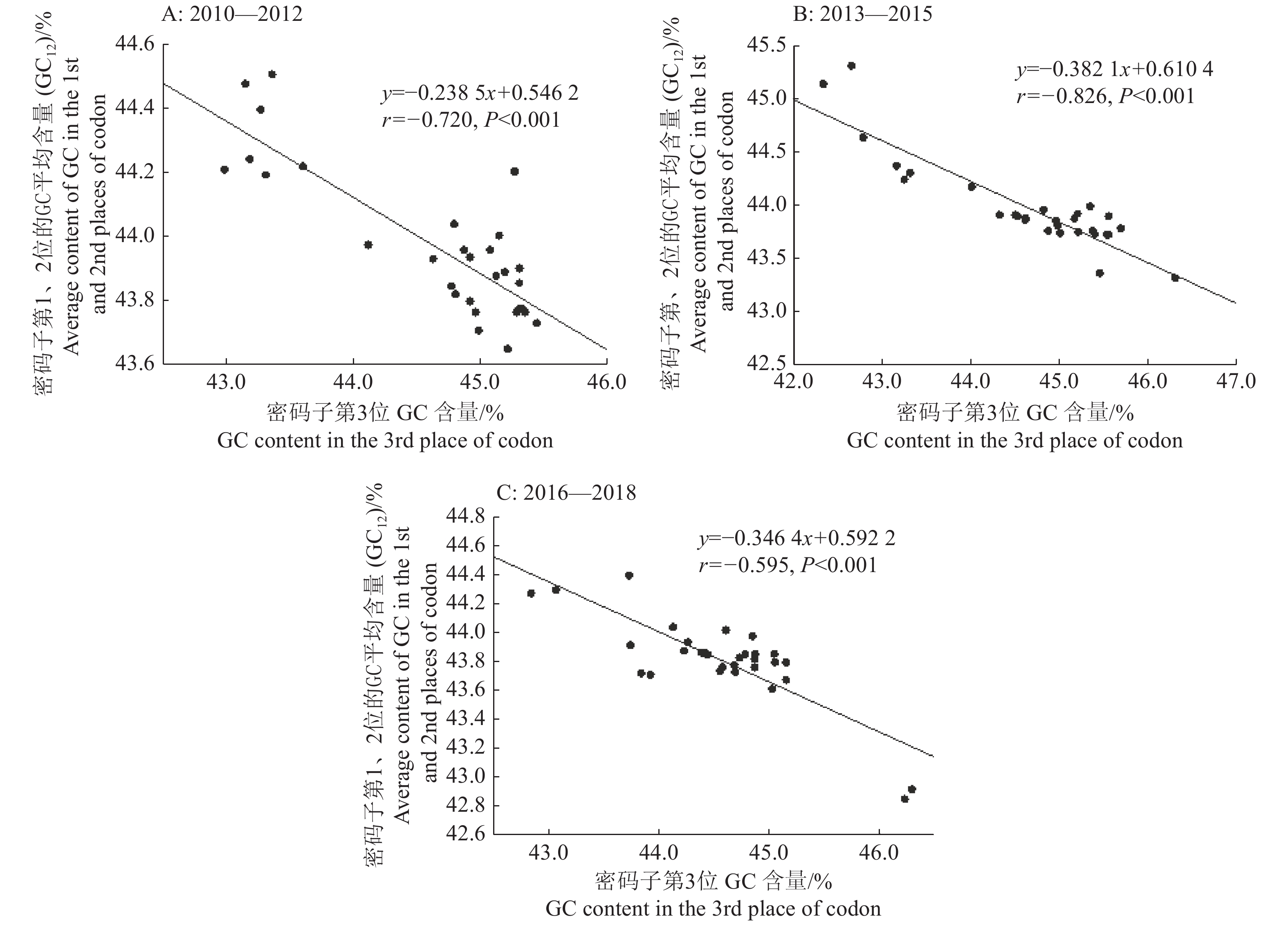
|
图 4 不同年份H9N2禽流感病毒的中性绘图分析 Fig. 4 Neutrality plot analysis of H9N2 avian influenza virus in different years |
H9N2 AIV全基因组的碱基组成特性和最优密码子结果显示,基因组中AU含量较多且偏好使用以A或U为末位碱基的同义密码子,这提示密码子使用偏好性可能对H9N2 AIV的进化产生影响。ENC与密码子使用偏好性呈负相关,密码子使用偏好性大小可通过ENC值来进行评估,本研究发现H9N2 AIV的ENC平均值为52.86,说明不同序列间的基因组组成保守且相对稳定,H9N2 AIV的密码子使用偏好性较低,这与其他A型流感病毒和一些RNA病毒相似,如H3N8(ENC=52.09)[14]、H5N1(ENC=50.91)[15]、H1N1pdm(ENC=52.5)[16]、H3N2(ENC=53.05)[3]、寨卡病毒(ENC=53.93)[17]和马尔堡病毒(ENC=54.2)[5]。低密码子使用偏好性病毒与宿主的合成竞争较低,可在与其密码子使用模式不相同的宿主细胞内进行有效的复制,从而在宿主体内维持其存活周期[5, 16]。密码子使用偏好性可能会受到突变压力、自然选择、基因长度、GC含量、基因表达水平和RNA结构等因素的影响,其中突变压力和自然选择为主要影响因素[18-19]。
为了确定突变压力对密码子使用偏好性的作用,对应性分析的第1、2轴与碱基组成指数的相关性分析结果显示第1、2轴与大部分碱基组成指数呈显著性相关,提示碱基组成对密码子使用偏好性产生影响,即突变压力为H9N2 AIV密码子使用偏好性的主要影响因素之一。但当病毒试图适应宿主细胞时,密码子使用偏好性往往会受到自然选择而降低[19]。对应性分析的第1、2轴与总平均亲水性、芳香性之间均呈现显著相关性,这说明氨基酸的疏水性和芳香性均显著影响着密码子使用偏好性,自然选择作用于H9N2 AIV的密码子使用偏好性。PR2绘图分析发现基因组对AU的使用度大于GC,揭示H9N2 AIV密码子使用偏好性可能由突变压力和其他因素(如自然选择)共同决定,但两者作用不相同。综合对应性分析、PR2绘图和相关性分析结果,可得出结论:突变压力和自然选择为主要影响因素,共同作用于H9N2 AIV密码子使用偏好性;两者作用的相对大小仍为未知。
为了衡量突变压力和自然选择对密码子使用偏好性作用的相对大小,ENC绘图分析发现所有基因组均聚集于标准曲线的下方,提示突变压力并非是H9N2 AIV密码子使用偏好性的主导因素,而是其他影响因素(如自然选择)起主导作用。但有报道[11]指出突变压力和自然选择可能影响ENC,使其不能有效评估突变压力和自然选择对密码子使用偏好性的相对作用;此外,基因组为AU偏向的物种,其密码子使用偏好性将与基因组为GC偏向的存在差异,因此ENC绘图分析可能具有误导性。
密码子适应指数是研究自然选择影响密码子使用偏好性的不二选择,可用于评估病毒对宿主的适应性,所以利用ENC和密码子适应指数之间的相关性来研究突变压力和自然选择对密码子使用偏好性的影响[20]。H9N2 AIV对鸡和人的密码子适应指数平均值偏高,提示H9N2 AIV已适应鸡和人的密码子使用模式,能够更好地利用宿主细胞资源进行病毒复制和繁殖,有利于病毒在宿主内存活和进化;而且H9N2 AIV可能会发展为对人类有致病性的病毒,已有多例H9N2 AIV感染人类事件发生[2, 21],随着H9N2 AIV逐渐适应人类密码子使用模式,对人类的致病性将逐渐增强,因此需要密切监测密码子适应指数变化。同时,密码子适应指数与ENC相关性分析发现大部分密码子适应指数与ENC均呈显著负相关且r≠−1,这说明自然选择对密码子使用偏好性的作用大于突变压力,但突变压力的作用也不能忽视。
进行中性绘图分析以揭示两者作用比例,从GC12含量与GC3含量的线性回归分析发现,突变压力对H9N2禽流感病毒密码子使用偏好性的作用分别为23.85%(2010—2012年)、38.21%(2013—2015年)和34.64%(2016—2018年),而自然选择对密码子使用偏好性的作用较大,分别为76.15%(2010—2012年)、61.79%(2013—2015年)和65.36%(2016—2018年)。中性绘图分析、ENC绘图分析、密码子适应指数及其相关性结果揭示,在H9N2 AIV密码子使用偏好性中,自然选择为主导作用(所占比例为61.79%~76.15%),而突变压力为次要作用(所占比例为23.85%~38.21%)。与H9N2同为A型流感病毒的H3N2[3]和H3N8[22]的密码子使用偏好性也是以自然选择为主要影响因素。
综上所述,H9N2 AIV全基因组为AU偏向且最优密码子多以A或U结尾,而且H9N2 AIV基因组的密码子使用偏好性较低,主要受到突变压力和自然选择的共同作用,其中自然选择为主导作用(所占比例为61.79%~76.15%),而突变压力为次要作用(所占比例为23.85%~38.21%)。H9N2 AIV已适应人类的密码子使用模式,可能对人类有致病性且致病性在逐渐增强。
| [1] |
陈伯伦, 张泽纪, 陈伟斌. 鸡A型禽流感病毒的分离与血清学初步鉴定[J]. 中国家禽, 1997(11): 4-6. (  0) 0) |
| [2] |
SUN Y, LIU J. H9N2 influenza virus in China: A cause of concern[J]. Protein Cell, 2015, 6(1): 18-25. DOI:10.1007/s13238-014-0111-7 (  0) 0) |
| [3] |
LI G, WANG R, ZHANG C, et al. Genetic and evolutionary analysis of emerging H3N2 canine influenza virus[J]. Emerg Microbes Infect, 2018: 7. doi: 10.1038/s41426-018-0079-0.
(  0) 0) |
| [4] |
胡明达. 禽流感病毒进化与传播规律研究[D]. 北京: 军事科学院, 2018.
(  0) 0) |
| [5] |
NASRULLAH I, BUTT A M, TAHIR S, et al. Genomic analysis of codon usage shows influence of mutation pressure, natural selection, and host features on Marburg virus evolution[J]. BMC Evol Biol, 2015: 15. doi: 10.1186/s12862-015-0456-4.
(  0) 0) |
| [6] |
BAKER S F, NOGALES A, MARTÍNEZ-SOBRIDO L. Downregulating viral gene expression: Codon usage bias manipulation for the generation of novel influenza A virus vaccines[J]. Future Virol, 2015, 10(6): 715-730. DOI:10.2217/fvl.15.31 (  0) 0) |
| [7] |
PUIGBÒ P, BRAVO I G, GARCIA-VALLVE S. CAIcal: A combined set of tools to assess codon usage adaptation[J]. Biol Direct, 2008: 3. doi: 10.1186/1745-6150-3-38.
(  0) 0) |
| [8] |
NAKAMURA Y, GOJOBORI T, IKEMURA T. Codon usage tabulated from international DNA sequence databases: Status for the year 2000[J]. Nucleic Acids Res, 2000, 28(1): 292. DOI:10.1093/nar/28.1.292 (  0) 0) |
| [9] |
WANG L, XING H, YUAN Y, et al. Genome-wide analysis of codon usage bias in four sequenced cotton species[J]. PLoS One, 2018, 13(3): e0194372. DOI:10.1371/journal.pone.0194372 (  0) 0) |
| [10] |
SHARP P M, LI W H. The codon adaptation index: A measure of directional synonymous codon usage bias, and its potential applications[J]. Nucleic Acids Res, 1987, 15(3): 1281-1295. DOI:10.1093/nar/15.3.1281 (  0) 0) |
| [11] |
CHEN Y, CHEN Y F. Analysis of synonymous codon usage patterns in duck hepatitis A virus: A comparison on the roles of mutual pressure and natural selection[J]. VirusDisease, 2014, 25(3): 285-293. DOI:10.1007/s13337-014-0191-2 (  0) 0) |
| [12] |
CRISTINA J, FAJARDO A, SOÑORA M, et al. A detailed comparative analysis of codon usage bias in Zika virus[J]. Virus Res, 2016, 223: 147-152. DOI:10.1016/j.virusres.2016.06.022 (  0) 0) |
| [13] |
金刚, 王丽萍, 龙凌云, 等. 普通野生稻线粒体蛋白质编码基因密码子使用偏好性的分析[J]. 植物科学学报, 2019, 37(2): 188-197. DOI:10.11913/PSJ.2095-0837.2019.20188 (  0) 0) |
| [14] |
KUMAR N, BERA B C, GREENBAUM B D, et al. Revelation of influencing factors in overall codon usage bias of equine influenza viruses[J]. PLoS One, 2016, 11(4): e0154376. DOI:10.1371/journal.pone.0154376 (  0) 0) |
| [15] |
ZHOU T, GU W J, MA J M, et al. Analysis of synonymous codon usage in H5N1 virus and other influenza A viruses[J]. Biosystems, 2005, 81(1): 77-86. DOI:10.1016/j.biosystems.2005.03.002 (  0) 0) |
| [16] |
ANHLAN D, GRUNDMANN N, MAKALOWSKI W, et al. Origin of the 1918 pandemic H1N1 influenza A virus as studied by codon usage patterns and phylogenetic analysis[J]. RNA, 2011, 17(1): 64-73. DOI:10.1261/rna.2395211 (  0) 0) |
| [17] |
BUTT A M, NASRULLAH I, QAMAR R, et al. Evolution of codon usage in Zika virus genomes is host and vector specific[J]. Emerg Microbes Infect, 2016, 5: e107. (  0) 0) |
| [18] |
YANG X, LUO X, CAI X. Analysis of codon usage pattern in Taenia saginata based on a transcriptome dataset[J]. Parasite Vector, 2014: 7. doi: 10.1186/s13701-014-0527-1.
(  0) 0) |
| [19] |
SHI S L, JIANG Y R, LIU Y Q, et al. Selective pressure dominates the synonymous codon usage in parvoviridae[J]. Virus Genes, 2013, 46(1): 10-19. DOI:10.1007/s11262-012-0818-6 (  0) 0) |
| [20] |
VICARIO S, MORIYAMA E N, POWELL J R. Codon usage in twelve species of Drosophila[J]. BMC Evol Biol, 2007: 7. doi: 10.1186/1471-2148-7-226.
(  0) 0) |
| [21] |
HUANG Y W, LI X D, ZHANG H, et al. Human infection with an avian influenza A (H9N2) virus in the middle region of China[J]. J Med Virol, 2015, 87(10): 1641-1648. DOI:10.1002/jmv.24231 (  0) 0) |
| [22] |
BERA B C, VIRMANI N, KUMAR N, et al. Genetic and codon usage bias analyses of polymerase genes of equine influenza virus and its relation to evolution[J]. BMC Genomics, 2017: 18. doi: 10.1186/s12864-017-4063-1.
(  0) 0) |
 2020, Vol. 41
2020, Vol. 41


