2. 黄埔海关,广东 广州 510730;
3. 重庆海关,重庆 401147
2. Huangpu Customs, Guangzhou 510730, China;
3. Chongqing Customs, Chongqing 401147, China
苹果壳色单隔孢溃疡病(Diplodiacanker)的病原菌为史蒂芬葡萄座腔菌Botryosphaeria stevensii,异名Physalospora mutila,无性态Diplodia mutila[1-2],是我国禁止入境的重要检疫性有害生物[3]。其寄主范围非常广泛,可为害苹果Malus spp.、梨Pyrus spp.、李Prunus spp.、葡萄Vitis spp.、栎Quercus spp.、棕榈Trachycarpus spp.等多达11科18属的重要经济植物[4]。根据国际应用生物科学中心(
准确、快速地进行物种鉴定是防止外来有害生物入侵的首要环节。由于葡萄座腔菌属Botryosphaeria真菌的有性态在自然环境和培养条件下都很难产生,而其无性态的形态特征多有重叠且不稳定,因此该属的形态分类鉴定十分困难[9-11]。通过核糖体DNA序列的分析比较进行种类鉴定已被广泛应用于这一真菌类群[2,12-14]。此外,RAPD[15-16]、ISSR[17-18]、ARDRA[19]、RFLP[20-21]、SSCP[22]、RAMS[23]、MSP-PCR和rep-PCR[24]等DNA指纹图谱技术也被应用于该类真菌的分类研究。针对葡萄座腔菌属真菌特定类群的特异性检测技术也有相关研究[25-29]。但目前国内外鲜见针对苹果壳色单隔孢溃疡病菌进行实时荧光PCR特异性检测的报道。
本研究根据苹果壳色单隔孢溃疡病菌的β微管蛋白(β-tubulin)基因保守序列,设计筛选特异引物及探针,旨在建立针对该病菌的实时荧光PCR检测技术,为口岸的植物检验检疫提供一种灵敏而高效的检测方法。
1 材料与方法 1.1 供试菌株供试菌株包括苹果壳色单隔孢溃疡病菌及其近缘种属、水果上常见的病原菌27株。来源于荷兰微生物菌种保藏中心(Centraalbureau voor Schimmelcultures, CBS)、中国林业微生物保藏管理中心(China Forestry Culture Collection Center, CFCC)和中国农业微生物菌种保藏管理中心(Agricultural Culture Collection of China, ACCC)等标准菌种库,以及海关从入境货物中分离保存的菌种。供试菌株来源、寄主等相关信息见表1。
|
|
表 1 本研究供试菌株的相关信息 Table 1 Information of strains used in the study |
供试菌株接种于PDA平板于24 ℃条件下光暗交替培养7~10 d,待菌落长到直径5~6 cm时,收集菌丝,加液氮研磨后,采用TIANGEN DP320植物基因组DNA提取试剂盒(天根生化科技有限公司,北京)提取基因组DNA,用Nanodrop ND-2000超微量核酸蛋白测定仪(Thermo Fisher Scientific, USA)测定DNA的浓度和纯度,并于−20 ℃条件下保存。
1.3 引物和探针设计基于前期获得的苹果壳色单隔孢溃疡病菌及其近缘种的β微管蛋白基因(β-tubulin基因)序列,以Primer Express 3.0(Thermo Fisher Scientific, USA)和Beacon Designer 7.5(Premier Biosoft Company, Canada)软件辅助设计实时荧光PCR引物和特异性探针,其中,引物BsF1: 5′-CGGACGAGACCTTCTGTATTGAC-3′和BsR1:5′-GCTCGAACCAGCCGACTAAG-3′,探针BsP267:FAM-CGAGGTACGTGAAATT-MGB。引物及探针委托生工生物工程(上海)股份有限公司合成。并将其输入GenBank数据库中运用Primer-BLAST进行序列比对及特异性检验。
1.4 阳性质粒构建将苹果壳色单隔孢溃疡病菌β-tubulin基因靶标序列片段转入含有氨苄卡那霉素(Ampicillin, Amp)抗性的pUC57载体上,得到苹果壳色单隔孢溃疡病菌β-tubulin基因重组阳性质粒,将目标质粒转入大肠埃希菌Escherichia coli DH5α后用甘油在−80 ℃条件下保存。
1.5 特异性测试以苹果壳色单隔孢溃疡病菌及其近缘种标准菌株的DNA为模板,进行实时荧光PCR的特异性检测。PCR试剂均购自东洋纺(上海)生物科技有限公司(TOYOBO)。PCR反应体系为20 μL,包括10 μL 2×Thunderbird Probe qPCR Mix(含1×ROX reference dye),上、下游引物(10 μmol/L)和探针(10 μmol/L)各0.4 μL,1 μL模板DNA,加无菌水补至20 μL。在ABI Step One Plus实时荧光PCR仪上设置反应程序为:95 ℃预变性60 s;95 ℃ 15 s,64 ℃ 36 s,循环40次。以无菌水为空白对照,以循环阈值CT≤35作为阳性标准阈值。
1.6 灵敏度测试及标准曲线的建立以带有苹果壳色单隔孢溃疡病菌β-tubulin基因靶标序列的重组阳性质粒pUC-BsBTU为模板,对TaqMan探针BsP267进行荧光PCR灵敏度测试。对质粒DNA进行系列稀释,得到DNA质量浓度为100 000、10 000、1 000、100、10、1、0.1 fg/μL的系列稀释液,各取1 μL作为模板进行实时荧光PCR扩增,反应体系和程序同“1.5”。以无菌水为空白对照,以CT≤35作为阳性标准阈值。以模板量的对数值与所得CT值建立标准曲线。
2 结果与分析 2.1 阳性质粒的构建将苹果壳色单隔孢溃疡病菌β-tubulin基因包含BsF1、BsP267和BsR1序列的400 bp片段(图1)插入到pUC57载体上,构建苹果壳色单隔孢溃疡病菌β-tubulin基因重组质粒,该阳性质粒命名为pUC-BsBTU。

|
图 1 靶标基因阳性质粒克隆序列 Fig. 1 Cloning sequence of positive plasmid of target gene 阴影部分从左到右依次为BsF1(绿色)、BsP267(黄色)和BsR1(蓝色) The shaded parts with different colours from left to right are BsF1(green), BsP267(yellow) and BsR1(blue), respectively |
采用引物BsF1/BsR1和探针BsP267对苹果壳色单隔孢溃疡病菌及其近缘种标准菌株以及从水果分离的常见病原真菌进行实时荧光PCR检测,结果(图2)显示,探针BsP267分别对供试3株苹果壳色单隔孢溃疡病菌目标菌株表现出特异性阳性扩增,荧光信号明显增加,而非目标菌株以及空白对照都没有检测到荧光信号,表现为阴性。结果表明本研究建立的苹果壳色单隔孢溃疡病菌实时荧光PCR检测方法特异性良好。

|
图 2 探针BsP267对苹果壳色单隔孢溃疡病菌的特异性检测 Fig. 2 Specificity idendification of Botryosphaeria stevensii using probe BsP267 1~3为Botryosphaeria stevensii基因组DNA(CBS 112553、CBS 120834、CQ01);4~27为其他无扩增曲线的样品(CBS112555、CBS112876、PP5.3-2、CFCC51611、CFCC50629、CFCC86567、CFCC84207、PP5.3-6、CFCC86251、CFCC86538、PP5.12-2、ACCC37263、CBS121886、CBS121887、CBS120835、CBS121104、CBS124253、CBS124130、CBS124462、CBS115176、11-40460、PG5.31、16-4966-2、15-128331),28为空白对照(无菌水) 1−3: Cultured mycelial DNA of Botryosphaeria stevensii(CBS 112553, CBS 120834, CQ01);4−27: Negative control (CBS112555, CBS112876, PP5.3-2, CFCC51611, CFCC50629, CFCC86567, CFCC84207, PP5.3-6, CFCC86251, CFCC86538, PP5.12-2, ACCC37263, CBS121886, CBS121887, CBS120835, CBS121104, CBS124253, CBS124130, CBS124462, CBS115176, 11-40460, PG5.31, 16-4966-2, 15-128331), 28: Blank control (ddH2O) |
采用探针BsP267对苹果壳色单隔孢溃疡病菌菌株的重组阳性质粒DNA不同稀释度模板进行实时荧光PCR检测。结果(图3)显示:质量浓度为100 000、10 000、1 000、100、10、1 fg/μL的阳性质粒DNA都能检测到明显的荧光信号,表现为阳性扩增;0.1 fg/μL的阳性质粒DNA的CT值为37.01,超过了35这一阳性标准阈值,这表明BsP267探针对苹果壳色单隔孢溃疡病菌阳性质粒DNA的检测灵敏度可达到1 fg/μL。
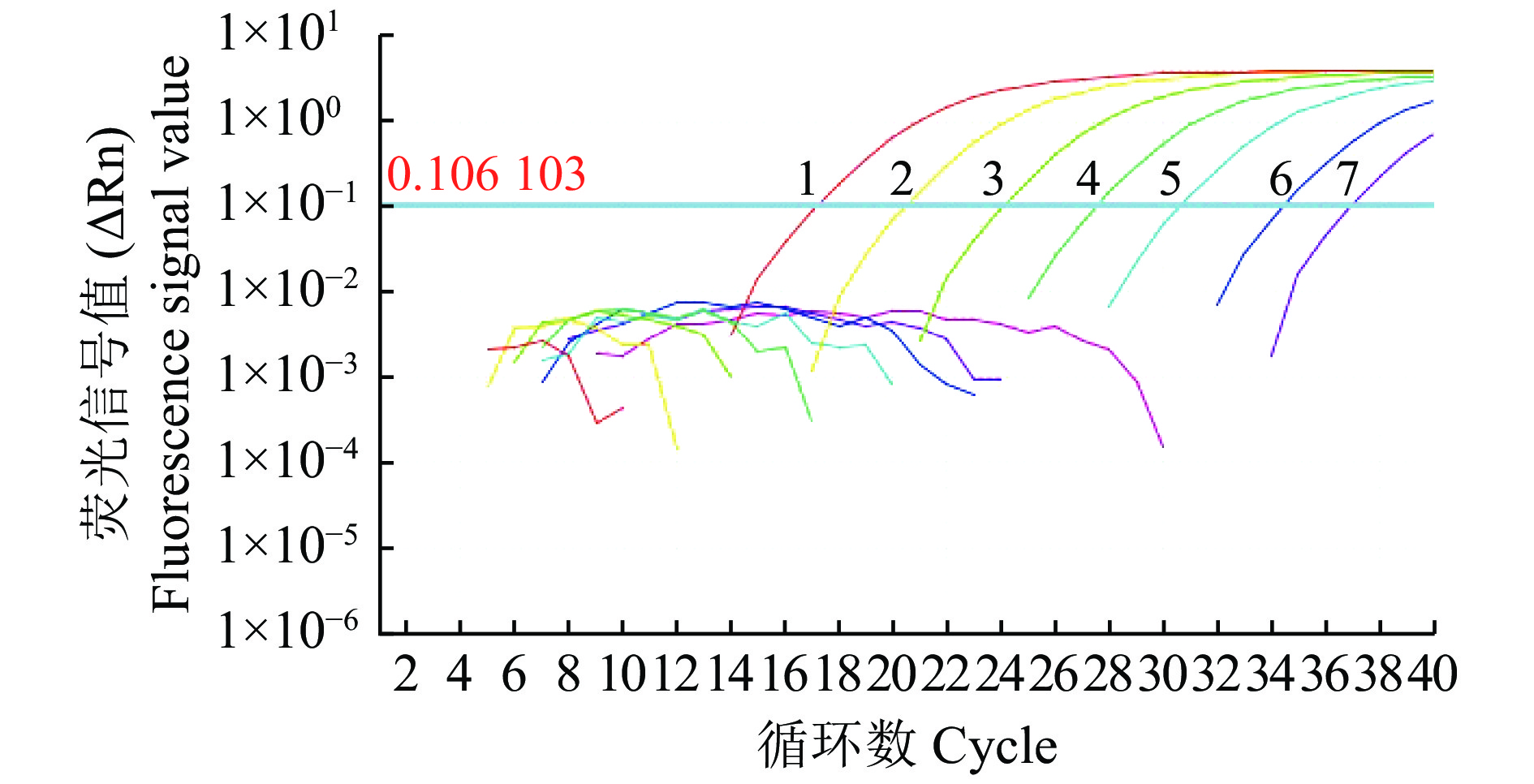
|
图 3 探针BsP267对阳性质粒DNA的灵敏度检测 Fig. 3 Sensitivity idendification of positive plasmid DNA using probe BsP267 1~7分别为100 000、10 000、1 000、100、10、1、0.1 fg质粒DNA 1−7 are 100 000、10 000、1 000、100、10、1、0.1 fg plasmid DNA, respectively |
将苹果壳色单隔孢溃疡病菌菌株的重组阳性质粒DNA,分别稀释至100 000、10 000、1 000、100、10、1、0.1 fg/μL 7个质量浓度梯度后,荧光定量PCR并构建标准曲线。如图4所示,CT值为17.37~37.01,线性回归方程为:y=−3.189lgx+36.692,决定系数(R2)为0.993(参考值>0.95),扩增效率为 105.858%(参考值范围为90%~110%),结果表明其线性相关性良好、扩增效率高、满足可信度要求,该方法可应用于苹果壳色单隔孢溃疡病菌的特异性检测。
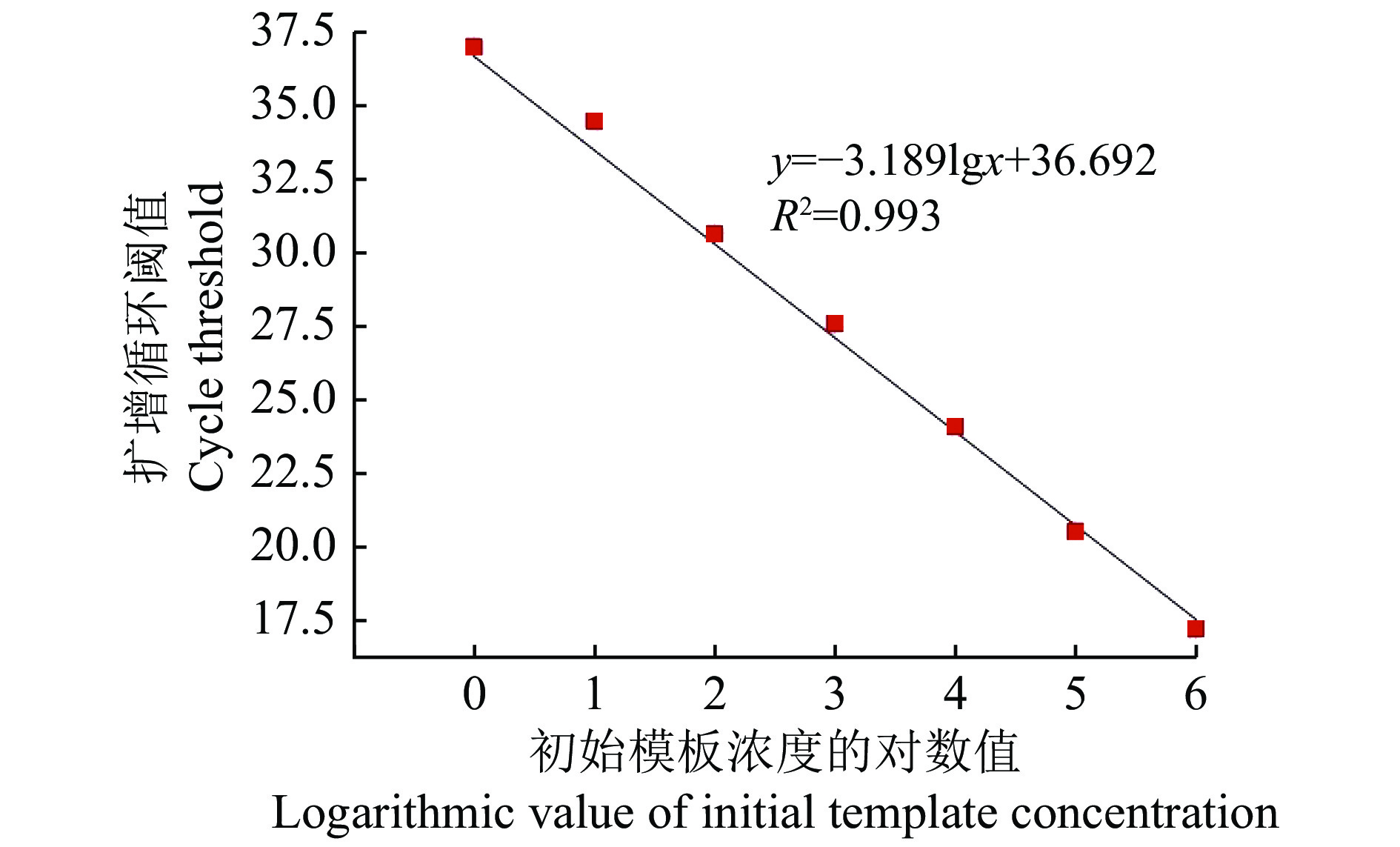
|
图 4 实时荧光PCR标准曲线 Fig. 4 Real-time fluorescent PCR standard curve |
开发适用于口岸植物检疫的快速诊断技术,对苹果壳色单隔孢溃疡病菌进行严格检疫,对防止该类病原菌的传入至关重要。前期研究中,对苹果壳色单隔孢溃疡病菌的翻译延长因子基因(EF-1α)、核糖体内转录间隔区基因(ITS)、RNA聚合酶II第2亚基基因(RPB2)、谷氨酰胺合成酶基因(GS)和核糖体大亚基(LSU)等基因序列进行了特异性引物和探针筛选,最后基于特异性和扩增效率,确定了β-tubulin基因作为苹果壳色单隔孢溃疡病菌特异性检测的靶标基因。β-tubulin基因在细胞生长过程中有着重要作用,其序列具有高度的保守性,具有管家基因的特性[30]。此外,该基因既有保守的外显子又有许多可变的内含子,在不同属、种间蕴含着丰富的变异,常作为标识片段用于真菌的物种鉴定[31-35]。
本研究使用TaqMan MGB探针实时荧光PCR检测壳色单隔孢溃疡病菌。通过大量文献分析及对前期基础研究的总结,选择苹果壳色单隔孢溃疡病菌特异性高、保守性强的β-tubulin基因作为检测靶标。引物BsF1/BsR1和探针BsP267组合对苹果壳色单隔孢溃疡病菌及其近缘种标准菌株以及从水果分离的常见病原真菌的实时荧光PCR检测结果显示,探针BsP267对供试阳性菌株均表现出特异性扩增,扩增效率为105.858%,最低检测DNA质量浓度下限为1 fg/μL;而非目标菌株以及空白对照都没有检测到特异性扩增。结果表明本研究建立的苹果壳色单隔孢溃疡病菌实时荧光PCR检测方法特异性良好,所设计的引物BsF1/BsR1和特异性探针BsP267可对未知样品进行壳色单隔孢溃疡病菌的特异检测。引物和探针的设计在遵循荧光PCR探针设计原则的基础上,将包含更多的种特异性位点作为最重要的考量,同时探针引入了小沟结合物(Minor groove binder, MGB)基团以确保其高特异性。基于TaqMan MGB探针技术的实时荧光PCR方法因为探针序列短,具有分辨率高、稳定性好、背景低、重复性好等优点。实时荧光PCR比常规PCR(包括巢式PCR和多重PCR)具有更高的检测灵敏度,其他针对葡萄座腔菌属真菌特定类群的特异性检测研究也印证了这一点[25-29]。
本研究建立了苹果壳色单隔孢溃疡病菌的TaqMan MGB探针实时荧光PCR检测方法。该方法特异性强、检测灵敏度高,可以提高苹果壳色单隔孢溃疡病菌的检出率和检测速度。既可用于苹果壳色单隔孢溃疡病菌的快速确诊,也可用于进境截获的可疑样品及未知苹果病果样品的快速筛查,在口岸植物检疫中具有很强的可操作性和适用性。该检测方法的建立可以防止苹果检疫性病原真菌的传入和传播,同时也可为进口水果的快速通关提供有效的技术支持。
| [1] |
VAJNA L. Branch canker and dieback of sessile oak (Quercus petraea) in Hungary caused by Diplodia mutila: I: Identification of the pathogen
[J]. European Journal of Forest Pathology, 1986, 16(4): 223-229. (  0) 0) |
| [2] |
ALVES A, CORREIA A, LUQUE J, et al. Botryosphaeria corticola, sp. nov. on Quercus species, with notes and description of Botryosphaeria stevensii and its anamorph, Diplodia mutila
[J]. Mycologia, 2004, 96(3): 598-613. DOI:10.1080/15572536.2005.11832956 (  0) 0) |
| [3] |
中华人民共和国农业部, 国家质量监督检验检疫总局. 中华人民共和国进境植物检疫性有害生物名录[M]. 北京: 中华人民共和国农业部. 2007.
(  0) 0) |
| [4] |
王卫芳, 张秋娥, 黄英, 等. 苹果壳色单隔孢溃疡病菌检疫鉴定方法: SN 3750—2013[S]. 北京: 中国标准出版社, 2013: 1-9.
(  0) 0) |
| [5] |
张秋娥, 王卫芳, 严进, 等. 苹果壳色单隔孢溃疡病菌[J]. 植物检疫, 2012, 26(1): 43-46. (  0) 0) |
| [6] |
PRZYBYŁ K. Fungi associated with necrotic apical parts of Fraxinus excelsior shoots
[J]. Forest Pathology, 2010, 32(6): 387-394. (  0) 0) |
| [7] |
TIBERI R, RAGAZZI A. Association between fungi and xylophagous insects of declining oaks in Italy[J]. Redia-giornale Di Zoologia, 1998, 81: 83-91. (  0) 0) |
| [8] |
张露茜, 宋绍祎, 焦彬彬, 等. 进境水曲柳原木中苹果壳色单隔孢溃疡病菌的检疫鉴定[J]. 植物检疫, 2015, 29(1): 25-29. (  0) 0) |
| [9] |
LAUNDON G F. Botryosphaeria obtusa, B. stevensii, and Otthia spiraeae in New Zealand
[J]. Transactions of the British Mycological Society, 1973, 61(2): 369-374. DOI:10.1016/S0007-1536(73)80158-5 (  0) 0) |
| [10] |
VAN NIEKERK J M, CROUS P W, GROENEWALD J Z, et al. DNA phylogeny, morphology and pathogenicity of Botryosphaeria species on grapevines
[J]. Mycologia, 2004, 96(4): 781-798. DOI:10.1080/15572536.2005.11832926 (  0) 0) |
| [11] |
SLIPPERS B, JOHNSON G I, CROUS P W, et al. Phylogenetic and morphological reevaluation of the Botryosphaeria species causing diseases of Mangifera indica
[J]. Mycologia, 2005, 97(1): 99-110. DOI:10.1080/15572536.2006.11832843 (  0) 0) |
| [12] |
ZHOU S, STANOSZ G R. Relationships among Botryosphaeria species and associated anamorphic fungi inferred from the analyses of ITS and 5.8s rDNA sequences
[J]. Mycologia, 2001, 93(3): 516-527. DOI:10.1080/00275514.2001.12063184 (  0) 0) |
| [13] |
TAYLOR A, HARDY G E ST J, WOOD P, et al. Identification and pathogenicity of Botryosphaeria species associated with grapevine decline in Western Australia
[J]. Australasian Plant Pathology, 2005, 34(2): 187-195. DOI:10.1071/AP05018 (  0) 0) |
| [14] |
CROUS P W, SLIPPERS B, WINGFIELD M J, et al. Phylogenetic lineages in the Botryosphaeriaceae[J]. Studies in Mycology, 2006, 55: 235-253. DOI:10.3114/sim.55.1.235 (  0) 0) |
| [15] |
STANOSZ G R, SWART W J, SMITH D R. Similarity between fungi identified as Diplodia pinea f. sp. cupressi in Israel and Botryosphaeria stevensii or Diplodia mutila on Juniperus in the United States
[J]. European Journal of Forest Pathology, 1998, 28(1): 33-42. DOI:10.1111/j.1439-0329.1998.tb01163.x (  0) 0) |
| [16] |
黄春燕, 刘开启. 苹果轮纹病及相关病害病原菌的RAPD分析[J]. 植物病理学报, 2001, 31(2): 164-169. DOI:10.3321/j.issn:0412-0914.2001.02.012 (  0) 0) |
| [17] |
ZHOU S, SMITH D R, STANOSZ G R. Differentiation of Botryosphaeria species and related anamorphic fungi using inter simple or short sequence repeat (ISSR) finger printing
[J]. Mycological Research, 2001, 105(8): 919-926. DOI:10.1016/S0953-7562(08)61947-4 (  0) 0) |
| [18] |
赵嘉平. 树木溃疡病菌: 葡萄座腔菌属及相关真菌系统分类研究[D]. 北京: 中国林业科学研究院, 2007.
(  0) 0) |
| [19] |
ALVES A, PHILLIPS A J L, HENRIQUES I, et al. Evaluation of amplified ribosomal DNA restriction analysis as a method for the identification of Botryosphaeria species
[J]. FEMS Microbiology Letters, 2005, 245(2): 221-229. DOI:10.1016/j.femsle.2005.03.005 (  0) 0) |
| [20] |
余仲东, 曹支敏, 张星耀. 杨树溃疡病、苹果轮纹病等病原菌的ITS-rDNA-RFLP解析[J]. 中国森林病虫, 2004, 23(2): 15-18. DOI:10.3969/j.issn.1671-0886.2004.02.005 (  0) 0) |
| [21] |
SLIPPERS B, SMIT W A, CROUS P W, et al. Taxonomy, phylogeny and identification of Botryosphaeriaceae associated with pome and stone fruit trees in South Africa and other regions of the world[J]. Plant Pathology, 2007, 56(1): 128-139. (  0) 0) |
| [22] |
RIDGWAY H J, AMPONSAH N T, BROWN D S, et al. Detection of botryosphaeriaceous species in environmental samples using a multi-species primer pair[J]. Plant Pathology, 2011, 60(6): 1118-1127. DOI:10.1111/j.1365-3059.2011.02474.x (  0) 0) |
| [23] |
王璠. 桃流胶病菌Botryosphaeria spp. 鉴定、分布、遗传多样性及PCR快速检测技术研究[D]. 武汉: 华中农业大学, 2012.
(  0) 0) |
| [24] |
ALVES A, PHILLIPS A J L, HENRIQUES I, et al. Rapid differentiation of species of Botryosphaeriaceae by PCR fingerprinting[J]. Research in Microbiology, 2007, 158(2): 112-121. DOI:10.1016/j.resmic.2006.10.003 (  0) 0) |
| [25] |
MA Z, MICHAILIDES T J. A PCR-based technique for identification of Fusicoccum sp. from pistachio and various other hosts in California
[J]. Plant Disease, 2002, 86(5): 515-520. DOI:10.1094/PDIS.2002.86.5.515 (  0) 0) |
| [26] |
LUCHI N, PRATESI N, SIMI L, et al. High-resolution melting analysis: A new molecular approach for the early detection of Diplodia pinea in Austrian pine
[J]. Fungal Biology, 2011, 115(8): 715-723. DOI:10.1016/j.funbio.2011.05.005 (  0) 0) |
| [27] |
SPAGNOLO A, MARCHI G, PEDUTO F, et al. Detection of Botryosphaeriaceae species within grapevine woody tissues by nested PCR, with particular emphasis on the Neofusicoccum parvum/N. ribiscom plex
[J]. European Journal of Plant Pathology, 2011, 129(3): 485-500. DOI:10.1007/s10658-010-9715-9 (  0) 0) |
| [28] |
NI H F, YANG H R, CHEN R S, et al. A nested multiplex PCR for species-specific identification and detection of Botryosphaeriaceae species on mango[J]. European Journal of Plant Pathology, 2012, 133(4): 819-828. DOI:10.1007/s10658-012-0003-8 (  0) 0) |
| [29] |
XU C, ZHANG H, CHI F, et al. Species-specific PCR-based assays for identification and detection of Botryosphaeriaceae species causing stem blight on blueberry in China[J]. Journal of Integrative Agriculture, 2016, 15(3): 573-579. DOI:10.1016/S2095-3119(15)61177-7 (  0) 0) |
| [30] |
SHEIR-NEISS G, LAI M H, MORRIS N R. Identification of a gene for β-tubulin in Aspergillus nidulans
[J]. Cell, 1978, 15(2): 639-647. DOI:10.1016/0092-8674(78)90032-6 (  0) 0) |
| [31] |
MOSTERT L, GROENEWALD J Z, SUMMERBELL R C, et al. Taxonomy and pathology of Togninia (Diaporthales) and its Phaeoacremonium anamorphs
[J]. Studies in Mycology, 2006, 54: 1-113. DOI:10.3114/sim.54.1.1 (  0) 0) |
| [32] |
BILODEAU G J, LE´VESQUE C A, DE COCK A W A M, et al. Molecular detection of Phytophthora ramorum by real-time polymerase chain reaction using TaqMan, SYBR Green, and molecular beacons
[J]. Phytopathology, 2007, 97(5): 632-642. DOI:10.1094/PHYTO-97-5-0632 (  0) 0) |
| [33] |
AROCA A, RAPOSO R, LUNELLO P. A biomarker for the identification of four Phaeoacremonium species using the β-tubulin gene as the target sequence
[J]. Applied Microbiology and Biotechnology, 2008, 80(6): 1131-1140. DOI:10.1007/s00253-008-1647-3 (  0) 0) |
| [34] |
ZAMPIERI E, MELLO A, BONFANTE P, et al. PCR primers specific for the genus Tuber reveal the presence of several truffle species in a truffle-ground
[J]. FEMS Microbiology Letters, 2009, 297(1): 67-72. DOI:10.1111/j.1574-6968.2009.01655.x (  0) 0) |
| [35] |
GUO Y, LI W, SUN H, et al. Detection and quantification of Rhizoctonia cerealis in soil using real-time PCR
[J]. Journal of General Plant Pathology, 2012, 78(4): 247-254. DOI:10.1007/s10327-012-0390-x (  0) 0) |
 2021, Vol. 42
2021, Vol. 42


