2. 福建农林大学 农学院,福建 福州 350002;
3. 福建省种子总站,福建 福州 350003;
4. 福建农林大学 生命科学学院,福建 福州 350002;
5. 福建省特种作物育种与利用工程技术研究中心,福建 福州 350002;
6. 福建农林大学 教育部作物遗传育种与综合利用重点实验室,福建 福州 350002
2. College of Agriculture, Fujian Agriculture and Forestry University, Fuzhou 350002, China;
3. Seed Centre of Fujian, Fuzhou 350003, China;
4. College of Life Science, Fujian Agriculture and Forestry University, Fuzhou 350002, China;
5. Fujian Engineering and Technology Research Center of Breeding and Utilization for Special Crop, Fuzhou 350002, China;
6. Key Laboratory of Genetics, Breeding and Multiple Application of Crops, Ministry of Education, Fujian Agriculture and Forestry University, Fuzhou 350002, China
淀粉是植物贮藏器官(块根、块茎)以及种子的主要营养成分之一,利用淀粉酶将储藏的淀粉分解为寡糖或单糖,可为植物的生长发育提供碳源和能源[1]。根据淀粉酶水解淀粉方式的不同,可分为α−淀粉酶和β−淀粉酶。其中,β−淀粉酶是广泛存在于植物和少数微生物中的一种外切水解酶,只能从淀粉侧链的非还原性末端开始,以麦芽糖为单位切割α−1,4−糖苷键,产生β型麦芽糖和限制性糊精[2]。β−淀粉酶作为自然界中将淀粉降解为麦芽糖的主要作用酶,广泛应用于啤酒、饴糖以及饮料等工业生产中,是食品加工和酿造行业的重要酶源[3]。
甘薯Ipomoea batatas是我国重要的农作物,具有适应能力强、种植面积广以及营养价值高等特点。有研究表明,β−淀粉酶是甘薯块根中的一种蛋白质成分,含量仅次于甘薯贮藏蛋白,约占根可溶性蛋白的5%[4]。除此之外,甘薯在蒸煮过程中所产生的香气也部分来源于β−淀粉酶水解淀粉所产生的麦芽糖,且β−淀粉酶含量高的甘薯的甜度也明显高于含量低的甘薯[5]。近年来甘薯β−淀粉酶的研究陆续报道,例如梁新红等[6]分析甘薯中β−淀粉酶的酶解条件时发现,甘薯β−淀粉酶酶解产物主要为麦芽糖,而麦芽糖可以诱导枯草芽孢杆菌发酵产生中温α−淀粉酶;陈显让等[7]对甘薯块根膨大后期β−淀粉酶活性进行测定,发现在薯块膨大后期β−淀粉酶活性多呈下降趋势,且不同品系间的酶活性差异较大。甘薯块根中的β−淀粉酶虽然很早就被分离纯化,对其晶体结构也进行了较为深入的研究[8],但是对甘薯β−淀粉酶基因家族序列分析和β−淀粉酶基因在甘薯生长发育过程中发挥的生物学功能作用的研究还较少。
2017年中国科学院上海辰山植物科学研究中心和植物生理生态研究所联合德国马克斯普朗克分子遗传研究所和分子植物生理研究所,利用新型单倍型基因组装配方法对甘薯品种‘泰中6号’进行全基因组测序,获得了首个甘薯栽培种的全基因组测序结果[9]。本研究以此测序数据为基础,从中挖掘出甘薯β−淀粉酶基因家族序列,分析它们的序列结构域特征、系统进化关系以及低温贮藏期间基因表达量的变化情况,为进一步研究甘薯β−淀粉酶基因的功能和进化奠定基础。
1 材料与方法 1.1 材料以甘薯‘泰中6号’全基因组测序数据为研究对象,利用snap程序检索甘薯15条染色体序列的CDS区,得到178 458个甘薯基因序列[10]。基因组数据来源于NCBI数据库(
以从TAIR数据库(
从甘薯全基因组CDS序列的ZFF文件中提取甘薯β−淀粉酶基因序列的染色体位置信息,利用circos[15]绘制甘薯β−淀粉酶基因家族染色体分布图。为研究甘薯β−淀粉酶基因序列之间的联系,利用blastn查找出其中具有潜在重复关系的基因对[16],并在图中用线条进行连接。
1.4 甘薯β−淀粉酶蛋白序列保守基序查找以甘薯β−淀粉酶家族成员蛋白序列为基础,利用MEME[17]检索其中的保守基序,设置检索数目为10,其他参数保持默认值。检索得到的保守基序氨基酸序列提交至SMART网站(
从Uniprot数据库(
Ji等[19]把‘徐薯15-1’和‘徐薯15-4’在4 ℃条件下贮藏0、2、6周后,取其薯块进行转录组测序。本研究从EBI数据库(
利用HMM检索甘薯全基因组蛋白序列,得到16个甘薯β−淀粉酶家族候选成员;利用blastp检索得到31个甘薯β−淀粉酶家族候选成员,其中包括HMM检索的全部结果,即2次检索一共得到31个β−淀粉酶候选序列。甘薯β−淀粉酶家族成员的确定主要根据Interproscan和CDD数据库鉴定β−淀粉酶候选序列是否含有Glyco_hydro_14保守结构域,共鉴定到12个序列含有该保守结构域。Totsuka等[22]分析了不同物种中的β−淀粉酶氨基酸序列特征,发现3个高度保守的区域(Region I、II和III),本研究将甘薯的12个β−淀粉酶家族成员的氨基酸序列进行多重比对,同样定位到了类似的3个保守区域,结果如图1所示。图1中各保守区域内均有个别序列出现该区域缺失,这可能是由于基因组测序质量的限制[23]。为了更全面地分析甘薯β−淀粉酶基因家族,本研究将这些基因一并归入甘薯β−淀粉酶基因家族中进行分析,甘薯β−淀粉酶基因序列信息如表1所示。
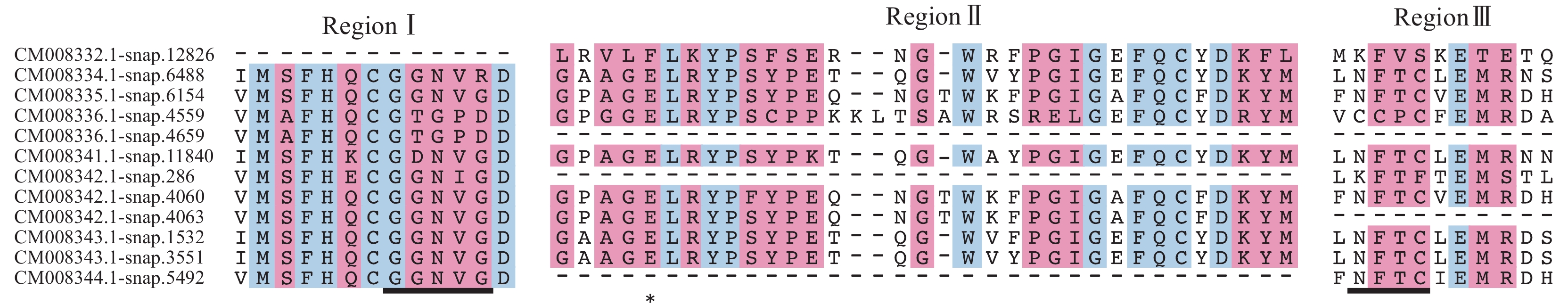
|
图 1 甘薯β−淀粉酶保守结构域分析 Fig. 1 Analysis of the conserved domains of β-amylase in Ipomoea batatas 蓝色阴影部分表示该位点氨基酸保守性为100%,红色阴影部分表示该位点氨基酸保守性在70%以上;统计时各区域内由空位所代替的序列不纳入计算;Region I画横线部分表示flexible loop,Region II“*”标注处表示催化活性位点,Region III画横线部分表示Inner loop The blue shaded part indicates that the amino acid conservation of the site is 100%, and the red shaded part indicates that the amino acid conservation of the site is over 70%; The series replaced by vacancies in each region are excluded in the calculation; The horizontal line in Region I represents the Flexible loop, the “*” in Region II shows the catalytic active site, and the horizontal line in Region III denotes the Inner loop |
|
|
表 1 甘薯β−淀粉酶基因序列信息1) Table 1 The sequence information of β-amylase gene in Ipomoea batatas |
3个保守区域内均含有多个保守性较高的位点(即氨基酸保守性在70%以上),Region I中有12个,Region II中有25个,Region III中有6个。比较3个区域之间的氨基酸保守性,Region I中的氨基酸组成显示出更高的保守性,该区域总共有13个氨基酸位点,有6个位点的氨基酸组成高度保守,其中还含有1个Flexible loop(画横线部分),共有序列的氨基酸组成为GGNVG。Region II和Region III的区域保守性虽然没有Region I高,但在Region II中含有与β−淀粉酶催化活性高度相关的谷氨酸(Glu,E)催化位点[24-25],即“*”标注处;Region III中则含有一个Inner loop(画横线部分),共有序列的氨基酸序列组成为NFTC。
2.2 甘薯β−淀粉酶基因家族在染色体上的分布鉴定得到的12个甘薯β−淀粉酶基因家族成员分布在第2、4、5、6、11、12、13和14号染色体上(图2)。研究表明,2个DNA序列比对后相似性(Similarity)大于75%且比对覆盖区域超过较长序列75%的基因之间存在潜在重复关系[16]。本研究从甘薯β−淀粉酶基因家族中鉴定得到8个具有潜在重复关系的基因对,并在图中用红色线条进行连接。已知8个具有潜在重复关系的基因对中,2对位于同一染色体内,分别为第12号染色体上的CM008342.1-snap.4060和CM008342.1-snap.4063以及第13号染色体上的CM008343.1-snap.1532和CM008343.1-snap.3551;第12号染色体上的基因对在染色体上紧密排列,形成一个基因簇。处于染色体间的潜在重复基因有6对,其中第4号染色体上的CM008334.1-snap.6488分别与第11号染色体上的CM008341.1-snap.11840以及第13号染色体上的CM008343.1-snap.1532和CM008343.1-snap.3551构成3个基因对,而CM008341.1-snap.11840又与CM008343.1-snap.1532基因形成1个潜在重复基因对。除此之外,第5号染色体上的基因和第12号染色体上的2个基因之间也有2个具有潜在重复关系的基因对。
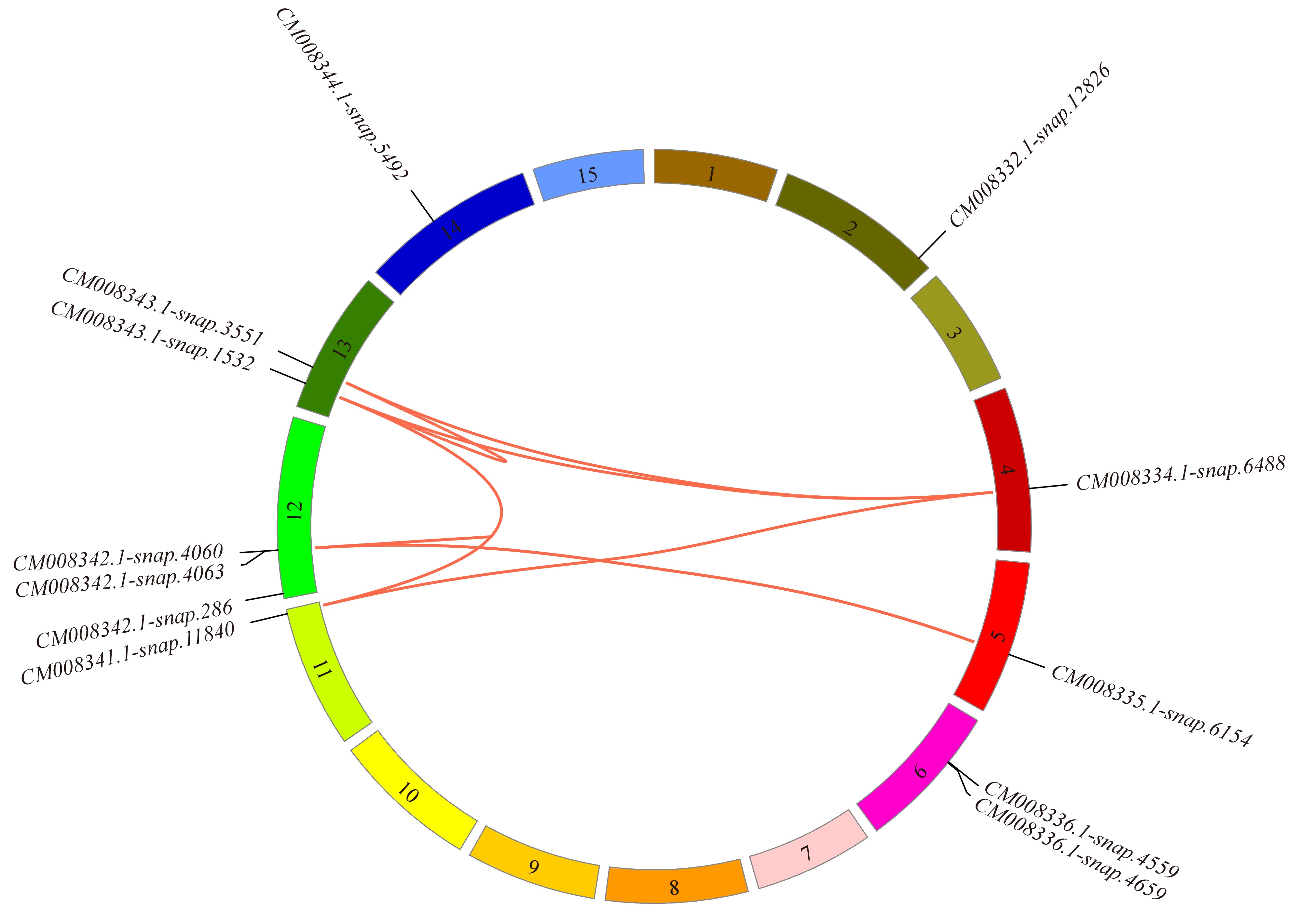
|
图 2 甘薯β−淀粉酶基因家族染色体定位及潜在重复关系分析 Fig. 2 Chromosomal localization and potential duplication relationship of the β-amylase gene family in Ipomoea batatas |
利用MEME从甘薯β−淀粉酶氨基酸序列中检索到10个保守基序(图3),各基序排列顺序基本遵循如下规律:Motif 8—Motif 7—Motif 1—Motif 3—Motif 6—Motif 4—Motif 5—Motif 2—Motif 10—Motif 9。从图3可以发现,甘薯β−淀粉酶各成员的motif组成数量有差异,如CM008335.1-snap.6154、CM008336.1-snap.4559、CM008334.1-snap.11840和CM008342.1-snap.4060 4个成员含有完整的10个motif;CM008336.1-snap.4659和CM008332.1-snap.12826中所含的motif数则较少,分别仅有3个和4个。每个motif在同一个序列中最多仅出现1次,不同motif在甘薯β−淀粉酶家族序列中出现的次数并不相同。在80%以上的成员中均有检索到Motif 7、Motif 1和Motif 2,其中Motif 7和Motif 1总是串联出现,在序列上紧密排列;Motif 3、Motif 4、Motif 5、Motif 6、Motif 8和Motif 10出现的频率在60%以上;个别motif出现的次数较少,例如Motif 9,仅有6个β−淀粉酶序列含有该保守基序。
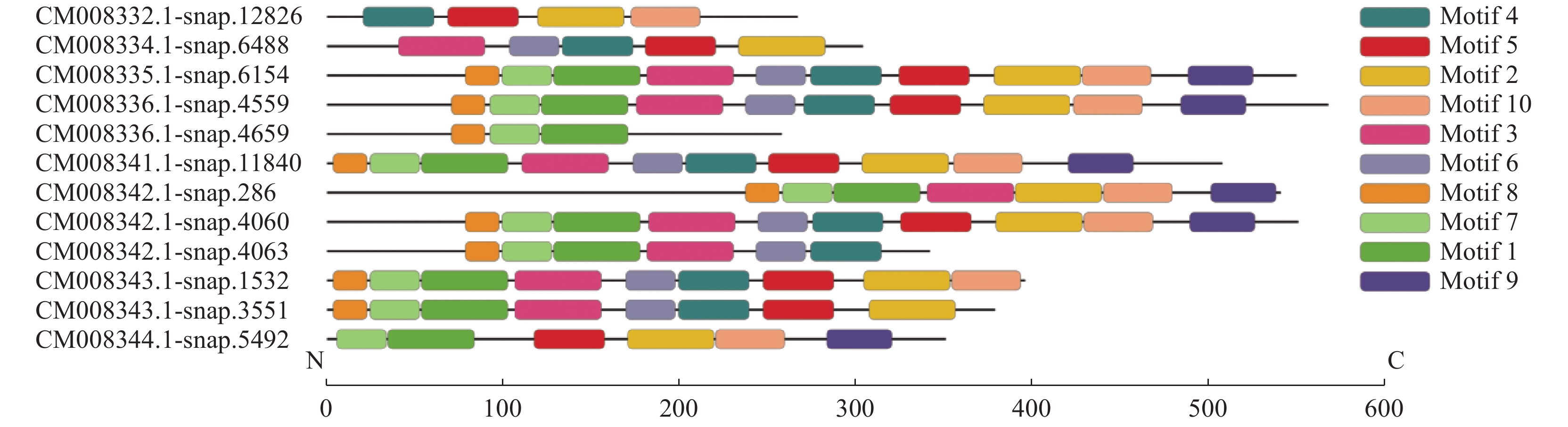
|
图 3 甘薯β−淀粉酶蛋白序列保守基序分析 Fig. 3 Conservative motif analysis of protein sequences of Ipomoea batatas β-amylases |
表2中为Motif 1~Motif 10的氨基酸序列信息通过SMART检索得到的功能注释结果。Motif 1、Motif 2、Motif 3、Motif 4、Motif 5和Motif 10检索结果均为Glyco_hydro_14结构域;Motif 9根据蛋白质的结构分类(Structural classification of proteins, SCOP)被归为d1byb_结构域,该结构域属于β−淀粉酶蛋白[26]。分析氨基酸组成特征,发现在一些保守基序中可以找到与图1对应的3个保守区域(画横线部分的氨基酸),如Motif 1和Motif 2中分别含有β−淀粉酶3个保守区域的Region I和Region II;Motif 6和Motif 4则包含了Region III。
|
|
表 2 甘薯β−淀粉酶保守基序氨基酸组成特征1) Table 2 Amino acid composition of the conservative motifs of the Ipomoea batatas β-amylase |
为了研究不同物种中β−淀粉酶基因的系统进化关系,下载了7个不同物种的β−淀粉酶蛋白序列,与甘薯β−淀粉酶蛋白序列合并后进行多重序列比对,利用NJ法构建系统发育树。这7个不同物种共50个蛋白序列分别为水稻10个、玉米12个、大豆3个、拟南芥9个、马铃薯7个、番茄7个以及大麦2个。各个物种β−淀粉酶蛋白序列的数据库查询编号以及所对应的简化后的蛋白名称如表3所示。
|
|
表 3 β−淀粉酶蛋白数据库查询编号 Table 3 The database query numbers of β-amylase proteins |
甘薯、拟南芥、马铃薯、番茄、大豆、水稻、玉米以及大麦8个物种共62个β−淀粉酶家族成员被分为S1~S7 7个亚组,如图4所示,树状图分枝上仅显示大于70%的Bootstrap值。图4中显示的蛋白名称与表3相对应。各个亚组中所包含的β−淀粉酶家族成员数并不相同,S7亚组包含的成员数最多,有15个;S4和S3亚组次之,分别有11个和8个;S1、S2和S6亚组成员数均为7个;S5亚组最少,仅有6个。甘薯12个β−淀粉酶家族成员主要分布于S2(2个)、S4(4个)、S5(1个)、S6(1个)以及S7(4个)亚组,S1和S3亚组中不含有甘薯β−淀粉酶家族成员。从图4可以发现,甘薯的β−淀粉酶与番茄、马铃薯和拟南芥的β−淀粉酶亲缘关系较为接近,在进化树上大多处于同一分支,例如甘薯CM008342.1-snap.286与马铃薯StBAM7、番茄SlBAM2以及拟南芥AtBAM7处于S6亚组的同一分支;S2亚组中的CM008336.1-snap.4559和CM008336.1-snap.4659与马铃薯StBAM4和番茄SlBAM7为同一进化树分支,且在图2的染色体定位结果中CM008336.1-snap.4559和CM008336.1-snap.4659所对应的基因在染色体上的位置相近。
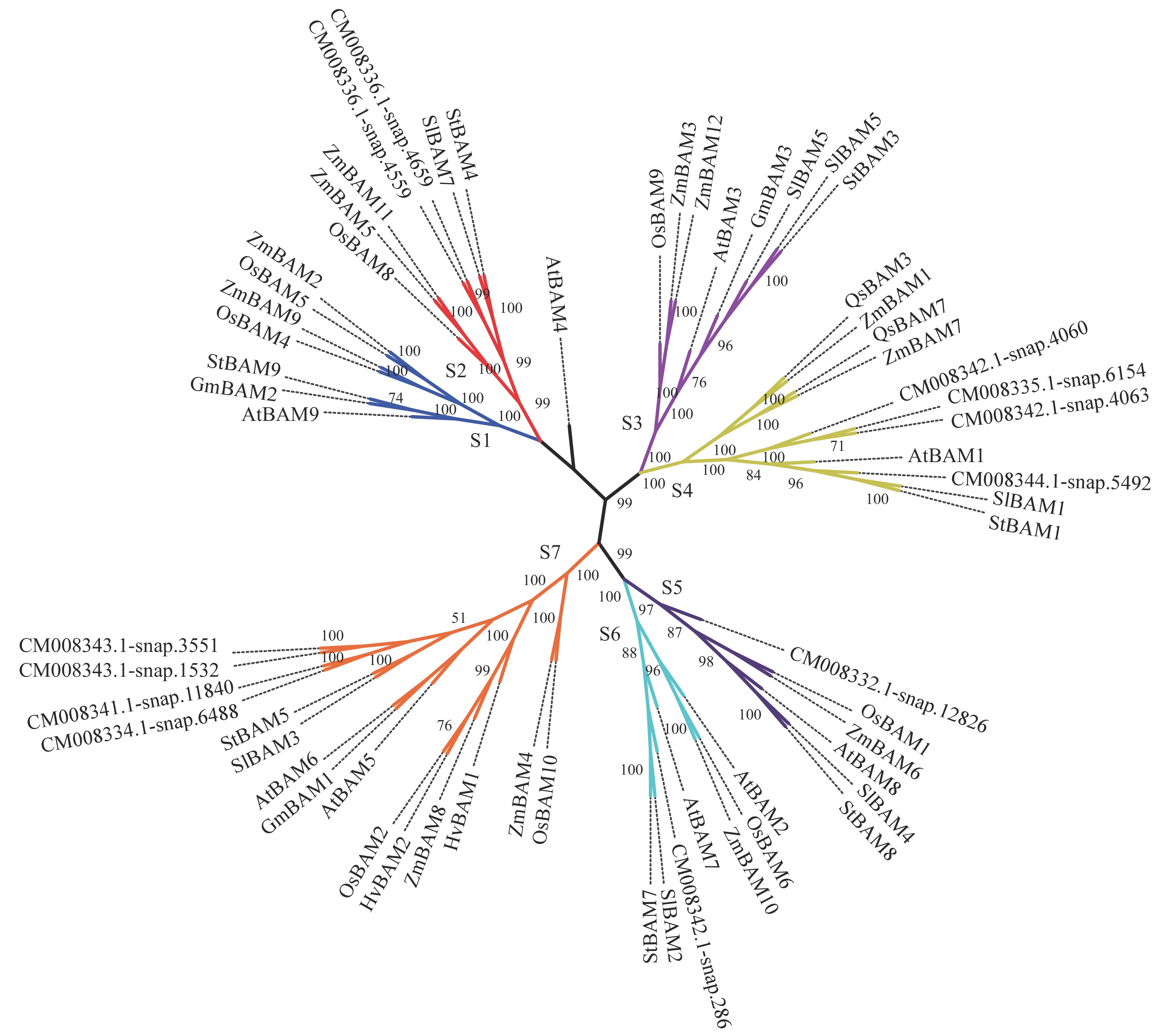
|
图 4 甘薯与拟南芥、马铃薯、番茄、大豆、水稻、玉米以及大麦的β−淀粉酶系统进化分析 Fig. 4 Phylogenetic analysis of β-amylases in eight species, i.e. Ipomoea batatas, Arabidopsis thaliana, Solanum tuberosum, Solanum lycopersicum, Glycine max, Oryza sativa, Zea mays and Hordeum vulgare S1~S7为7个亚组 S1−S7 are seven subgroups |
利用转录组测序数据[19]分析得到了甘薯在低温贮藏期间β−淀粉酶基因的表达量变化模式图(图5)。图5共有4个分组,分别为具有不同耐低温能力的甘薯品种‘徐薯15-1(高)’和‘徐薯15-4(低)’在4 ℃条件下贮藏2和6周后,β−淀粉酶基因表达量相比于对照组(贮藏0周)的变化情况。甘薯12个β−淀粉酶基因中共有6个基因在低温贮藏过程中表达量值出现上调(红色)或下调(绿色)变化。其中,‘徐薯15-1’有4个基因下调表达,2个基因上调表达;而在‘徐薯15-4’中则仅有2个下调表达的基因。在‘徐薯15-1’中,储藏2周时仅有2个基因表达量发生变化,储藏6周后则有6个基因表达量出现变化,说明随着贮藏时间的延长,‘徐薯15-1’中响应低温胁迫的基因数有所增加。
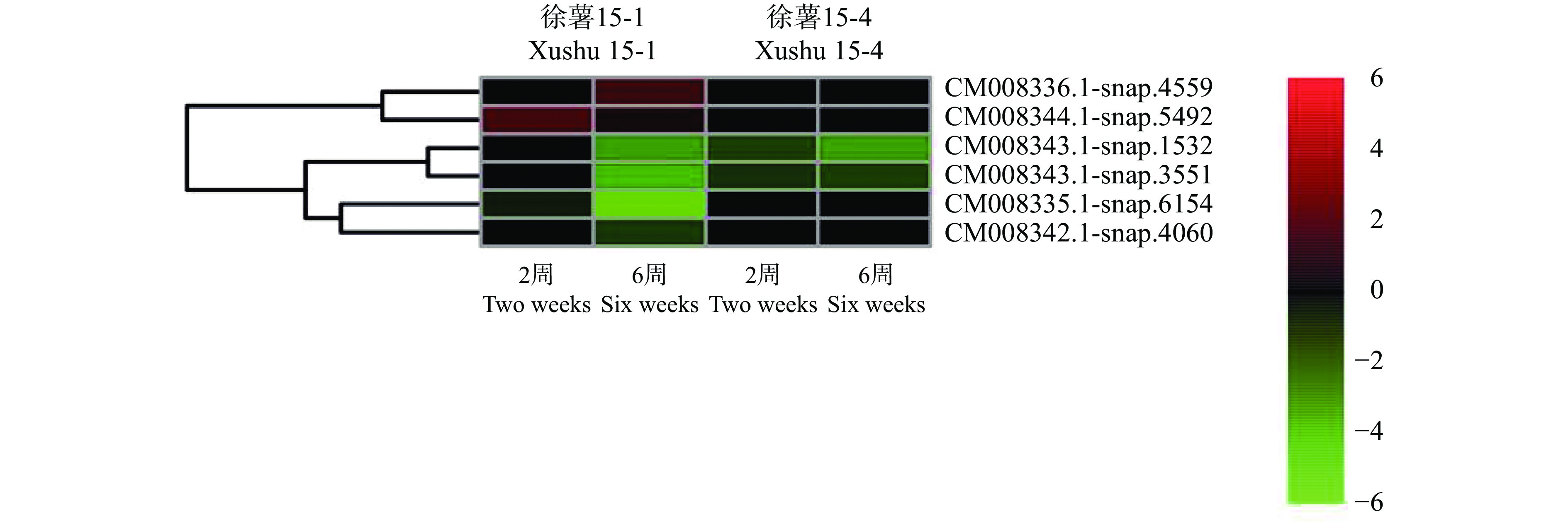
|
图 5 甘薯β−淀粉酶基因低温胁迫下不同贮藏时间的表达分析 Fig. 5 Expression analysis of β-amylase gene in Ipomoea batatas at different storage time under low temperature stress |
CM008343.1-snap.1532和CM008343.1-snap.3551在‘徐薯15-1’和‘徐薯15-4’中均表现为下调,在‘徐薯15-1’中贮藏2周时并未出现表达量的变化,6周时才出现明显的下调趋势;在‘徐薯15-4’中贮藏6周时的表达量下调变化比2周时更明显,复制基因分析发现这2个基因之间具有潜在重复关系(图2)。大部分甘薯β−淀粉酶基因在低温贮藏过程中下调表达,但CM008336.1-snap.4559和CM008344.1-snap.5492上调表达,推测这2个基因可能在甘薯块根受到低温胁迫时发挥作用。
3 讨论与结论本研究利用HMM法从甘薯全基因组中鉴定得到12个甘薯β−淀粉酶家族成员,这些成员均含有糖苷水解酶14家族的保守结构域Glyco_hydro_14,数量与拟南芥9个[27]、水稻10个以及玉米13个等[28]差异不大。植物中β−淀粉酶家族属于多基因家族,序列中功能结构域的保守性和保守基序的组成是多基因家族的重要特征。本研究表明,甘薯β−淀粉酶氨基酸序列中含有3个保守性较高的区域(region I~III),其中region II中的谷氨酸催化位点是影响甘薯β−淀粉酶催化活性的有效部位[2]。甘薯β−淀粉酶家族序列中保守基序的组成并不完全相同,若干存在于大多数β−淀粉酶成员中的保守基序体现了甘薯β−淀粉酶家族的共性,例如motif 1、motif 2和motif 7;多基因家族在进化过程中往往会出现功能的分化,一些仅存在于少部分β−淀粉酶成员中的保守基序造成了甘薯β−淀粉酶家族成员序列之间的差异,这些差异可能与甘薯β−淀粉酶家族在进化过程中功能的分化相关。
将甘薯与其他物种的β−淀粉酶序列共同构建系统进化树,以分析甘薯β−淀粉酶的进化关系和可能的功能特征。根据树状图的分支特点和遗传距离的远近将得到的进化树结果分为7个亚组(S1~S7),Thalmann等[24]对陆地植物中的β−淀粉酶家族进行分析,系统进化分析结果中将8种不同植物的β−淀粉酶分为8个亚组(I~VIII),其中拟南芥的9个β−淀粉酶家族成员被归入其中的7个亚组,分别为BAM1(I组)、BAM3(III组)、BAM4(IV组)、BAM9(V组)、BAM5和BAM6(VI组)、BAM2和BAM7(VII组)以及BAM8(VIII组)。本研究中,拟南芥的9个成员同样分散于不同的亚组,且各成员的分类与Thalmann等[24]的分类结果相一致,例如同样的AtBAM2和AtBAM7为一组(S6),AtBAM5和AtBAM6为一组(S7),其他成员则单独归入不同的亚组中。本研究中甘薯β−淀粉酶成员所处的分支基本由拟南芥、马铃薯和番茄的β−淀粉酶成员组成,尤其与亲缘关系相近的马铃薯和番茄β−淀粉酶家族成员在进化上拥有更紧密的联系。通过分析与甘薯β−淀粉酶家族成员遗传距离相近的马铃薯或番茄β−淀粉酶家族成员的功能,对甘薯β−淀粉酶基因的功能进行预测,Hou等[29]研究表明沉默马铃薯StBAM1基因会降低马铃薯中的β−淀粉酶活性,导致淀粉积累。甘薯β−淀粉酶基因CM008344.1-snap.5492与StBAM1基因处于同一分支,蛋白序列比对结果显示两者的相似性为86%,推测CM008344.1-snap.5492与StBAM1基因之间可能存在功能相似性,转录组测序结果显示CM008344.1-snap.5492基因在低温贮藏过程中上调表达;因此CM008344.1-snap.5492基因很可能对甘薯低温贮藏过程中淀粉的降解发挥作用。
本研究基于甘薯‘泰中6号’全基因组测序数据鉴定得到甘薯β−淀粉酶家族成员,由于基因组测序质量的限制[23],在个别序列组成上存在部分区域氨基酸的缺失,因而本研究结合SRA中的转录组测序数据,对鉴定得到的甘薯β−淀粉酶序列进行修补。研究表明,β−淀粉酶是甘薯块根中的主要酶[30]。甘薯收获后通常是在低温贮藏,在贮藏的过程中甘薯薯块中的β−淀粉酶可将淀粉降解为还原糖,提高甘薯的甜度,同时还可促进薯块发芽[31];因此,研究β−淀粉酶在甘薯低温贮藏过程中的活性变化对指导淀粉专用型甘薯品种的育种以及控制鲜食甘薯品种的采后品质具有重要意义。Ji等[19]通过高通量测序技术获得了‘徐薯15-1’和‘徐薯15-4’的块根在低温贮藏条件下的转录组数据,并利用qRT-PCR技术进行了可靠性验证;本研究基于该转录组数据分析了甘薯β−淀粉酶基因在低温贮藏过程中基因表达量的变化情况,获得了6个差异表达基因,这些结果可为甘薯β−淀粉酶基因功能的研究提供重要参考。
| [1] |
张勇为, 张义正, 谭文芳, 等. 甘薯贮藏期间淀粉酶种类变化及其部分性质分析[J]. 四川大学学报(自然科学版), 2018, 55(1): 197-200. DOI:10.3969/j.issn.0490-6756.2018.01.032 (  0) 0) |
| [2] |
TODA H, NITTA Y, ASANAMI S, et al. Sweet potato β-amylase: Primary structure and identification of the active-site glutamyl residue
[J]. European Journal of Biochemistry, 1993, 216(1): 25-38. DOI:10.1111/j.1432-1033.1993.tb18112.x (  0) 0) |
| [3] |
孙俊良, 梁新红, 贾彦杰, 等. 植物β−淀粉酶研究进展
[J]. 河南科技学院学报(自然科学版), 2011, 39(6): 1-4. DOI:10.3969/j.issn.1008-7516.2011.06.001 (  0) 0) |
| [4] |
LI H S, ÔBA K. Major soluble proteins of sweet potato roots and changes in proteins after cutting, infection, or storage[J]. Agricultural and Biological Chemistry, 2014, 49(3): 737-744. (  0) 0) |
| [5] |
NAKAMURA K, OHTO M A, YOSHIDA N, et al. Sucrose-induced accumulation of beta-amylase occurs concomitant with the accumulation of starch and sporamin in leaf-petiole cuttings of sweet potato[J]. Plant Physiology, 1991, 96(3): 902-909. DOI:10.1104/pp.96.3.902 (  0) 0) |
| [6] |
梁新红, 李英, 孙俊良, 等. β−淀粉酶酶解甘薯淀粉条件分析
[J]. 食品工业科技, 2014, 35(7): 178-181. (  0) 0) |
| [7] |
陈显让, 李红兵, 康乐, 等. 甘薯块根膨大后期β−淀粉酶和淀粉含量相关性分析
[J]. 食品工业科技, 2013, 34(19): 93-96. (  0) 0) |
| [8] |
CHEONG C G, EOM S H, CHANG C, et al. Crystallization, molecular replacement solution, and refinement of tetrameric beta-amylase from sweet potato[J]. Proteins, 1995, 21(2): 105-117. DOI:10.1002/prot.340210204 (  0) 0) |
| [9] |
YANG J, MOEINZADEH M H, KUHL H, et al. Haplotype-resolved sweet potato genome traces back its hexaploidization history[J]. Nature Plants, 2017, 3(9): 696-703. DOI:10.1038/s41477-017-0002-z (  0) 0) |
| [10] |
黄小芳, 毕楚韵, 石媛媛, 等. 甘薯基因组NBS-LRR类抗病家族基因挖掘与分析[J]. 作物学报, 2020, 46(8): 1195-1207. (  0) 0) |
| [11] |
ALTSCHUL S F, GISH W, MILLER W, et al. Basic local alignment search tool[J]. Journal of Molecular Biology, 1990, 215(3): 403-410. DOI:10.1016/S0022-2836(05)80360-2 (  0) 0) |
| [12] |
LU S, WANG J, CHITSAZ F, et al. CDD/SPARCLE: The conserved domain database in 2020[J]. Nucleic Acids Research, 2020, 48(D1): D265-D268. DOI:10.1093/nar/gkz991 (  0) 0) |
| [13] |
QUEVILLON E, SILVENTOINEN V, PILLAI S, et al. InterProScan: Protein domains identifier[J]. Nucleic Acids Research, 2005, 33: W116-W120. DOI:10.1093/nar/gki442 (  0) 0) |
| [14] |
SIEVERS F, HIGGINS D G. Clustal Omega for making accurate alignments of many protein sequences[J]. Protein Science, 2018, 27(1): 135-145. DOI:10.1002/pro.3290 (  0) 0) |
| [15] |
KRZYWINSKI M, SCHEIN J, BIROL I, et al. Circos: An information aesthetic for comparative genomics[J]. Genome Research, 2009, 19(9): 1639-1645. DOI:10.1101/gr.092759.109 (  0) 0) |
| [16] |
GU Z, CAVALCANTI A, CHEN F C, et al. Extent of gene duplication in the genomes of drosophila, nematode, and yeast[J]. Molecular Biology and Evolution, 2002, 19(3): 256-262. DOI:10.1093/oxfordjournals.molbev.a004079 (  0) 0) |
| [17] |
BAILEY T L, BODEN M, BUSKE F A, et al. MEME SUITE: Tools for motif discovery and searching[J]. Nucleic Acids Research, 2009, 37: W202-W208. DOI:10.1093/nar/gkp335 (  0) 0) |
| [18] |
KUMAR S, STECHER G, LI M, et al. MEGA X: Molecular evolutionary genetics analysis across computing platforms[J]. Molecular Biology and Evolution, 2018, 35(6): 1547-1549. DOI:10.1093/molbev/msy096 (  0) 0) |
| [19] |
JI C Y, KIM H S, LEE C J, et al. Comparative transcriptome profiling of tuberous roots of two sweetpotato lines with contrasting low temperature tolerance during storage[J]. Gene, 2020, 727. DOI:10.1016/j.gene.2019.144244 (  0) 0) |
| [20] |
KIM D, LANGMEAD B, SALZBERG S L. HISAT: A fast spliced aligner with low memory requirements[J]. Nature Methods, 2015, 12(4): 357-360. DOI:10.1038/nmeth.3317 (  0) 0) |
| [21] |
LOVE M I, HUBER W, ANDERS S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2[J]. Genome Biology, 2014, 15(12). DOI:10.1186/s13059-014-0550-8 (  0) 0) |
| [22] |
TOTSUKA A, NONG V H, KADOKAWA H, et al. Residues essential for catalytic activity of soybean beta-amylase[J]. European Journal of Biochemistry, 1994, 221(2): 649-654. DOI:10.1111/j.1432-1033.1994.tb18777.x (  0) 0) |
| [23] |
WU S, LAU K H, CAO Q, et al. Genome sequences of two diploid wild relatives of cultivated sweetpotato reveal targets for genetic improvement[J]. Nature Communications, 2018, 9. DOI:10.1038/s41467-018-06983-8 (  0) 0) |
| [24] |
THALMANN M, COIRO M, MEIER T, et al. The evolution of functional complexity within the β-amylase gene family in land plants
[J]. BMC Evolutionary Biology, 2019, 19. DOI:10.1186/s12862-019-1395-2 (  0) 0) |
| [25] |
KANG Y N, ADACHI M, UTSUMI S, et al. The roles of Glu186 and Glu380 in the catalytic reaction of soybean beta-amylase[J]. Journal of Molecular Biology, 2004, 339(5): 1129-1140. DOI:10.1016/j.jmb.2004.04.029 (  0) 0) |
| [26] |
CHEN Y, CRIPPEN G M. An iterative refinement algorithm for consistency based multiple structural alignment methods[J]. Bioinformatics, 2006, 22(17): 2087-2093. DOI:10.1093/bioinformatics/btl351 (  0) 0) |
| [27] |
VALERIO C, COSTA A, MARRI L, et al. Thioredoxin-regulated beta-amylase (BAM1) triggers diurnal starch degradation in guard cells, and in mesophyll cells under osmotic stress[J]. Journal of Experimental Botany, 2011, 62(2): 545-555. DOI:10.1093/jxb/erq288 (  0) 0) |
| [28] |
杨泽峰, 徐暑晖, 王一凡, 等. 禾本科植物β−淀粉酶基因家族分子进化及响应非生物胁迫的表达模式分析
[J]. 科技导报, 2014, 32(31): 29-36. DOI:10.3981/j.issn.1000-7857.2014.31.002 (  0) 0) |
| [29] |
HOU J, ZHANG H, LIU J, et al. Amylases StAmy23, StBAM1 and StBAM9 regulate cold-induced sweetening of potato tubers in distinct ways[J]. Journal of Experimental Botany, 2017, 68(9): 2317-2331. DOI:10.1093/jxb/erx076 (  0) 0) |
| [30] |
HATTORI T, FUKUMOTO H, NAKAGAWA S, et al. Sucrose-induced expression of genes coding for the tuberous root storage protein, sporamin, of sweet potato in leaves and petioles[J]. Plant and Cell Physiology, 1991, 32(1): 79-86. (  0) 0) |
| [31] |
唐君, 周志林, 林冬兰, 等. 甘薯贮藏过程淀粉酶活性变化及对薯块芽萌发的影响[J]. 福建农业学报, 2010, 25(6): 699-702. DOI:10.3969/j.issn.1008-0384.2010.06.008 (  0) 0) |
 2021, Vol. 42
2021, Vol. 42


