2. 广东省林业科学研究院/广东省森林培育与保护利用重点实验室,广东 广州 510520
2. Guangdong Academy of Forestry/Guangdong Provincial Key Laboratory of Silviculture, Protection and Utilization, Guangzhou 510520, China
代谢组学利用现代分析技术对生物体内小分子化合物进行定性定量研究[1],结合现代生物信息学可实现对代谢物全面地、无偏倚地分析[2]。研究微生物代谢物的变化动态,有助于我们更加了解微生物内部的生理变化。目前代谢组分析技术主要有气相色谱−质谱联用(GC-MS)[3]、液相色谱−质谱联用(LC-MS)[4]和核磁共振(NMR)[5]等。基于GC-MS技术的样品前处理步骤主要包括:淬灭、提取、衍生化和检测。在代谢物淬灭过程中,应尽可能地保证微生物细胞的完整性,避免胞内代谢物外流[6],可以通过迅速降低温度或者在菌体培养液中加入有机溶剂等手段抑制代谢酶活性。冷甲醇淬灭是普遍使用的方法[7],它可以在亚秒时间内阻断代谢反应,将胞内外的代谢物区分开[8-9]。常用的提取方法有冷甲醇液氮反复冻融法[10]和75%乙醇煮沸法[11]等。常用的提取液有热乙醇、高氯酸或碱、甲醇和三氯甲烷等。在破碎细胞时,提取液将胞内的代谢物溶解,为了确保提取到稳定可靠的代谢物,可以加入4−羟乙基哌嗪乙磺酸(HEPES)和哌嗪乙磺酸(PIPES)等盐离子进行缓冲[12]。此外超临界流体萃取、微波辅助萃取[13]、固相萃取、固相微萃取等方法也可用于微生物代谢物提取。但至今,仍然没有一种适用于所有微生物的提取方法[14],不同微生物、不同提取方法提取的代谢物倾向性和分析技术亦不同。因此在每一项新的代谢组学研究中,提取方法一定要有针对性的优化和测试。
近年来,以绿僵菌Metarhizium spp.、白僵菌Beauveria spp.为代表的真菌生物农药已成为生物防治领域的热门研究[15]。在菌种保存和应用过程中,继代培养造成菌株形态变异和毒力减退等退化现象普遍发生,极大地影响了真菌杀虫剂在生产上的应用。继代培养后分生孢子产量与毒力的下降常常伴随着次生代谢产物、代谢路径和代谢网络等层面的变化[16-19]。目前微生物代谢组学研究主要集中于大肠埃希菌Escherichia coli[20]、酿酒酵母Saccharomyces cerevisiae[21]及枯草芽孢杆菌Bacillus subtilis[22-23]等几种模式微生物,并且没有一种方法可以适用于所有微生物的代谢研究。为了获得真实、可靠和全面的代谢组学信息,需要一个准确、稳定的代谢组学检测方法,但目前关于绿僵菌代谢组仅有少量基于LC-MS检测的研究[24],鲜见基于GC-MS的相关研究。本文以绿僵菌Ma09为研究对象,采用高分辨率的GC-MS技术,对绿僵菌代谢物前处理技术进行优化,以期建立适用于绿僵菌的代谢组检测方法,为获得和解析绿僵菌的“代谢物指纹图谱”提供技术支持,为绿僵菌的深入研究及应用提供参考。
1 材料与方法 1.1 供试菌株绿僵菌Ma09,由广东省林业科学研究院提供,绿僵菌斜面保存于4 ℃冰箱备用。转接于PPDA液体培养基,200 r/min、25 ℃条件下培养72 h。
1.2 方法 1.2.1 淬灭参照Tsuchido等[25]的方法采用D260 nm和D280 nm回收率法评价淬灭剂对细胞膜损伤的程度。核酸和蛋白的特异吸光波长分别为260和280 nm,淬灭后菌液的D260 nm和D280 nm增加,意味着细胞内的核酸和蛋白已渗出到菌液中,说明细胞膜已产生损伤。取72 h菌体培养液10 mL,4 ℃、6000 r/min离心10 min,离心所得沉淀再用9 g/L NaCl溶液润洗1次去除残留培养基。分别用10 mL不同淬灭剂(0 ℃)将菌体重悬,以9 g/L NaCl溶液作为阴性对照、热9 g/L NaCl (70 ℃, 30 min)溶液作为阳性对照,4 ℃、6000 r/min离心10 min,得到上清液,试验重复3次。测定上清液的D260 nm和D280 nm,根据如下公式计算核酸的回收率(η260 nm)和蛋白的回收率(η280 nm)。
| $ \begin{array}{l} \!\! {\eta }_{260{\rm{\;nm}}}\!\!=\!\!\dfrac{{D}_{{260}{\rm{\;nm}}}{(}{{\text{处理}}}{)}-{D}_{{260}{\rm{\;nm}}}{({\text{阴性对照}})}}{{D}_{{260}{\rm{\;nm}}}{({\text{阳性对照}})}-{D}_{{260}{\rm{\;nm}}}{({\text{阴性对照}})}}{ \!\times\! 100{\text{%}}}{,} \\ \!\! {\eta }_{{280}{\rm{\;nm}}}\!=\!\dfrac{{D}_{{280}{\rm{\;nm}}}{(}{{\text{处理}}}{)}\!-\!{D}_{{280}{\rm{\;nm}}}{({\text{阴性对照}})}}{{D}_{{280}{\rm{\;nm}}}{({\text{阳性对照}})}\!-\!{D}_{{280}{\rm{\;nm}}}{({\text{阴性对照}})}}{ \!\times\! 100{\text{%}}}{\text{。}} \end{array}$ |
淬灭剂参考前人的研究[7, 26],选择甲醇、乙醇和甘油溶液,体积分数分别为20%、40%和60%。添加的缓冲盐为9 g/L的NaCl、NaCN和(NH4)2CO3,10 mmol/L EDTA及10 mmol/L HEPES溶液。
1.2.2 绿僵菌代谢物气相色谱质谱检测将绿僵菌Ma09菌液以40%(φ)乙醇溶液淬灭,−40 ℃冷甲醇提取后N2吹干,加入20 mg/mL的甲氧基胺盐酸盐吡啶溶液在37 ℃反应60 min,加入含1%(φ)三甲基氯硅烷(TMCS)的N, O−双(三甲基硅烷基)三氟乙酰胺(φ为96%的BSTFA )衍生1 h。采用Agilent 8890+5977B GC/MS系统,毛细管色谱柱为HP-5ms(30 m×0.25 mm×0.25μm) (φ为5%的苯基甲基硅氧烷)非极性弹性石英毛细管柱。质谱条件:电子轰击源(EI),离子源温度为230 ℃,四极杆温度为150 ℃,接口温度为280 ℃,电子能量为70 eV,调谐方式为标准调谐,质谱扫描方式为全扫描,扫描范围为30~500 aum。使用NIST谱库进行图谱检索,试验重复3次。
气相色谱以纯度大于99.99%(φ)的高纯氦气(Ne)为载气,载气流速1 mL/min。采用不分流进样方式,进样量l μL。进样口温度280 ℃。气相色谱条件如下:
条件A:起始温度50 ℃,保持3 min;以10 ℃/min升至150 ℃,保持5 min;然后以5 ℃/min升至200 ℃,保持5 min;以10 ℃/min升至280 ℃,保持5 min。
条件B:参考明明[27]的GC条件。起始温度70 ℃,保持4 min;以3 ℃/min升至200 ℃;以10 ℃/min升至280 ℃,保持5 min。
条件C:参考王洪彬等[23]的GC条件。初始温度70 ℃,恒温保持2 min;以3 ℃/min升到133 ℃;以2 ℃/min升至200 ℃;再以3 ℃/min升至220 ℃;以5 ℃/min升温至280 ℃。
条件D:参考胡志宏等[28]的GC条件,起始温度65 ℃,保持2 min;以5 ℃/min升至185 ℃;以1 ℃/min升至200 ℃;以15 ℃/min升至280 ℃,保持5 min。
条件E:参考郭刚等[29]的GC条件,起始温度70 ℃,保持4 min;以5 ℃/min升至280 ℃,保持4 min。
1.2.3 提取热乙醇提取法参考Castrillo等[11]的方法略作修改,淬灭后的绿僵菌沉淀加入5 mL75%(φ)热乙醇溶液(含0.25 mol/L HEPES溶液),pH为7.5,95 ℃水浴3 min;然后迅速在冰浴中冷却3 min,12000 r/min离心10 min,取上清液在90 ℃条件下旋转蒸发至1 mL。N2吹干后待衍生处理。
热甲醇提取法参照Maharjan等[12]的方法。淬灭后的绿僵菌沉淀中加入2 mL的甲醇溶液[V(甲醇)∶V(H2O)=2∶1]混合均匀后迅速放入70 ℃水中平衡30 min,然后以12000 r/min离心5 min,取上清液N2吹干待后衍生化处理。
冷甲醇提取法参考前人研究[30-32]并作相应修改,淬灭后的绿僵菌沉淀用2 mL预冷的甲醇(−20 ℃)溶解,迅速放入−20 ℃冰箱中保存20 min,冰浴冻融10 min后以12000 r/min离心5 min。上清液转移至另一离心管,取上清液1 mL将沉淀再次悬起,重复上述步骤,2次上清液合并,N2吹干后待衍生化处理。
甲醇氯仿提取法参考Maharjan等[12]的方法。淬灭后的绿僵菌沉淀用1 mL甲醇溶解后,加入1 mL冷三氯甲烷(−40 ℃)涡旋30 s后置于−20 ℃中保存60 min,此过程中每隔15 min涡旋1次,最后12000 r/min离心10 min,取上清液N2吹干待衍生化处理。
氯仿−甲醇−缓冲液提取法参考Maharjan等[12]的方法略作修改。淬灭后的绿僵菌沉淀用5 mL冷三氯甲烷(−40 ℃)溶解后,加入2 mL含有3 mmol/L HEPES和3 mmol/L EDTA的冷缓冲溶液(0 ℃),混合均匀后12000 r/min离心5 min。收集上层液并在剩余的三氯甲烷相中加入2 mL预冷的甲醇(−40 ℃)和2 mL冷缓冲液,涡旋30 s后10000 r/min离心5 min,收集上层水相并与前一次提取液合并,N2吹干待衍生化处理。
1.2.4 衍生化代谢物经淬灭和提取后必须进行衍生化处理后才能进行GC-MS检测分析。对衍生时间进行比较,取吹干后的样品,加入20 mg/mL的甲氧基胺盐酸盐吡啶溶液80 μL,剧烈振荡30 s后,在37 ℃条件下反应90 min,反应结束后冷却至室温,然后加入80 μL含1%(φ)TMCS的BSTFA衍生剂,在70 ℃条件下分别衍生0.5、1.0和1.5 h,冷却至室温后进行GC-MS检测,3次重复。
1.3 数据处理利用Mass Hunter软件对结果进行分析,提取离子峰并用NIST MS数据库进行定性分析,将所有离子峰的峰面积数据归一化,采用SPSS 20.0统计软件进行数据处理及分析,采用Duncan’s法进行差异显著性分析。
2 结果与分析 2.1 整体水平的细胞损伤及代谢物的渗出D260 nm和D280 nm是评价细胞渗出物的指标。图1的结果显示,D260 nm最小的淬灭剂为40%(φ)乙醇溶液和20%(φ)甘油溶液,两者差异不显著,但明显低于其他淬灭剂处理的(P<0.05)。D280 nm最小的淬灭剂为60%(φ)甲醇溶液和20%(φ)甘油溶液,且两者间差异不显著,但明显低于其他淬灭剂处理的(P<0.05)。
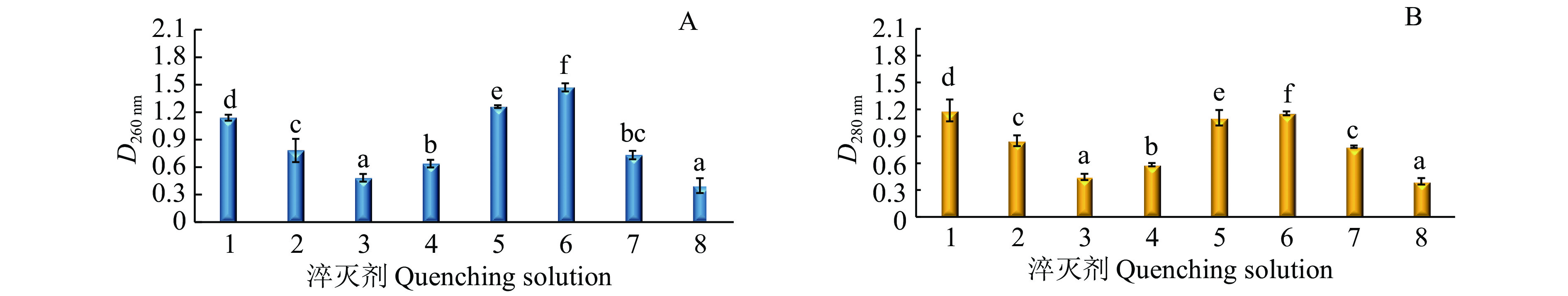
|
图 1 不同淬灭剂处理后菌液的D260 nm和D280 nm Fig. 1 D260 nm and D280 nm of bacterial fluid with different quenching solutions 1、2、3分别表示体积分数为60%, 40% 和20%的甘油溶液;4、5、6分别表示体积分数为60%, 40% 和20%的甲醇溶液;7、8分别表示体积分数为60%和40%的乙醇溶液;各图中的不同小写字母表示差异显著(P<0.05,Duncan’s法) 1, 2 and 3 represent glycerol solutions with volume fractions of 60%, 40% and 20%, respectively; 4, 5 and 6 represent methanol solutions with volume fractions of 60%, 40% and 20%, respectively; 7, 8 represent ethanol solutions with volume fractions of 60% and 40%, respectively; Different lowercase letters in each figure indicate significant differences (P<0.05,Duncan’s test) |
D260 nm和D280 nm越小说明淬灭过程中的代谢物渗出越少,淬灭效果越好。回收率计算结果(表1)显示,40%(φ)乙醇溶液和20%(φ)甘油溶液2种处理的核酸和蛋白回收率最低。
|
|
表 1 不同淬灭剂处理后核酸和蛋白的回收率 Table 1 The recovery rates of nucleic acid and protein using different quenching solutions |
以40%(φ)乙醇溶液为空白对照,分别加入不同种类的离子盐,从图2可以看出缓冲盐的加入降低了菌液的D260 nm和D280 nm,减少了菌体细胞的损伤,但是多重比较并未发现几种离子盐之间差异显著[F(D260 nm)=0.893,F(D280 nm)=2.009]。操作中发现,20%(φ)甘油溶液由于黏稠度比较大,不利用后期的浓缩,因此本试验选择40%(φ)乙醇溶液且不加入任何离子盐作为最佳淬灭剂。
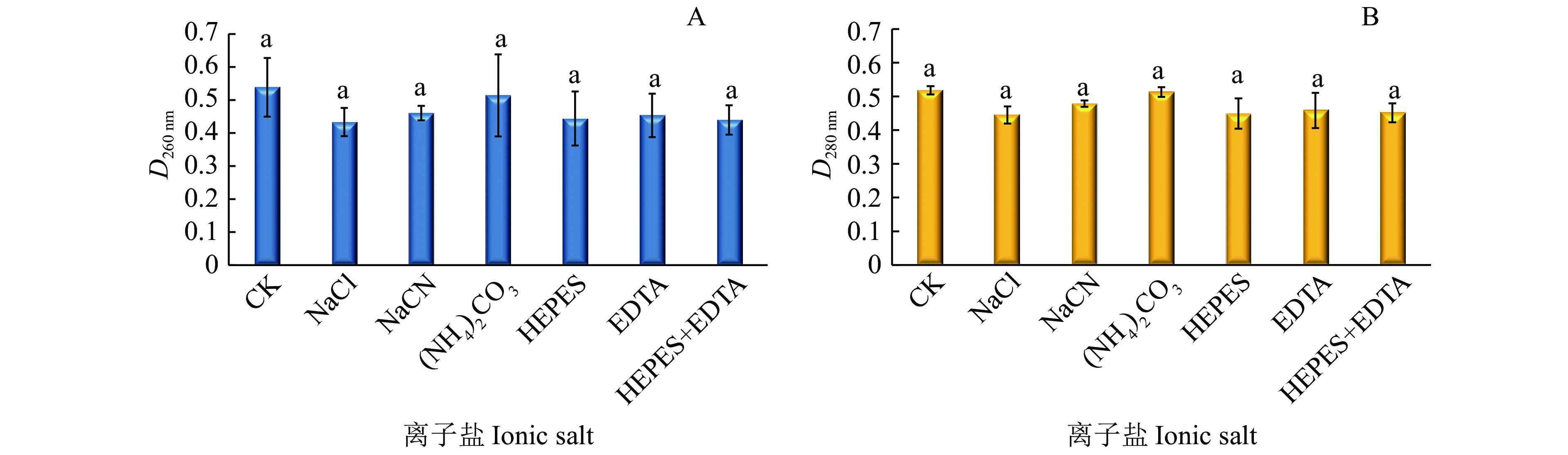
|
图 2 不同离子盐加入后菌液的D260 nm和D280 nm Fig. 2 D260 nm and D280 nm of bacterial fluid added with different ionic salt 各图中不同小写字母表示差异显著(P<0.05,Duncan’s法) Different lowercase letters in each figure indicate significant differences (P<0.05,Duncan’s test) |
图3为5种气相色谱条件的总离子流图。由于条件不同,各处理所用的时间也不同,条件C的总时最长为75 min,条件A仅需46 min。此外出峰时间和出峰数量存在明显差异。
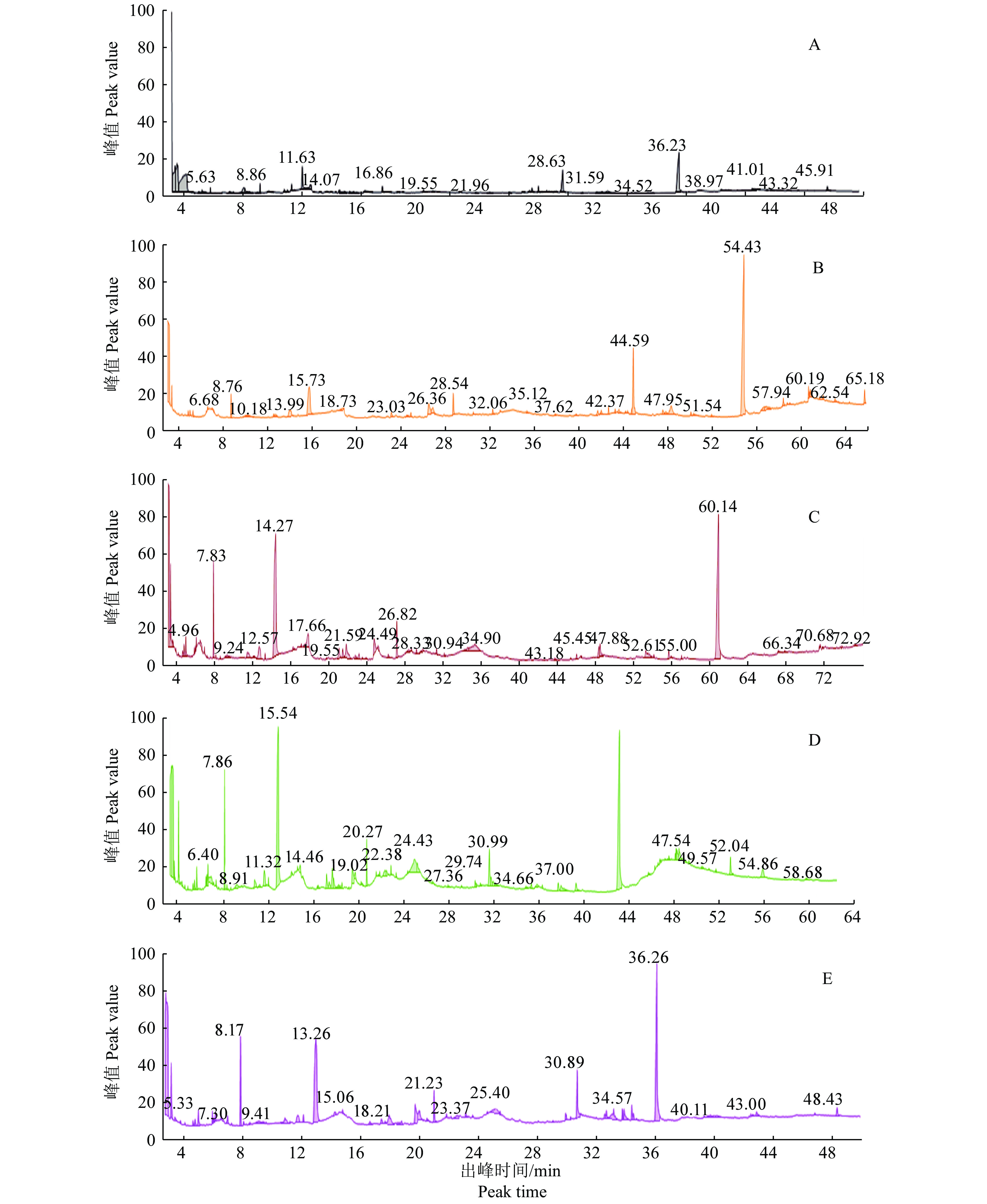
|
图 3 不同气相色谱条件检测出的总离子流图 Fig. 3 Graph of total ion current (TIC) under different chromatographic conditions A:起始温度50 ℃保持3 min,以10 ℃/min升至150 ℃保持5 min,5 ℃/min升至200 ℃保持5 min,10 ℃/min升至280 ℃保持5 min;B:起始温度70 ℃保持4 min,以3 ℃/min升至200 ℃,10 ℃/min升至280 ℃保持5 min;C:初始温度70 ℃保持2 min,以3 ℃/min至133 ℃,以2 ℃/min升至200 ℃,以3 ℃/min升至220 ℃,以5 ℃/min升至280 ℃;D:起始温度65 ℃保持2 min,以5 ℃/min升至185 ℃,以1 ℃/min升至200 ℃,以15 ℃/min升至280 ℃保持5 min;E:起始温度70 ℃保持4 min,以5 ℃/min升至280 ℃保持4 min A: The initial temperature was 50 ℃ for 3 min, then increased to 150 ℃ for 5 min at 10 ℃/min, then increased to 200 ℃ at 5 ℃/min and maintained for 5 min, and finally increased to 280 ℃ at 10 ℃/min and maintained for 5 min; B: The initial temperature was 70 ℃ for 4 min, then increased to 200 ℃ at 3 ℃ /min and then increased to 280 ℃ at 10 ℃ /min for 5 min; C: The initial temperature was 70 ℃ for 2 min, the temperature rose to 133 ℃ at 3 ℃/min, then rose to 200 ℃ at 2 ℃/min, then rose to 220 ℃ at 3 ℃/min, and finally rose to 280 ℃ at 5 ℃/min; D: The initial temperature was 65 ℃ for 2 min, then increased to 185 ℃ at 5 ℃/min, 200 ℃ at 1 ℃/min, and finally increased to 280 ℃ at 15 ℃/min for 5 min; E: The initial temperature was 70 ℃ for 4 min, and then increased to 280 ℃ at 5 ℃/min and maintained for 4 min |
根据检测出来的有效峰的数量可以判断出条件A的效果最好,条件A所获得的有效峰为97.33个,显著多于其他几种条件的(P<0.05)。
2.4 提取方法对绿僵菌代谢物提取效果的影响选用了GC-MS对5种不同方法的提取效果进行分析,根据有效峰数量评判最优方法,各处理获得有效峰数量差异显著(P<0.05)。各处理样本TIC谱图见图4。
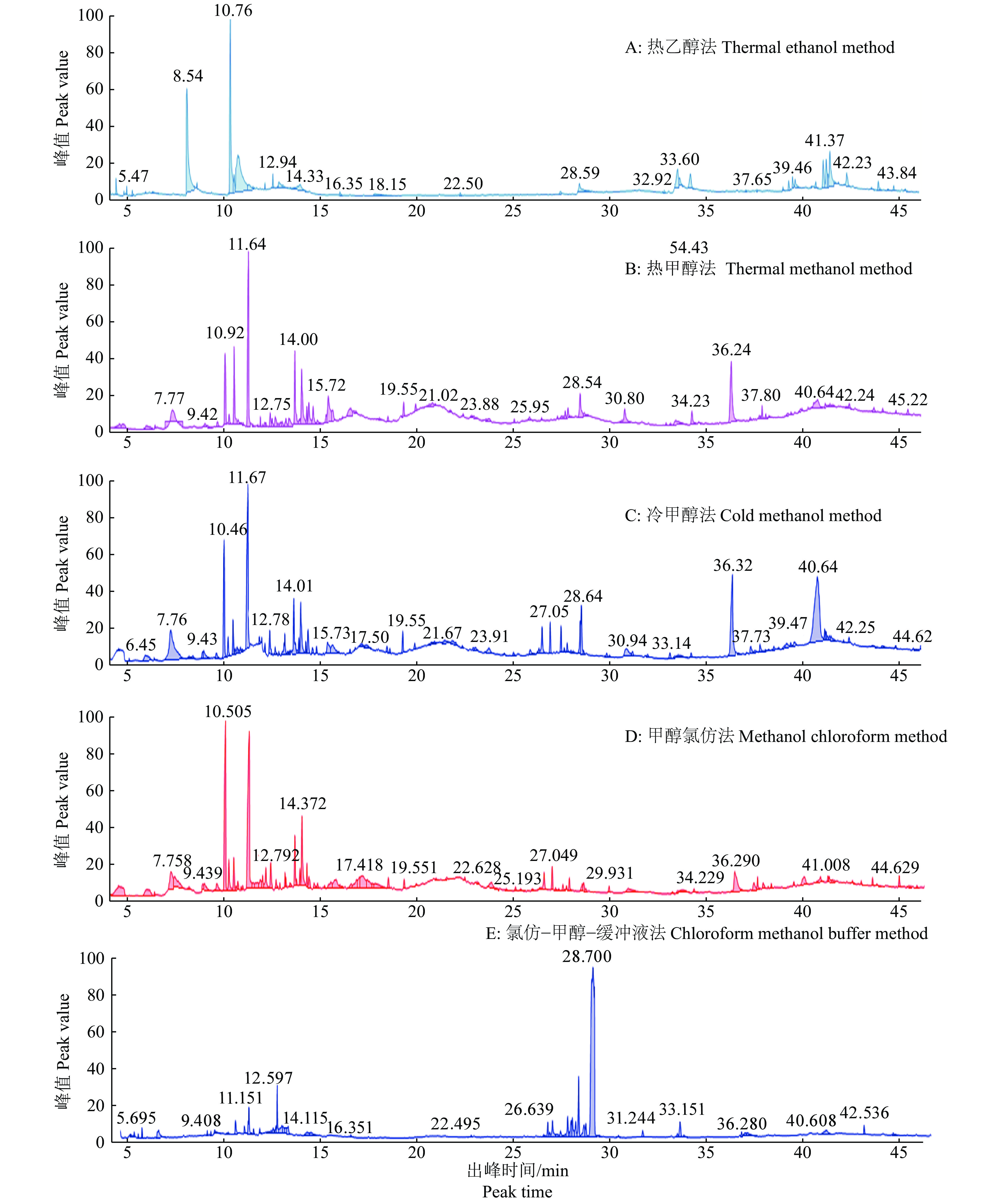
|
图 4 不同提取方法检测出的总离子流图 Fig. 4 Graph of total ion current (TIC) using different extraction methods |
根据有效峰的数量和强度判断,冷甲醇法效果最佳,可以检测出有效峰109.4个,其次是热乙醇法。
2.5 不同衍生物比例和时间的影响不同衍生时间的总离子流图见图5,以图5中物质数量和峰强度作为判断标准。根据图6有效峰数量可知,衍生0.5 h时大多数物质浓度较低且有多种物质无法检测到,而衍生1.0和1.5 h时检测到的物质数量显著提高了(P<0.05),衍生1.5 h后可以检测到90.33个有效峰。
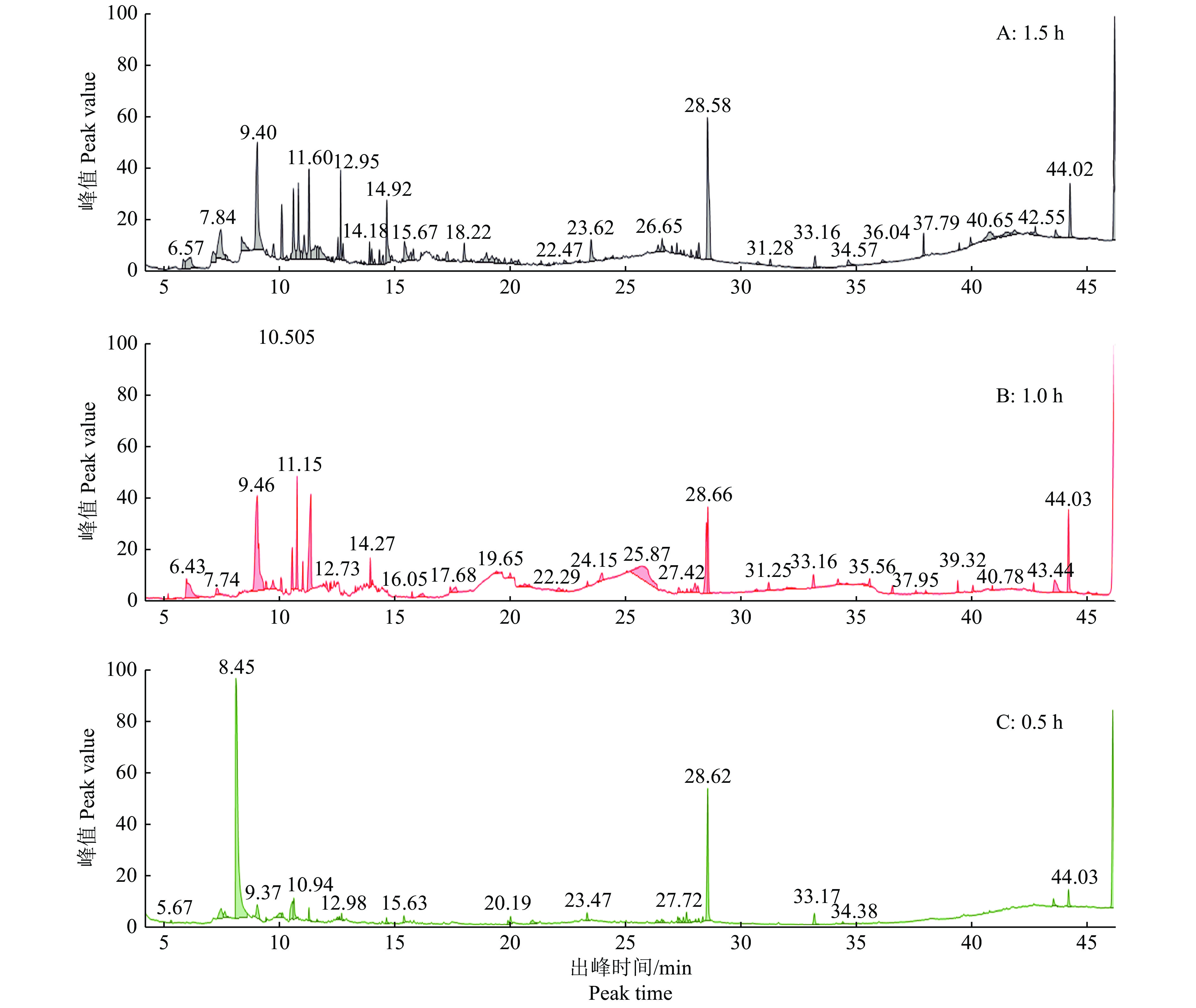
|
图 5 不同衍生时间的总离子流图(TIC) Fig. 5 Graph of total ion current (TIC) at different derivative time |
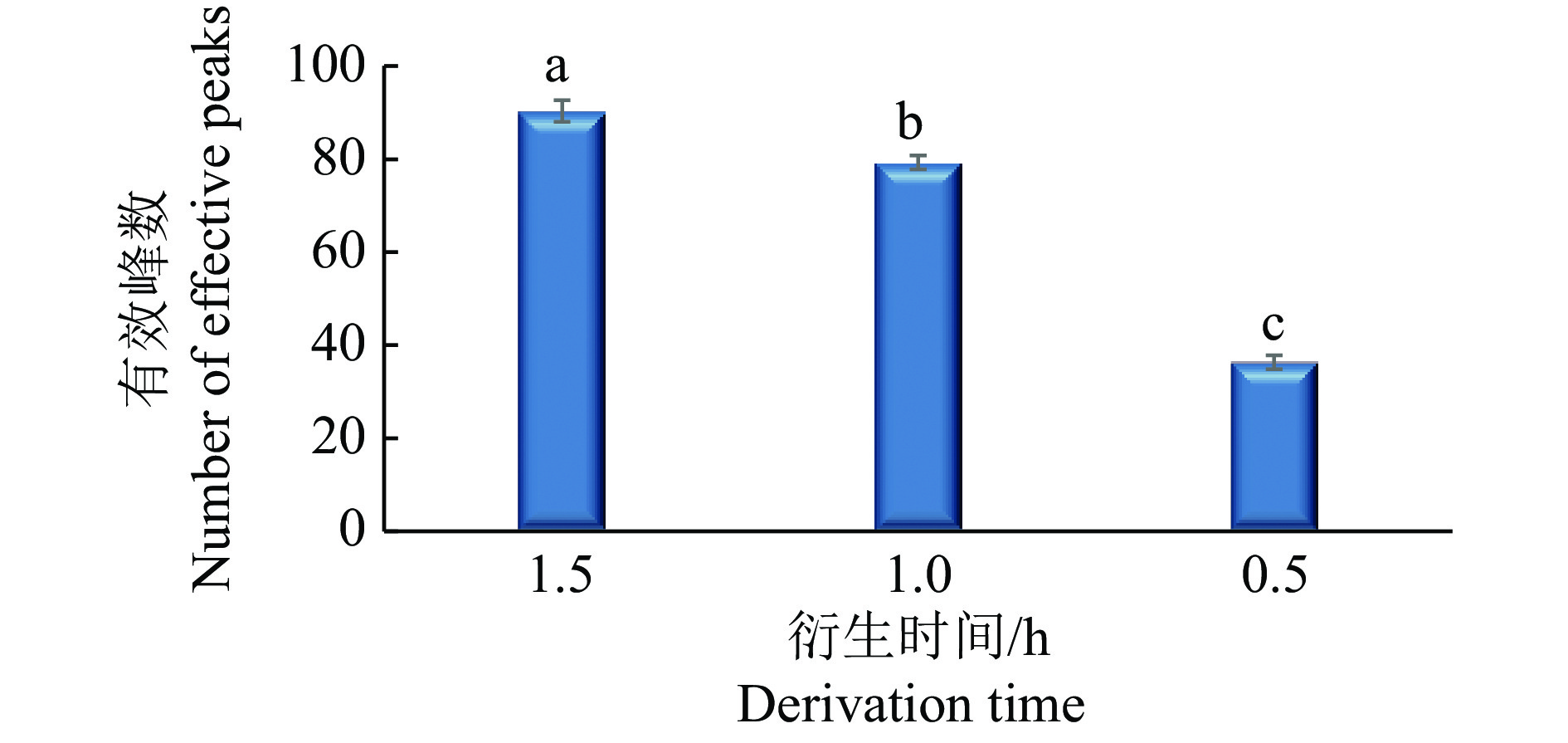
|
图 6 不同衍生时间的有效峰 Fig. 6 Effective peak at different derivative time 图中不同小写字母表示差异显著(P<0.05,Duncan’s法) Different lowercase letters indicate significant difference (P<0.05,Duncan’s test) |
取10 mL培养72 h的绿僵菌菌液,40%冷乙醇溶液淬灭后用2 mL冷甲醇提取,离心后将上清液用N2吹干,加入80 μL 20 mg/mL的甲氧基胺盐酸盐吡啶溶液,剧烈振荡30 s后,在37 ℃环境中反应90 min,反应结束后冷却至室温,然后加入80 μL BSTFA(φ为1% TMCS)衍生剂,在70 ℃条件下反应,衍生1.5 h。在气相色谱条件A下进行代谢物检测鉴定。采用该方法进行3次重复测定,根据其保留时间相对标准偏差进行重复性评价,公式如下:
| $ {\text{相对标准偏差}}=\frac{{\text{保留时间标准偏差}}}{{\text{保留时间平均值}}}\times 100{\text{%}} {\text{。}}$ |
从表2可以看出该方法检测保留时间相对标准偏差为0.000002~0.005789,均小于0.05,说明该方法稳定性和重复性较好,且精密度较高,可以满足代谢组学分析的要求。采用优化方法处理的样品经GC-MS分析后共鉴定到50种代谢物。
|
|
表 2 绿僵菌代谢物检测最适方法保留时间的重复性 Table 2 Repeatability of the retention time for the optimal method of Metarhizium metabolites detection |
代谢物分析的关键要选择高敏感性的检测手段,要求最大程度、最大范围地提取细胞内代谢物,从而更加真实的反映菌体胞内代谢情况。虽然微生物代谢组分析已经成为一种广泛使用的方法,但只使用一个已建立起来的方法进行代谢组分析所能检测出的代谢物覆盖率很低[33],目前为止还没有一种普遍适用的淬灭方法。另外丝状真菌的生理和形态与细菌、酵母菌有很大的不同,培养物通常高度黏稠且不均匀,液体培养基中有时会形成菌丝团或菌球,很难从发酵过程中获得具有代表性的样品[34]。因此不能采取与细菌、酵母菌相同的方法,需要对代谢物样品制备步骤进行系统优化。
理想的淬灭应具有以下几个条件:灭活新陈代谢的速度比样品中代谢物的转化速度更快;在淬灭步骤中保护样品的完整性;淬灭尽量不引起代谢物的化学和物理性质的变化;样品淬灭以后能够适合接下来的分析步骤;有可靠的重复性[35]。Wittmann等[36]认为微生物突然进入低于−60 ℃的环境中会产生一个冷休克的状态,但是冷甲醇会造成细胞内代谢物的大量泄漏。对于甲醇是否真的能够引起细胞代谢物泄漏的说法不一,Villas等[37]用冷甲醇处理酿酒酵母很少发生泄漏,而Maharjan[12]用冷甲醇处理谷氨酸棒杆菌胞内代谢物则易泄漏。目前大部分研究都选用60%冷甲醇溶液作为淬灭剂,也有选用甘油和乙醇作为淬灭剂的研究。Villas等[37]认为60%(φ)冷甘油−盐水溶液是淬灭酵母和细菌最好的淬灭剂。Spura等[7]发现40%(φ)的乙醇加8 g/L氯化钠溶液(−20 ℃)作为淬灭剂效果要远远好于60%甲醇溶液。本试验比较了甲醇、乙醇和甘油作为淬灭剂的效果,其中,效果最好的是40%(φ)乙醇溶液和20%(φ)甘油溶液,60%(φ)甲醇溶液效果次之。此外对于使用甘油作为淬灭剂也一直存在着争议,Villas-bôas等[26]认为在60%(φ)冷甘油加盐水溶液淬灭酵母和细菌可以很好的解决细胞泄露问题,本试验中发现,甘油虽然可以很好的保护细胞膜的完整性,但由于黏稠度较大,易被色谱柱吸附,影响检测质量,在色谱总离子图(TIC)中前端产生大片覆盖区域,不利于分离。缓冲盐对淬灭效果的影响也存在2种说法。Faijes等[9]认为添加了HEPES和 (NH4)2CO3可以维持菌体内外渗透压平衡,显著减少胞内代谢物的泄露。Canelas等[38]则认为加入缓冲盐会加速代谢物渗出。由此可见,微生物淬灭方法和淬灭剂的选择是一个艰难的过程,需要根据研究对象进行优化评价,建立适合相应微生物淬灭的方法。本试验中在淬灭剂加入几种离子盐后,D260 nm、D260 nm与对照相比较均有小幅度降低,但是多重比较结果表明与对照差异并不显著。这说明针对不同的菌种,是否需要加入缓冲盐也是应该考量的。本试验认为40%乙醇溶液不加入缓冲盐更适合作为绿僵菌代谢组的淬灭剂。
在不改变理化性质的基础上最大限度地将微生物代谢物从样品中提取出来;提取后的样品形式要与分析检测技术的需求一致;尽可能地去除干扰物质;便于浓缩[39]。提取剂的极性决定着最后获得代谢物的极性范围[40],利用提取液与代谢物的相似相容原理,可以破坏菌体细胞结构,从而提取到代谢物质。Maharjan等[12]使用6种提取剂提取大肠埃希菌代谢物,包括酸、碱、冷甲醇、热甲醇、热乙醇和甲醇+三氯甲烷,结果表明冷甲醇提取到了更多的代谢物。此外,利用热乙醇提取氨基酸和途径中间产物,简单快速且无盐加入,易蒸发和浓缩,但高温对热不稳定的代谢物有破坏作用。Castrillo等[11]用4种提取方法分别提取了酵母菌的代谢组,不同方法产生了明显不同的代谢物。Meyer等[41]对比了6种方法提取金黄色葡萄球菌代谢物,即冷甲醇、冷乙醇、冷甲醇、热乙醇、冷氯仿+水+乙醇和热水,发现冷乙醇效果最好。Canelas等[10]认为甲醇反复冻融法和酸性甲腈乙醇不适用于酵母的代谢物提取。本试验比较了绿僵菌代谢组的5种提取方法,效果最好的还是冷甲醇法,这与大部分报道是一致的。
生物体内的低极性难挥发的代谢物质如氨基酸、脂肪、糖类和核苷酸类等需要GC-MS分析时,必须先进行衍生化。目前常用的衍生化方法为肟化硅烷化衍生法[42]。肟化硅烷化衍生法实际包括2个步骤:首先是甲氧基胺与醛酮类物质的加成反应。但对于种类不同的醛酮物质影响也是不同的。对于还原性糖类物质主要影响因素为反应时间[11]。而对于α−酮酸类物质,pH是主要因素[43]。其次,肟化后需要进行全面的硅烷化反应,常用的硅烷化试剂有N−(叔丁基二甲基硅烷基)−N−甲基三氟乙酰胺(MTBSTFA)、双(三甲基硅烷基)三氟乙酰胺(BSTFA)等,通常会在使用中加入1%(φ)的三甲基氯硅(TMCS)。Kanani等[44]认为BSTFA衍生化时间对回收的代谢物种类产生较大影响,本试验选取了甲氧基胺盐酸盐37 ℃条件下肟化90 min,加入BSTFA(φ为1% TMCS)70 ℃条件下进行硅烷化,衍生时间为1.5 h时效果最好。
气相色谱主要用于代谢物质的分离,质谱则是对代谢物的相对分子质量和分子结构进行鉴定,为了能够更有效地分离样品中的代谢物,对气相色谱升温程序做了具体的优化选择,起始温度过高不利于代谢物的检测,因此本试验中色谱条件的起始温度定为50 ℃,采取程序升温法。程序开始时柱温较低,低沸点物质得到分离,中等沸点物质缓慢移动,随着柱温升高,低沸点和高沸点物质得到分离,最后升温至280 ℃,几乎所有物质都已经被气化分离。
本文对金龟子绿僵菌菌液的前处理技术及气相色谱−质谱联用分析方法进行研究,对前处理过程中的淬灭、提取和衍生时间进行了优化。所建方法对微生物主要代谢物如糖、氨基酸、醇、有机酸、烷烃、酯等均可检出。此方法简单、方便、高效,且稳定性好,重复性良好,该方法的建立有助于开展更深入的代谢机制相关研究,并可为农、林领域开展病原微生物代谢组相关研究提供参考。
| [1] |
NICHOLSON J K, LINDON J C. System biology-metabonomics[J]. Nature, 2008, 455(7216): 1054-1056. DOI:10.1038/4551054a (  0) 0) |
| [2] |
PATTI G J, YANES O, SIUZDAK G. Metabolomics: The apogee of the omics trilogy[J]. Nature Reviews Molecular Cell Biology, 2012, 13(4): 263-269. DOI:10.1038/nrm3314 (  0) 0) |
| [3] |
KARPE A V, BEALE D J, MORRISON P D, et al. Untargeted metabolic profiling of Vitis vinifera during fungal degradation
[J]. FEMS Microbiology Letters, 2015, 362(10): fnv060. (  0) 0) |
| [4] |
VIPUL S B, PRASUN B, GIRISH H R, et al. Amelioration of biomass and lipid in marine alga by an endophytic fungus Piriformospora indica[J]. Biotechnology for Biofuels, 2019, 12: 176. https://doi.org/10.1186/s13068-019-1516-6.
(  0) 0) |
| [5] |
OSMAN S M, FARIBA T, SICONG Z, et al. Correlations between LC-MS/MS-detected glycomics and NMR-detected metabolomics in Caenorhabditis elegans development
[J]. Frontiers in Molecular Biosciences, 2019(6): 49. (  0) 0) |
| [6] |
SHEN Y, FATEMEH T, TANG L, et al. Quantitative metabolic network profiling of Escherichia coli: An overview of analytical methods for measurement of intracellular metabolites
[J]. Trac-Trends in Analytical Chemistry, 2016, 75: 141-150. DOI:10.1016/j.trac.2015.07.006 (  0) 0) |
| [7] |
SPURA J, CHRISTIAN REIMER L, WIELOCH P, et al. A method for enzyme quenching in microbial metabolome analysis successfully applied to gram-positive and gram-negative bacteria and yeast[J]. Analytical Biochemistry, 2009, 394(2): 192-201. DOI:10.1016/j.ab.2009.07.016 (  0) 0) |
| [8] |
WINDER C L, DUNN W B, SCHULER S, et al. Global metabolic profiling of Escherichia coli cultures: An evaluation of methods for quenching and extraction of intracellular metabolites
[J]. Analytical Chemistry, 2008, 80(8): 2939-2948. DOI:10.1021/ac7023409 (  0) 0) |
| [9] |
FAIJES M, MARS A E, SMID E J. Comparison of quenching and extraction methodologies for metabolome analysis of Lactobacillus plantarum
[J]. Microbial Cell Factories, 2007, 6: 27. DOI:10.1186/1475-2859-6-27 (  0) 0) |
| [10] |
CANELAS A B, TEN PIERICK A, RAS C, et al. Quantitative evaluation of intracellular metabolite extraction techniques for yeast metabolomics[J]. Analytical Chemistry, 2009, 81(17): 7379-7389. DOI:10.1021/ac900999t (  0) 0) |
| [11] |
CASTRILLO J I, HAYES A, MOHAMMED S, et al. An optimized protocol for metabolome analysis in yeast using direct infusion electrospray mass spectrometry[J]. Phytochemistry, 2003, 62(6): 929-937. DOI:10.1016/S0031-9422(02)00713-6 (  0) 0) |
| [12] |
MAHARJAN P R, FERENCI T. Global metabolite analysis: The influence of extraction methodology on metabolome profiles of Escherichia coli
[J]. Analytical Biochemistry, 2003, 313(1): 145-154. DOI:10.1016/S0003-2697(02)00536-5 (  0) 0) |
| [13] |
MARCINOWSKA R, TRYGG J, WOLF-WATZ H, et al. Optimization of a sample preparation method for the metabolomic analysis of clinically relevant bacteria[J]. Journal of Microbiological Methods, 2011, 87(1): 24-31. DOI:10.1016/j.mimet.2011.07.001 (  0) 0) |
| [14] |
CAJKA T, FIEHN O. Toward merging untargeted and targeted methods in mass spectrometry-based metabolomics and lipidomics[J]. Analytical Chemistry, 2015, 88(1): 524-545. (  0) 0) |
| [15] |
ARTHURS S, DARA S K. Microbial biopesticides for invertebrate pests and their markets in the United States[J]. Journal of Invertebrate Pathology, 2019, 165: 13-21. DOI:10.1016/j.jip.2018.01.008 (  0) 0) |
| [16] |
任春光, 谭玉梅, 任秀秀, 等. 冠突曲霉veA基因缺失型与野生型的差异代谢物研究
[J]. 菌物学报, 2018, 37(2): 193-204. (  0) 0) |
| [17] |
罗飞飞, 李淑林, 陈龙云, 等. 代谢组学方法鉴定球孢白僵菌孢子萌发和杀虫毒力相关的标记物[J]. 微生物学报, 2014, 54(1): 33-41. (  0) 0) |
| [18] |
PARK S J, HYUN S, SUH H W, et al. Biochemical characterization of cultivated Cordyceps bassiana mycelia and fruiting bodies by 1H nuclear magnetic resonance spectroscopy
[J]. Metabolomics, 2013, 9(1): 236-246. DOI:10.1007/s11306-012-0442-4 (  0) 0) |
| [19] |
de BEKKER C, SMITH P B, PATTERSON A D, et al. Metabolomics reveals the heterogeneous secretome of two entomopathogenic fungi to ex vivo cultured insect tissues
[J]. PLos One, 2013, 8(8): e70609. DOI:10.1371/journal.pone.0070609 (  0) 0) |
| [20] |
SAJED T, MARCU A, RAMIREZ M, et al. ECMDB 2.0: A richer resource for understanding the biochemistry of E. coli
[J]. Nucleic Acids Research, 2016, 44(D1): D495-D501. DOI:10.1093/nar/gkv1060 (  0) 0) |
| [21] |
ZAKHARTSEV M, VIELHAUER O, HORN T, et al. Fast sampling for quantitative microbial metabolomics: New aspects on cold methanol quenching: Metabolite co-precipitation[J]. Metabolomics, 2015, 11(2): 286-301. DOI:10.1007/s11306-014-0700-8 (  0) 0) |
| [22] |
刘阳, 邓静, 吴华昌, 等. 盐胁迫对枯草芽孢杆菌发酵代谢产物的影响[J]. 食品与发酵工业, 2015, 41(7): 29-33. (  0) 0) |
| [23] |
王洪彬, 杨泓喆, 杨霁菡, 等. 枯草芽孢杆菌代谢组样品前处理方法的比较研究[J]. 分析化学, 2015, 43(8): 1169-1174. (  0) 0) |
| [24] |
LUO F, WANG Q, YIN C, et al. Differential metabolic responses of Beauveria bassiana cultured in pupae extracts, root exudates and its interactions with insect and plant
[J]. Journal of Invertebrate Pathology, 2015, 130: 154-164. DOI:10.1016/j.jip.2015.01.003 (  0) 0) |
| [25] |
TSUCHIDO T, NISHINO T, KATO Y, et al. Involvement of membrane lipids in cold shock-induced autolysis of Bacillus subtilis cells
[J]. Bioscience, Biotechnology, and Biochemistry, 1995, 59(9): 1636-1640. DOI:10.1271/bbb.59.1636 (  0) 0) |
| [26] |
VILLAS-BÔAS S G, BRUHEIM P. Cold glycerol-saline: The promising quenching solution for accurate intracellular metabolite analysis of microbial cells[J]. Analytical Biochemistry, 2007, 370(1): 87-97. DOI:10.1016/j.ab.2007.06.028 (  0) 0) |
| [27] |
明明. 基于GC-MS代谢组学方法的建立及对酿酒酵母乙醇耐受性机制研究[D]. 吉林: 吉林化工学院, 2019.
(  0) 0) |
| [28] |
胡志宏, 常旭念, 代探, 等. 基于GC-MS的灰葡萄孢菌代谢组分析[J]. 分析测试学报, 2017, 36(5): 633-639. DOI:10.3969/j.issn.1004-4957.2017.05.009 (  0) 0) |
| [29] |
郭刚, 唐丹, 田萍萍, 等. 基于GC-MS的阿维链霉菌代谢物组学研究方法的建立[J]. 生物技术通报, 2015(5): 61-67. (  0) 0) |
| [30] |
孙茂成, 李艾黎, 霍贵成, 等. 乳酸菌代谢组学研究进展[J]. 微生物学通报, 2012, 39(10): 1499-1505. (  0) 0) |
| [31] |
张剑霜, 喻浩, 钟欣, 等. 基于GC-MS代谢组学技术比较冬虫夏草与蝉花的质量[J]. 中国实验方剂学杂志, 2018, 24(18): 23-29. (  0) 0) |
| [32] |
李娟, 任路静, 孙冠男, 等. 气相色谱−质谱联用技术及其在代谢组学中的应用[J]. 生物工程学报, 2013, 29(4): 434-446. (  0) 0) |
| [33] |
WERF M J V D, OVERKAMP K M, MUILWIJK B, et al. Microbial metabolomics: Toward a platform with full metabolome coverage[J]. Analytical Biochemistry, 2007, 370(1): 17-25. DOI:10.1016/j.ab.2007.07.022 (  0) 0) |
| [34] |
VILLAS-BÔAS S G, MAS S, ÅKESSON M, et al. Mass spectrometry in metabolome analysis[J]. Mass Spectrometry Reviews, 2005, 24(5): 613-646. DOI:10.1002/mas.20032 (  0) 0) |
| [35] |
BUZIOL S, BASHIR I, BAUMEISTER A, et al. New bioreactor-coupled rapid stopped-flow sampling technique for measurements of metabolite dynamics on a subsecond time scale[J]. Biotechnology and Bioengineering, 2002, 80(6): 632-636. DOI:10.1002/bit.10427 (  0) 0) |
| [36] |
WITTMANN C, KRÖMER J O, KIEFER P, et al. Impact of the cold shock phenomenon on quantification of intracellular metabolites in bacteria[J]. Analytical Biochemistry, 2004, 327(1): 135-139. DOI:10.1016/j.ab.2004.01.002 (  0) 0) |
| [37] |
VILLAS-BÔAS S G, HØJER-PEDERSEN J, ÅKESSON M, et al. Global metabolite analysis of yeast: Evaluation of sample preparation methods[J]. Yeast, 2005, 22(14): 1155-1169. DOI:10.1002/yea.1308 (  0) 0) |
| [38] |
CANELAS A B, RAS C, TEN PIERICK A, et al. Leakage-free rapid quenching technique for yeast metabolomics[J]. Metabolomics, 2008, 4(3): 226-239. DOI:10.1007/s11306-008-0116-4 (  0) 0) |
| [39] |
ÁLVAREZ-SÁNCHEZ B, PRIEGO-CAPOTE F, CASTRO M D L D. Metabolomics analysis: II: Preparation of biological samples prior to detection[J]. Trac-Trends in Analytical Chemistry, 2010, 29(2): 120-127. DOI:10.1016/j.trac.2009.12.004 (  0) 0) |
| [40] |
KIM H K, VERPOORTE R. Sample preparation for plant metabolomics[J]. Phytochemical Analysis, 2010, 21(1): 4-13. DOI:10.1002/pca.1188 (  0) 0) |
| [41] |
MEYER H, LIEBEKE M, LALK M. A protocol for the investigation of the intracellular Staphylococcus aureus metabolome
[J]. Analytical Biochemistry, 2010, 401(2): 250-259. DOI:10.1016/j.ab.2010.03.003 (  0) 0) |
| [42] |
熊喜悦, 盛小奇, 王华, 等. 代谢组学气相色谱-质谱分析方法中样品衍生化技术的新进展[J]. 化学通报, 2015, 78(7): 602-607. (  0) 0) |
| [43] |
NASUTION U, van GULIK W M, KLEIJN R J, et al. Measurement of intracellular metabolites of primary metabolism and adenine nucleotides in chemostat cultivated Penicillium chrysogenum
[J]. Biotechnology and Bioengineering, 2006, 94(1): 159. DOI:10.1002/bit.20842 (  0) 0) |
| [44] |
KANANI H, CHRYSANTHOPOULOS P K, KLAPA M I. Standardizing GC-MS metabolomics[J]. Journal of Chromatography B, 2008, 871(2): 191-201. DOI:10.1016/j.jchromb.2008.04.049 (  0) 0) |
 2021, Vol. 42
2021, Vol. 42


