2. 赣州市农业科学研究所,江西 赣州 341000;
3. 广东省微生物信号与作物病害防控重点实验室,广东 广州 510642
2. Institute of Agricultural Sciences in Ganzhou, Ganzhou 341000, China;
3. Key Laboratory of Microbial Signals and Crop Disease Control of Guangdong Province, Guangzhou 510642, China
茄科雷尔氏菌Ralstonia solanacearum俗称青枯菌,是危害严重的世界性植物病原细菌[1],广泛分布于热带、亚热带、温带地区,寄主范围极广[2]。青枯菌侵染番茄引起的番茄青枯病,在我国南方番茄产区普遍发生且危害严重[3-4]。
青枯菌具有高度变异性及适应性,菌系分化明显,不同菌株间基因组变异性较强。明确不同来源青枯菌的致病力分化,探索青枯菌的种内遗传多样性,具有重要意义。关于青枯菌的种下分类,Hayward等[5]、华静月等[6]根据3种双糖和3种己醇的利用能力不同,划分出5个生化变种或生化型。Prior等[7]根据地理起源的密切相关性,将青枯菌分为4个演化型,即亚洲分支演化型(Ⅰ)、美洲分支演化型(Ⅱ)、非洲分支演化型(Ⅲ)和印度尼西亚分支演化型(Ⅳ),而在演化型内根据内切葡聚糖酶(Endoglucanase)基因egl序列的差异,又细分为多个序列变种(Sequevar)。目前,国际上已经鉴定出51个序列变种,来自中国的青枯菌属于演化型Ⅰ和Ⅱ,以及1、12、13、14、15、16、17、18、34、44和48等11个序列变种[8]。尽管我国有些省份对番茄青枯病菌有研究报道[9],但来自江西省的番茄青枯菌菌系研究较少。
近年来,在江西省赣南地区种植番茄、辣椒和茄子等茄科蔬菜面积扩大,发展态势迅猛,然而,由于青枯病的严重危害,给当地蔬菜生产造成了巨大的经济损失。目前,番茄青枯病的防治尚无有效的防治药剂,虽然有研究表明选择合适的噬菌体或噬菌体组合对作物青枯病具有较好的防治作用[10-12],但其应用仍受到许多限制。选育抗病或耐病品种是青枯病防治最经济有效的方法,但由于青枯菌菌系分化明显,且与寄主、环境之间构成复杂的互作关系,使得生产上推广的抗病品种往往随着种植年限的延长、青枯菌菌系致病力的变化,导致品种的抗性丧失[13-15],因此,抗病育种工作任务艰巨。分离赣南地区的番茄青枯菌菌株,建立该地区青枯菌资源库,明确青枯菌的生化变种、致病力、序列变种以及对噬菌体的敏感性,将有助于解析青枯病的发生流行机理、指导番茄抗青枯病育种和制定相关的病害防治措施。
1 材料与方法 1.1 供试培养基及噬菌体固体培养基:牛肉膏3 g·L−1,酵母膏3 g·L−1,蛋白胨3 g·L−1,硫酸镁0.25 g·L−1,磷酸氢二钾2 g·L−1,磷酸二氢钾0.5 g·L−1,蔗糖15 g·L−1,琼脂粉18 g·L−1。
半固体培养基:牛肉膏3 g·L−1,酵母膏3 g·L−1,蛋白胨3 g·L−1,硫酸镁0.25 g·L−1,磷酸氢二钾2 g·L−1,磷酸二氢钾0.5 g·L−1,蔗糖15 g·L−1,琼脂粉8 g·L−1。
LB液体培养基:酵母提取物5 g·L−1,氯化钠10 g·L−1,胰蛋白胨10 g·L−1。
生化变种鉴定基础培养基:蛋白胨1.0 g·L−1,磷酸二氢胺 1.0 g·L−1,氯化钾 0.2 g·L−1,七水硫酸镁0.2 g·L−1,溴百里酚蓝指示剂3.0 ml·L−1,pH调至7.0。
2,3,5−三苯基氯化四氮唑(TTC)培养基:水解干酪素1 g·L−1,蛋白胨10 g·L−1,甘油5 mL·L−1,琼脂粉32 g·L−1,使用前,每1 L培养基加5 mL 质量浓度为10 g·L−1的TTC溶液。
牛肉膏蛋白胨(NA)液体培养基:牛肉浸膏3 g·L−1,葡萄糖10 g·L−1,蛋白胨5 g·L−1,酵母粉0.5 g·L−1,pH调至7.0。
供试噬菌体:编号分别为P1556-1、P1556-2、P7-1、P574、P1521、P1555-L、P1555-1和P1555-M,分离自江西、广东等地作物青枯病土壤,由华南农业大学植物细菌研究室提供并保存。
1.2 青枯菌的分离纯化与保存2019—2020年,在番茄青枯病发病高峰期间,前往江西省赣南地区各市、县区采集番茄青枯病标本,用TTC选择性培养基分离青枯菌,30 ℃条件下培养24~48 h,挑取青枯菌典型单菌落,在TTC平板上划线纯化,继代3次,获得纯化菌株。用青枯菌特异性引物759(5′-GTCGCCGTCAACTCACTTTCC-3′)和760(5′-GTCGCCGTCAGCAATGCGGAATCG-3′)[7]进行PCR鉴定,将扩增得到280 bp特异目的片段的青枯菌菌株,−80 ℃甘油保存。同时,用细菌基因组DNA提取试剂盒,提取各菌株DNA,−20 ℃保存备用。
1.3 青枯菌生化变种测定参照Hayward等[5]、华静月等[6]方法。分别将乳糖、甘露醇、山梨醇和甜醇加入基础培养基中,质量浓度均为10 g·L−1,分装试管,每管4~5 mL,110 ℃条件下灭菌20 min。纤维二糖和麦芽糖经过滤灭菌后,分别加入灭菌的基础培养基中,质量浓度为10 g·L−1。每个菌株每种化合物接种3支试管,以不接种青枯菌为对照,28 ℃条件下培养21 d。
1.4 青枯菌致病性测定及数据处理参照何自福等[16]的方法,选择对青枯病表现不同抗性的5个番茄品种,分别为红圣佳二号(抗病)、金艳(中抗)、多宝(中抗)、粉霸(感病)和精棚T红(高感),作为致病性分化的鉴别品种。将健康番茄种子播于穴盘消毒基质中,待4~5片叶苗龄时用于接种。采用浸根接种方法,将番茄根部浸于1×108 CFU·mL−1青枯菌悬液中,15 min后移栽到盆钵中,每盆种2株,以浸无菌水的植株为空白对照,每个菌株接种每个番茄品种20株,定期记录发病情况,接种后第45天,各处理病情稳定,统计发病率。采用离差平均和的系统聚类方法(即Ward聚类)对数据进行聚类分析。
1.5 egl基因扩增和系统发育树构建内源葡聚糖酶基因(egl基因)扩增引物:Endo-F(5′-ATGCATGCCGCTGGTCGCCGC-3′)和Endo-R(5′-GCGTTGCCCGGCACGAACACC-3′)。PCR扩增反应程序:96 ℃预变性9 min;95 ℃变性1 min,70 ℃退火1 min,72 ℃延伸2 min,30个循环;72 ℃延伸10 min,扩增产物交由睿博兴科生物技术公司进行测序,将序列提交到GenBank数据库,并与相关序列进行比对(参考序列信息见表1)。用Clustalx和MEGA软件进行系统发育分析,采用Jukes and Cantor模型邻接法(Neighbor-joining,NJ)构建系统发育树,1000次重复Bootstrap统计学检验后构建发育树。
|
|
表 1 参考序列信息 Table 1 Referenced sequence information |
|
|
噬菌体培养:在15 mL的LB液体培养基中,将青枯菌与噬菌体按照体积比1︰1混匀,在30 ℃、180 r·min−1条件下培养过夜。
双层平板制备:取青枯菌菌液100 μL加入15 mL、50 ℃的半固体培养基,迅速混匀倒入固体培养基平板上,制成双层平板,点接3 μL噬菌体液,30 ℃条件下培养24 h,观察噬菌斑产生情况。根据青枯菌被侵染的噬菌体数量(n/个)判别青枯菌对噬菌体的敏感性,标准:n≤2,敏感性弱;3≤n≤5,敏感性中等;6≤n≤7,敏感性强;n≥8,敏感性特强。
2 结果与分析 2.1 青枯菌的分离纯化2019—2020年,在江西省赣南地区各市、县番茄种植地均有青枯病发生,采集自于都、上犹、石城、瑞金等9个县(市)的番茄青枯病病株中,共分离和鉴定出44个青枯菌株,其中于都县9个,上犹县7个,石城县4个,瑞金市2个,大余县3个,安远县3个,会昌县6个,兴国县2个,全南县8个。
2.2 青枯菌的生化变种鉴定根据对6种碳水化合物的利用情况(表2),44个番茄青枯菌可划分为生化变种Ⅲ和Ⅳ,其中,来自大余县的3个青枯菌菌株为生化变种Ⅳ,其余的41个青枯菌均为生化变种Ⅲ,表明赣南地区番茄青枯菌以生化变种Ⅲ为主。
|
|
表 2 青枯菌生化变种鉴定1) Table 2 Biovar identification of Ralstonia solanacearum |
青枯菌接种红圣佳二号(抗病)、金艳(中抗)、多宝(中抗)、粉霸(感病)、精棚T红(高感)5个抗性程度不同的番茄品种,结果表明,44个菌株在不同的番茄品种上的致病力存在明显差异(表3)。
|
|
表 3 44个青枯菌接种5个番茄品种的发病率及聚类分组 Table 3 Incidence of 44 Ralstonia solanacearum strains inoculated to five tomato cultivars and their clustering results |
采用系统聚类中的离差平均和的聚类方法(即Ward聚类),对44个菌株接种5个番茄品种的青枯病发病率结果进行聚类分析,明确青枯菌之间的致病力差异。采用离差平均和方法,样本间的距离采用欧氏距离,该距离数值的大小反映样本之间的相似度,数值越小,2个样本之间的相似度越高,数值越大,则相似度越低。本研究以欧氏距离1.2为参考标准,将44个青枯菌聚为3个组(图1)。第Ⅰ组共有29个菌株,分别来自除大余县外的8个县(市);第Ⅱ组的7个菌株分离自于都、上犹、石城、会昌4个县,该组致病力为中等强度;第Ⅲ组的8个菌株分离自于都、上犹、石城、大余、安远5个县,致病力弱,其中来自上犹县的Tm1919致病力最弱。上述结果表明,赣南地区番茄青枯菌致病力分化现象明显,在于都、上犹和石城县均存在致病力强、中、弱3种类型的菌株,但总体来看,赣南地区番茄青枯菌以致病较强的菌株占优势。
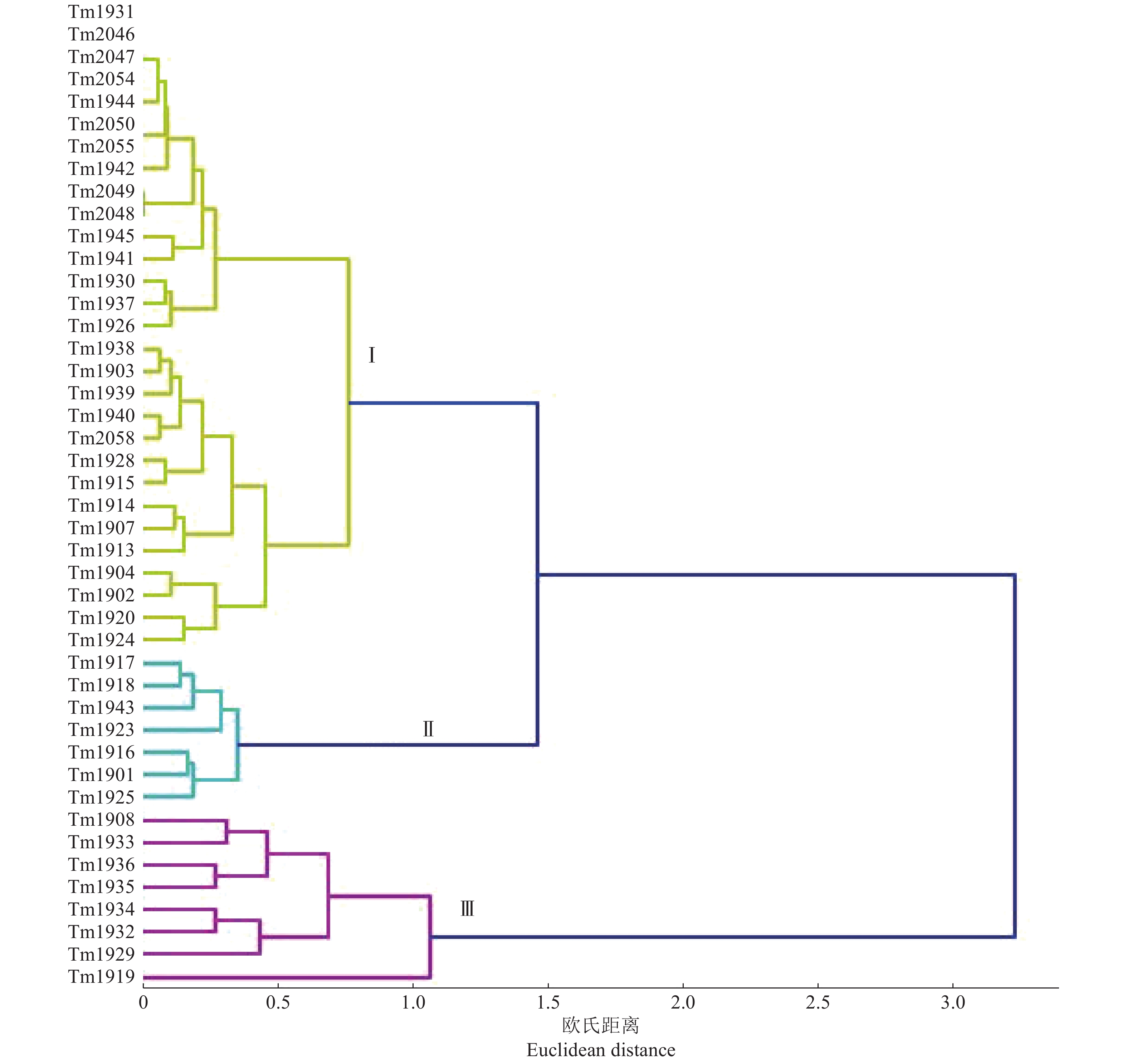
|
图 1 基于44个青枯菌接种5个番茄品种发病率的聚类分析结果 Fig. 1 Clustering results based on the incidence of 44 Ralstonia solanacearum strains inoculated to five tomato cultivars |
44个青枯菌的egl基因片段序列与标准参考菌株进行比对,构建系统发育树(图2)。结果表明,江西省赣南地区的番茄青枯菌属于亚洲分支、演化型I(PhylotypeⅠ)的8个序列变种(Sequevar)。其中Tm1929、Tm1935和Tm1937与马铃薯青枯菌JT523(留尼汪岛)属于Sequevar 13;Tm1908、Tm1920、Tm1923、Tm1924、Tm1930、Tm1931、Tm1933、Tm1934、Tm1940~Tm1943、Tm2046、Tm2054共14个菌株与来自中国的番茄青枯菌PSS8属于Sequevar 14;Tm1925、Tm1928、Tm1932、Tm2055和Tm2058与来自中国的番茄青枯菌PSS358属于Sequevar 15;Tm1913~Tm1919、Tm2047共8个菌株与来自中国的花生青枯菌P11属于Sequevar 17;Tm1901~Tm1904、Tm1907、Tm1926、Tm1938、Tm1939共8个菌株与来自法国的番茄青枯菌GMI1000均为Sequevar 18;Tm1944与来自中国台湾的番茄青枯菌PSS219为Sequevar 34;Tm1936与来自中国的烟草青枯菌Tb28为Sequevar 44;Tm1945、Tm2048~Tm2050共4个菌株与来自中国的桑青枯菌M2同为Sequevar 48。
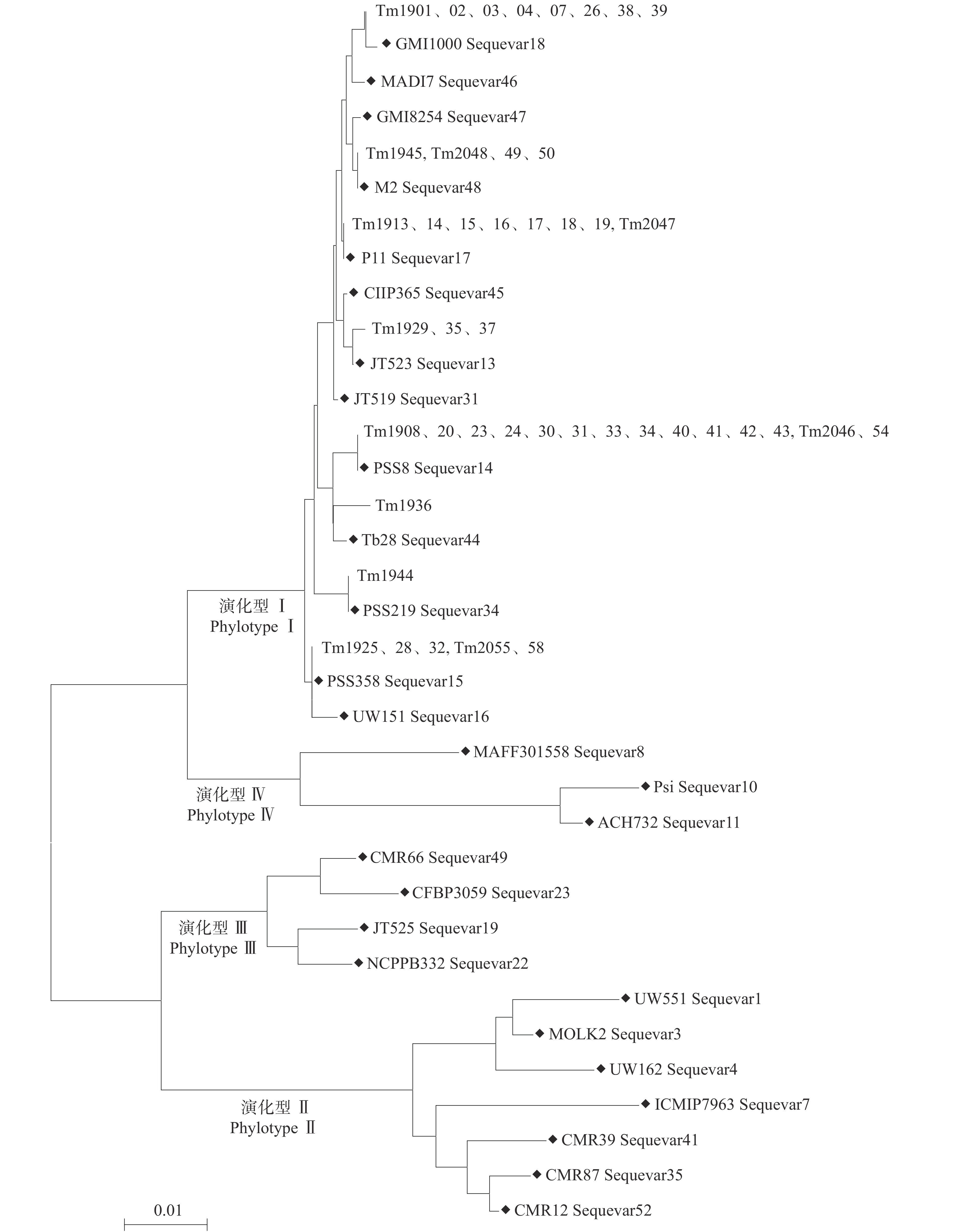
|
图 2 基于青枯菌egl基因序列的系统发育分析 Fig. 2 Phylogenetic analysis based on sequence of egl gene of Ralstonia solanacearum 将序列变种相同的菌株置于同一水平分支上,“◆”代表参考菌株 Strains with the same sequevar are settled at the same branch, “◆” indicates reference strain |
在所分离的44个番茄青枯菌中,Sequevar 14共有14个菌株,分布于于都、瑞金、大余、会昌、兴国、全南6个县(市);Sequevar 18有8个菌株,分布于会昌、石城、于都3个县;Sequevar 17有8个菌株,其中7个分布于上犹县,1个来自兴国县;Sequevar 15有5个菌株,来自于石城、大余、全南3个县;Sequevar 48的4个菌株,全部来自全南县;Sequevar 13有3个菌株,分布于石城和安远县;Sequevar 34和Sequevar 44各有1个菌株,分别来自全南和安远县。上述结果表明,赣南地区番茄青枯菌序列变种存在丰富的多样性。
2.6 青枯菌对噬菌体的敏感性测定双层平板法检测8个噬菌体裂解青枯菌的试验结果(表4)表明,44个番茄青枯菌中,只有菌株Tm2047对噬菌体的敏感性弱,有12个菌株对噬菌体的敏感性表现为中等,有4个菌株对噬菌体的敏感性较强,另有27个菌株都能被8个噬菌体裂解,其敏感性表现为特强。上述结果表明,赣南地区番茄青枯菌对噬菌体普遍表现敏感。
|
|
表 4 44个青枯菌对8个噬菌体的敏感性测定 Table 4 Sensitivity determination of 44 Ralstonia solanacearum to eight bacteriophages |
不同地理来源的青枯菌在与寄主长期协同进化的过程中,演化出明显的生理分化型或菌系多样性。国外报道,番茄青枯菌属于1号生理小种,分为演化型Ⅰ、Ⅱ、Ⅲ和Ⅳ[17-18],生化变种Ⅰ、II、III和IV[19-20],存在Sequevar 1、4、5、6、7、8、9、10、11、13、14、15、18、20、29、31、35、41、38、39、46、52等22个序列变种[21-23]。在我国,已报道的番茄青枯菌属于1号生理小种、演化型Ⅰ,分为生化变种II、III和IV,存在Sequevar13、14、15、16、17、18、34、44、48和54等10个序列变种[24-25]。曾宪铭等[26]曾测定广东省13种农作物上的129个青枯菌,其生化变种鉴定为Ⅰ、Ⅱ、Ⅲ、Ⅳ及其亚型,其中番茄青枯菌为生化变种Ⅲ和Ⅳ,郑向华等[27]也获得类似的研究结果,将番茄青枯菌鉴定为生化变种Ⅲ。2009年,Xu等[28]基于演化型分类框架,对来自我国13个省、17种不同寄主植物上的286株青枯菌进行多样性分析,明确了我国青枯病菌具有丰富的遗传多样性,其中番茄青枯菌属于演化型Ⅰ,并有Sequevar 13、14、15、16、17、18、44等7个序列变种。2011年,Xue等[24]分析了我国15个省、14种寄主植物上的319株青枯菌,基于PCR指纹图谱及演化型框架,将其中的番茄青枯菌鉴定为演化型Ⅰ,存在Sequevar 13、14、15、17、18、34、44、48等8个序列变种。
本研究对江西省赣南地区的番茄青枯菌进行了分离和菌系分析,结果显示,在分离鉴定出的44个青枯菌中,有41个菌株为生化变种Ⅲ(占93.2%),另有3个菌株属于生化变种IV,不存在生化变种I和II,与国内报道的番茄青枯菌生化变种结果基本相似。尽管我国的番茄青枯菌均属于1号生理小种,但菌株之间致病力存在差异。本研究通过接种试验,根据在5个不同青枯病抗性的番茄品种上的发病情况,将来自赣南地区的番茄青枯菌分为Ⅰ、Ⅱ、Ⅲ共3个致病类型,而且致病力较强的菌株占65.91%,15.91%的菌株致病力中等,18.18%的菌株致病力较弱,可见,赣南地区番茄青枯菌的致病力存在明显差异,而且致病力较强的菌株是本地区的优势菌群,这将为赣南地区的番茄抗青枯病育种工作提供指导。演化型和egl基因序列分析结果表明,44个番茄青枯菌株均属于演化型I,符合亚洲起源的演化型Ⅰ分类框架[7],进一步研究显示,赣南地区番茄青枯菌存在8个序列变种,分别为Sequevar 13、14、15、17、18、34、44和48,未发现Sequevar 16,其中 Sequevar 14(占31.8%)在本地区的番茄主要种植地区均有分布,表明赣南地区番茄青枯菌存在丰富的遗传多样性。
噬菌体是侵染细菌的病毒,具有高度的特异性及快速裂解宿主(寄主)的能力,可用于植物细菌病害的防治[29-31]。有研究表明,通过使用噬菌体组合的方法,能够显著降低青枯菌数量,对番茄青枯病具有较好的防治效果[32]。而测定青枯菌对噬菌体的敏感性,可为利用噬菌体防治植物青枯病提供参考。本研究结果表明,在44个青枯菌中,有61.36%的青枯菌对供试的8个噬菌体敏感,即都能被8个噬菌体裂解,表明赣南地区番茄青枯菌对噬菌体普遍表现敏感。然而,仍有12个菌株对8个噬菌体的综合敏感性表现为中等,而Tm2047菌株对噬菌体不敏感。由此可见,生产上应根据本地区青枯菌菌系的多样性及对噬菌体的敏感性,有选择性地进行噬菌体组合,以减轻番茄青枯病的发生危害。
| [1] |
冯洁. 植物病原细菌分类最新进展[J]. 中国农业科学, 2017, 50(12): 2305-2314. DOI:10.3864/j.issn.0578-1752.2017.12.011 (  0) 0) |
| [2] |
HAYWARD A C. Biology and epidemiology of bacterial wilt caused by Pseudomonas solanacearum
[J]. Annual Review of Phytopathology, 1991, 29(1): 65-87. DOI:10.1146/annurev.py.29.090191.000433 (  0) 0) |
| [3] |
乔俊卿, 陈志谊, 刘邮洲, 等. 茄科作物青枯病研究进展[J]. 植物病理学报, 2013, 43(1): 1-10. DOI:10.3969/j.issn.0412-0914.2013.01.001 (  0) 0) |
| [4] |
王杰, 龙世芳, 王正文, 等. 番茄青枯病防治研究进展[J]. 中国蔬菜, 2020, 40(1): 22-30. (  0) 0) |
| [5] |
HAYWARD A. Characteristics of Pseudomonas solanacearum
[J]. Journal of Applied Bacteriology, 1964, 27(2): 265-277. DOI:10.1111/j.1365-2672.1964.tb04912.x (  0) 0) |
| [6] |
华静月, 张长龄, 何礼远. 我国植物青枯菌的生化型和其他生理差异[J]. 植物保护学报, 1984, 11(1): 43-50. (  0) 0) |
| [7] |
PRIOR P, FEGAN M. Recent development in the phylogeny and classification of Ralstonia solanacearum
[J]. Acta Horticulturae, 2005, 695(14): 127-136. (  0) 0) |
| [8] |
徐进, 冯洁. 植物青枯菌遗传多样性及致病基因组学研究进展[J]. 中国农业科学, 2013, 46(14): 2902-2909. DOI:10.3864/j.issn.0578-1752.2013.14.006 (  0) 0) |
| [9] |
JIANG G, WEI Z, XU J, et al. Bacterial wilt in China: History, current status, and future perspectives[J/OL]. Frontiers in Plant Science, 2017, 8: 1549. [2021-01-18]. https://doi.org/10.3389/fpls.2017.01549.
(  0) 0) |
| [10] |
ADDY H S, ASKORA A, KAWASAKI T, et al. Loss of virulence of the phytopathogen Ralstonia solanacearum through infection by φRSM filamentous phages
[J]. Phytopathology, 2012, 102(5): 469-477. DOI:10.1094/PHYTO-11-11-0319-R (  0) 0) |
| [11] |
ADDY H S, ASKORA A, KAWASAKI T, et al. The filamentous phage фRSS1 enhances virulence of phytopathogenic Ralstonia solanacearum on tomato
[J]. Phytopathology, 2012, 102(3): 244-251. DOI:10.1094/PHYTO-10-11-0277 (  0) 0) |
| [12] |
LIU N, LEWIS C, ZHENG W, et al. Phage cocktail therapy: Multiple ways to suppress pathogenicity[J]. Trends in Plant Science, 2020, 25(4): 315-317. DOI:10.1016/j.tplants.2020.01.013 (  0) 0) |
| [13] |
佘小漫, 何自福. 作物青枯病研究进展[J]. 广东农业科学, 2020, 47(12): 82-89. (  0) 0) |
| [14] |
汪国平, 林明宝, 吴定华. 番茄青枯病抗性遗传研究进展[J]. 园艺学报, 2004, 31(3): 403-407. DOI:10.3321/j.issn:0513-353X.2004.03.033 (  0) 0) |
| [15] |
陈胜华. 番茄青枯病生物防治策略研究[J]. 农业与技术, 2018, 38(6): 24. (  0) 0) |
| [16] |
何自福, 虞皓, 罗方芳. 广东茄科青枯菌致病力分化及其DNA多态性分析[J]. 植物病理学报, 2003, 33(5): 415-420. DOI:10.3321/j.issn:0412-0914.2003.05.007 (  0) 0) |
| [17] |
WICKER E, GRASSART L, CORANSON-BEAUDU R, et al. Ralstonia solanacearum strains from Martinique (French West Indies) exhibiting a new pathogenic potential
[J]. Applied & Environmental Microbiology, 2007, 73(21): 6790-6801. (  0) 0) |
| [18] |
PEREZ A S, MEJIA L, FEGAN M, et al. Diversity and distribution of Ralstonia solanacearum strains in Guatemala and rare occurrence of tomato fruit infection
[J]. Plant Pathology, 2010, 57(2): 320-331. (  0) 0) |
| [19] |
NORMAN D J, ZAPATA M, GABRIEL D W, et al. Genetic diversity and host range variation of Ralstonia solanacearum strains entering North America
[J]. Phytopathology, 2009, 99(9): 1070-1077. DOI:10.1094/PHYTO-99-9-1070 (  0) 0) |
| [20] |
POUSSIER S, VANDEWALLE P, LUISETTI J. Genetic diversity of African and worldwide strains of Ralstonia solanacearum as determined by PCR-restriction fragment length polymorphism analysis of the hrp gene region
[J]. Applied and Environmental Microbiology, 1999, 65(5): 2184-2194. DOI:10.1128/AEM.65.5.2184-2194.1999 (  0) 0) |
| [21] |
CHESNEAU T, MAIGNIEN G, BOYER C, et al. Sequevar diversity and virulence of Ralstonia solanacearum phylotype Ⅰ on Mayotte Island (Indian Ocean)[J]. Frontiers in Plant Science, 2017, 8: 2209. Doi: 10.3389/fpls.2017.02209.
(  0) 0) |
| [22] |
JI P, ALLEN C, SANCHEZ-PEREZ A, et al. New diversity of Ralstonia solanacearum strains associated with vegetable and ornamental crops in Florida
[J]. Plant Disease, 2007, 91(2): 195-203. DOI:10.1094/PDIS-91-2-0195 (  0) 0) |
| [23] |
MAHBOU SOMO TOUKAN G, CELLIER G, WICKER E, et al. Broad diversity of Ralstonia solanacearum strains in Cameroon
[J]. Plant Disease, 2009, 93(11): 1123-1130. DOI:10.1094/PDIS-93-11-1123 (  0) 0) |
| [24] |
XUE Q Y, YIN Y N, YANG W, et al. Genetic diversity of Ralstonia solanacearum strains from China assessed by PCR-based fingerprints to unravel host plant-and site-dependent distribution patterns
[J]. FEMS Microbiology Ecology, 2011, 75(3): 507-519. DOI:10.1111/j.1574-6941.2010.01026.x (  0) 0) |
| [25] |
SHE X M, HE Z F, LI H P. Genetic structure and phylogenetic relationships of Ralstonia solanacearum strains from diverse origins in Guangdong Povince, China
[J]. Journal of Phytopathology, 2017, 166(3): 177-186. (  0) 0) |
| [26] |
曾宪铭, 董春. 广东农作物青枯病菌的生化型[J]. 华南农业大学学报, 1995, 16(1): 50-53. (  0) 0) |
| [27] |
郑向华, 杨帆, 邓海滨, 等. 我国植物青枯菌的生物型及RAPD分析[C]. //中国植物病理学会. 中国植物病理学会2008年学术年会论文集. 广州: 中国农业科学技术出版社, 2008: 329-337.
(  0) 0) |
| [28] |
XU J, PAN Z C, PRIOR P, et al. Genetic diversity of Ralstonia solanacearum strains from China
[J]. European Journal of Plant Pathology, 2009, 125(4): 641-653. DOI:10.1007/s10658-009-9512-5 (  0) 0) |
| [29] |
马超, 丛聪, 王丽丽, 等. 噬菌体控制植物细菌性疾病的研究进展[J]. 中国抗生素杂志, 2017, 42(9): 749-754. DOI:10.3969/j.issn.1001-8689.2017.09.006 (  0) 0) |
| [30] |
DI LALLO G, EVANGELISTI M, MANCUSO F, et al. Isolation and partial characterization of bacteriophages infecting Pseudomonas syringae pv. actinidiae, causal agent of kiwifruit bacterial canker
[J]. Journal of Basic Microbiology, 2015, 54(11): 1210-1221. (  0) 0) |
| [31] |
WEI C H, LIU J L, MAINA A, et al. Developing a bacteriophage cocktail for biocontrol of potato bacterial wilt[J]. Virologica Sinica, 2017, 32(6): 476-484. DOI:10.1007/s12250-017-3987-6 (  0) 0) |
| [32] |
WANG X F, WEI Z, YANG K M, et al. Phage combination therapies for bacterial wilt disease in tomato[J]. Nature Biotechnology, 2019, 37(12): 1513-1520. DOI:10.1038/s41587-019-0328-3 (  0) 0) |
 2022, Vol. 43
2022, Vol. 43


