石斑鱼Epinephelus spp.是亚洲许多国家最重要的经济水产养殖品种之一,其繁殖速度较快,肉质鲜嫩、营养价值高,具有相当大的经济价值。近年来,随着集约化、规模化养殖的快速发展,细菌性和病毒性传染病暴发的频率急剧增加,对石斑鱼养殖业造成了严重的危害。目前已报道的感染石斑鱼的致病性病毒主要是虹彩病毒和神经坏死病毒[1]。
新加坡石斑鱼虹彩病毒(Singapore grouper iridovirus,SGIV)隶属于虹彩病毒科Iridoviridae蛙病毒属Ranavirus,是从养殖石斑鱼体分离得到的一种高致病性病原,由SGIV引起的疾病暴发多发生在仔鱼和成年石斑鱼中,患病鱼脾脏肿大,并伴有出血等症状,病症持续数周,患病仔鱼死亡率超过90%,给养殖业造成巨大经济损失[2]。鱼类病毒性神经坏死病是由神经坏死病毒引起的一类疾病,是水产养殖过程中常见的鱼类流行性传染病之一,患病鱼苗体表异常着色,食欲不振,间歇螺旋运动,脑和视网膜有空泡化病[3-4]。而中国大多数海洋鱼类病毒性神经坏死病是由赤点石斑鱼神经坏死病毒(Red-spotted grouper nervous necrosis virus , RGNNV)引起的,对石斑鱼的成鱼以及幼鱼危害很大,严重时死亡率可达100%,严重威胁到石斑鱼养殖产业的健康发展。然而针对该病毒性疾病,目前还没找到有效的防治方法。
单核苷酸多态性(Single nucleotide polymorphism,SNP)指生物体的基因组DNA序列中单个核苷酸发生突变而产生的多态性现象,其突变形式包括位点的转换、颠换、插入及缺失等,一般情况下,等位基因的频率在同物种的群体中不小于1%[5]。SNP作为一种理想的遗传标记,是基因和表型之间的研究载体,它拉近了分子遗传标记与育种的距离,成为研究疾病、生长和繁殖性能等经济性状与相关候选基因关联性的新工具。随着SNP检测技术的进步,其在水产动物中的应用也越来越广泛,在水产经济动物中进行特定经济性状关联的SNP位点的挖掘及功能验证也越来越受到研究者们的重视。在水产动物抗病SNP分子标记研究中,高焕等[6]对中国明对虾Penaeus chinensis抗菌肽SNP位点进行了筛查,并对与白斑综合征病毒抗性的关联程度进行了分析,结果发现其SNP位点很少,且在抗感和易感群体中无明显偏向分布。Bao等[7]在海湾扇贝Argopecten irradians超氧化物歧化酶家族的3个基因的启动子和基因组DNA序列区域均检测到SNP位点,其中3个位于启动子区域的SNP位点与鳗弧菌Vibrio anguillarum的抗性显著相关。Fu等[8]在尖吻鲈Lates calcarifer的LECT2基因中发现了3个SNP位点,并发现其与瘦大肚皮病的抗性之间存在显著的相关性。
PPAR-δ(Peroxisome proliferator-activated receptors δ)是核受体超家族PPARs中的一员,是一类转录因子,其在多种生理刺激下调节基因表达,参与调节机体的脂代谢、免疫反应等多种生理过程。其中PPARs的免疫调节作用的研究得到了越来越多的关注[9-10]。在不同人体组织中,均发现PPAR-δ对炎症反应的密切调控关系[10]。除了保持稳定的炎症反应,PPAR-δ也影响干扰素信号通路,在哺乳动物免疫细胞中,PPAR-δ抑制干扰素基因以及相关细胞因子的表达[9],而在另外一些免疫细胞中PPAR-δ则会增强干扰素信号的强度,影响细胞凋亡[10-12]。前期研究也表明,鱼类PPARs在抗病毒免疫反应中发挥着重要作用[13]。
开展石斑鱼抗病品系的选育研究是解决石斑鱼病毒性病害的重要途径之一,开发抗病相关分子标记,利用分子标记定向选育石斑鱼抗病品系,可以有效避免选育的盲目性,从而加快选育进程。本研究分别针对石斑鱼虹彩病毒感染群体和神经坏死病毒感染群体进行PPAR-δ基因组DNA序列的SNP位点筛选,并对筛选到的SNP位点进行抗病毒关联分析,以期获得与抗病毒性状关联的SNP位点或基因型,为石斑鱼抗病选育提供基础资料。
1 材料与方法 1.1 试验材料斜带石斑鱼E. coioides,体质量40~60 g,体长10~14 cm,购自福建省厦门市小嶝岛水产养殖公司,在25~30 ℃的海水循环系统中驯化并饲养2周,每天以3%的体质量作为喂食量,密切监测海水的溶解氧、pH和氨浓度。
攻毒用SGIV和RGNNV分别从发病石斑鱼中分离获得,后利用实验室构建的石斑鱼脾脏细胞系(Grouper spleen, GS)进行病毒的培养与制备,GS细胞系用含有体积分数为10% 胎牛血清(Invitrogen)的Leibovitz’s L15培养基放置于28 ℃培养箱中传代培养[14]。利用TCID50法对制备的SGIV和RGNNV进行滴度测定。制备后的病毒于−80 ℃条件下保存备用。
1.2 半致死浓度的测定为了获得感染石斑鱼苗的SGIV和RGNNV的最适浓度,随机选取210尾石斑鱼苗进行浓度梯度腹腔注射感染试验,2周后统计每组累计死亡率,选取可导致50%~80%石斑鱼死亡率的SGIV和RGNNV感染浓度,进行抗感与易感群体筛选的正式试验。
试验结果显示当SGIV的滴度为1×107 TCID50/mL时,其感染鱼苗后的死亡率为72%,当RGNNV滴度为1×105 TCID50/mL时,其感染鱼苗后的死亡率为65%,因此选择上述2个病毒滴度进行正式感染试验。
1.3 石斑鱼抗感和易感群体的采集随机选取520尾鱼苗进行攻毒感染试验,共分为3组,对照组40条鱼腹腔注射100 μL PBS溶液,SGIV感染组240尾鱼腹腔注射100 μL病毒滴度为1×107 TCID50/mL的SGIV溶液,RGNNV感染组240尾鱼腹腔注射100 μL病毒滴度为1×105 TCID50/mL的RGNNV溶液,感染后每隔6 h观察1次鱼苗死亡情况,参考前期研究中评判抗感和易感样品的标准[15-16],将感染后5 d内死亡的个体作为易感个体,感染后14 d后还存活的个体作为抗感个体,采集易感、抗感个体的尾鳍样品,于样品稳定剂(TaKaRa)中,−20 ℃保存备用。
1.4 基因组DNA提取基因组DNA提取使用Trelief ™ Animal Genomic DNA Kit(TsingKe)试剂盒,具体方法步骤参考试剂盒说明书,并用15 g/L琼脂糖凝胶电泳检测基因组DNA提取质量。
1.5 石斑鱼PPAR-δ基因组DNA序列的克隆与SNP位点筛选以斜带石斑鱼鳍条DNA为模板,根据石斑鱼基因组中的PPAR-δ基因gDNA序列,利用Primer Primer 5.0软件设计引物(表1),由北京擎科生物有限公司合成,以上述易感和抗感个体基因组DNA为模板进行PCR扩增。PCR反应体系和反应条件参考文献[15]。PCR扩增产物经胶回收以后,克隆至 pMD19-T 载体,抗感组和易感组各送5个阳性克隆样品至北京擎科生物有限公司进行测序。测序结果使用MEGA 6.0软件进行多重比对,观察测序结果峰图,预测SNP 位点。同一位点不同碱基出现比例大于30%则认定为 SNP 位点。
|
|
表 1 PPAR-δ基因组DNA序列扩增引物 Table 1 Primers for PPAR-δ genomic DNA sequence amplification |
依据上述方法预测的PPAR-δ基因SNP位点,采用SNaPshot法[17],分别对SGIV感染组和RGNNV感染组的易感样品和抗感样品进行SNP分型,其中SGIV易感组、SGIV抗感组、RGNNV易感组和RGNNV抗感组的样品数目均为50尾。SNaPshot分型由北京擎科生物有限公司完成。
使用Popgen 32和PIC软件分析SNP位点的观测杂合度(Observed heterorozygosity, Ho)、期望杂合度(Expected heterozygosity, He)、等位基因频率、有效等位基因数(Effective number of alleles, Ne)、哈迪−温伯格平衡(Hardy-Weinberg equilibrium),使用Cervus3.0软件计算位点的多态信息含量(Polymorphism information content, PIC)等。使用Haploview 4.2软件进行各个SNP位点间的连锁不平衡和单倍型的分析。使用SPSS17软件中的χ2检验对SNP位点在抗病组和易感组群体中的基因型频率和单倍型频率进行差异显著性分析。
2 结果与分析 2.1 石斑鱼PPAR-δ基因结构图以及筛查的SNP位点信息根据石斑鱼基因组中的PPAR-δ基因gDNA序列和转录组中的cDNA序列,以斜带石斑鱼鳍条DNA为模板,使用4对引物(表1)对PPAR-δ基因的内含子及外显子序列进行扩增,对克隆进行测序比对,绘制PPAR-δ基因结构图(图1)。以易感组和抗感组各5个基因组DNA样品作为模板进行PCR扩增,对测序结果进行多重比对分析。在SGIV感染的易感组和抗感组样品中共筛查到9个SNP位点,分别将其命名为SNP-S1(g.940T>A)、SNP-S2(g.994C>G)、SNP-S3(g.1644A>C)、SNP-S4(g.2541C>T)、SNP-S5(g.4188A>T)、SNP-S6(g.4479G>A)、SNP-S7(g.4595T>A)、SNP-S8(g.4791T>A)和SNP-S9(g.4852T>A),所有SNP位点均位于内含子中(图1A)。在RGNNV感染的易感组和抗感组样品中共筛查到8个SNP位点,分别将其命名为SNP-N1(g.324G>A)、SNP-N2(g.883A>G)、SNP-N3(g.1140A>T)、SNP-N4(g.1702A>T)、SNP-N5(g.2510C>T)、SNP-N6(g.5253G>C)、SNP-N7(g.5522G>A)和SNP-N8(g.5564T>C),其中SNP-N1位于外显子中,属于同义突变,没有改变编码的氨基酸,其余SNP均位于内含子中(图1B)。
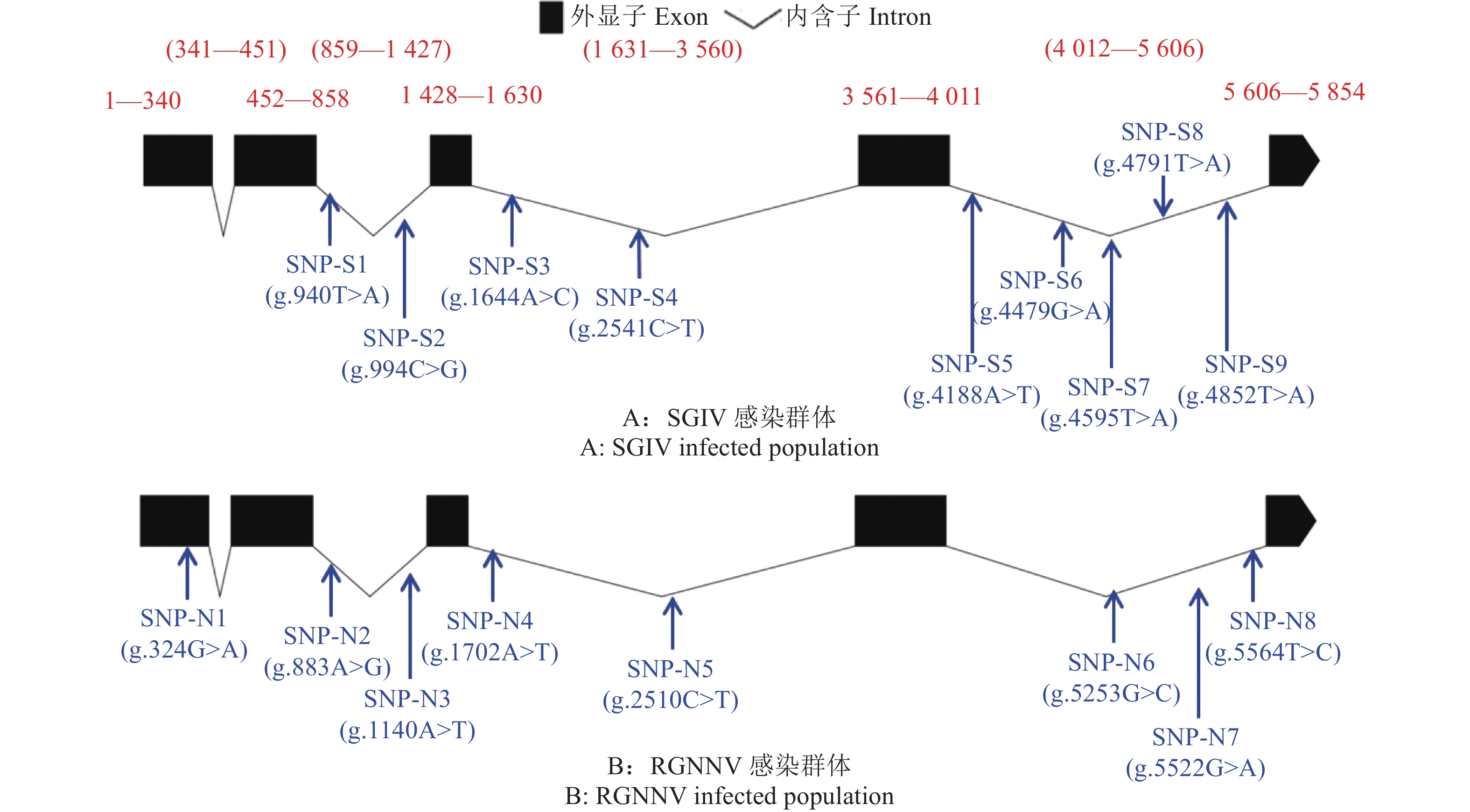
|
图 1 石斑鱼PPAR-δ基因结构及SNP位点 Fig. 1 DNA structure of PPAR-δ gene and its SNP loci of grouper |
采用SnaPshot分型法对石斑鱼群体进行基因分型分析。在SGIV感染组中,100个送样样品中共获得100个有效结果,其中SGIV抗感组50个,SGIV易感组50个。使用Popgen32和PIC软件对9个SNP位点在SGIV抗感组和易感组中的遗传多态性进行分析,结果(表2)显示:在SGIV抗感组中,PIC的范围为0.136~0.370,SNP-S1属于低度多态(PIC<0.25),其余SNP位点属于中度多态(0.25≤PIC<0.50);在SGIV易感组中,PIC的范围为0.177~0.375,其中SNP-S1属于低度多态(PIC<0.25),其余SNP位点属于中度多态(0.25≤PIC<0.50)(表2)。χ2检验结果表明,在SGIV抗感组中,除SNP-S4、SNP-S5、SNP-S6、SNP-S7、SNP-S8、SNP-S9不符合Hardy-Weinberg平衡外(P<0.05),其余SNP均处于Hardy-Weinberg平衡状态(P>0.05);在SGIV易感组中,除SNP-S9不符合Hardy-Weinberg平衡外(P<0.05),其余SNP均处于Hardy-Weinberg平衡状态(P>0.05)。
|
|
表 2 9个SNP在石斑鱼SGIV抗感组和易感组中的遗传多态性信息 Table 2 Genetic polymorphisms of nine SNPs in SGIV resistant and susceptible grouper groups |
采用SnaPshot分型法对石斑鱼群体进行基因分型分析。在RGNNV感染组中,100个送样样品中共获得97个有效结果,其中RGNNV抗感组48个,RGNNV易感组49个。使用Popgen32和PIC软件对8个SNP位点在抗感组和易感组中的遗传多态性进行分析,结果(表3)显示:在RGNNV抗感组中,PIC的范围为0.136~0.358,SNP-N1和SNP-N2属于低度多态(PIC<0.25),其余SNP属于中度多态(0.25≤PIC<0.50);在RGNNV易感组中,PIC的范围为0.106~0.317,其中SNP-N1和SNP-N2属于低度多态(PIC<0.25),其余SNP属于中度多态(0.25≤PIC<0.50)。χ2检验结果表明,在RGNNV抗感组中,SNP-N1、SNP-N2和SNP-N4处于Hardy-Weinberg平衡状态(P>0.05),其余SNP均不符合Hardy-Weinberg平衡(P<0.05);在RGNNV易感组中,除SNP-N3不符合Hardy-Weinberg平衡外(P<0.05),其余SNP均处于Hardy-Weinberg平衡状态(P>0.05)。
|
|
表 3 8个SNP在石斑鱼RGNNV抗感组和易感组中的遗传多态性信息 Table 3 Genetic polymorphisms of eight SNPs in RGNNV resistant and susceptible grouper groups |
PPAR-δ基因在SGIV抗感组和易感组的SNP统计分析结果(表4)显示:SNP-S7基因型频率在抗感组和易感组中存在显著差异分布(P<0.05);SNP-S7的TT基因型在抗感组中分布频率为60%,易感组中分布频率为40%,AT基因型在抗感组中分布频率为24%,易感组中分布频率为52%,而AA基因型在抗感组中分布频率为16%,在易感组中分布频率为8%,表明TT和AA这2种纯合子基因型与SGIV抗感性状相关,而AT杂合子基因型与SGIV易感性相关。
|
|
表 4 石斑鱼PPAR-δ基因在2种病毒抗感组和易感组的SNP统计分析 Table 4 PPAR-δ gene SNPs analysis in resistant and susceptible grouper groups infected by two viruses |
使用Haploview 4.2软件中的Four Gamete Rule计算方法对9个SNP位点进行连锁不平衡分析,结果显示:SNP-S4、SNP-S5和SNP-S6这3个位点高度连锁,构成单倍块1,可形成TTT、AAA、TAA和TAT共4个单倍型;SNP-S7、SNP-S8和SNP-S9这 3个SNP位点高度连锁,构成单倍块2,可形成CAG、TTA和CAA共3个单倍型(图2)。将上述单倍型与石斑鱼抗虹彩病毒抗性性状进行关联分析,结果显示上述单倍型与石斑鱼抗虹彩病毒抗性性状均无显著的相关性(P>0.05)(表5)。
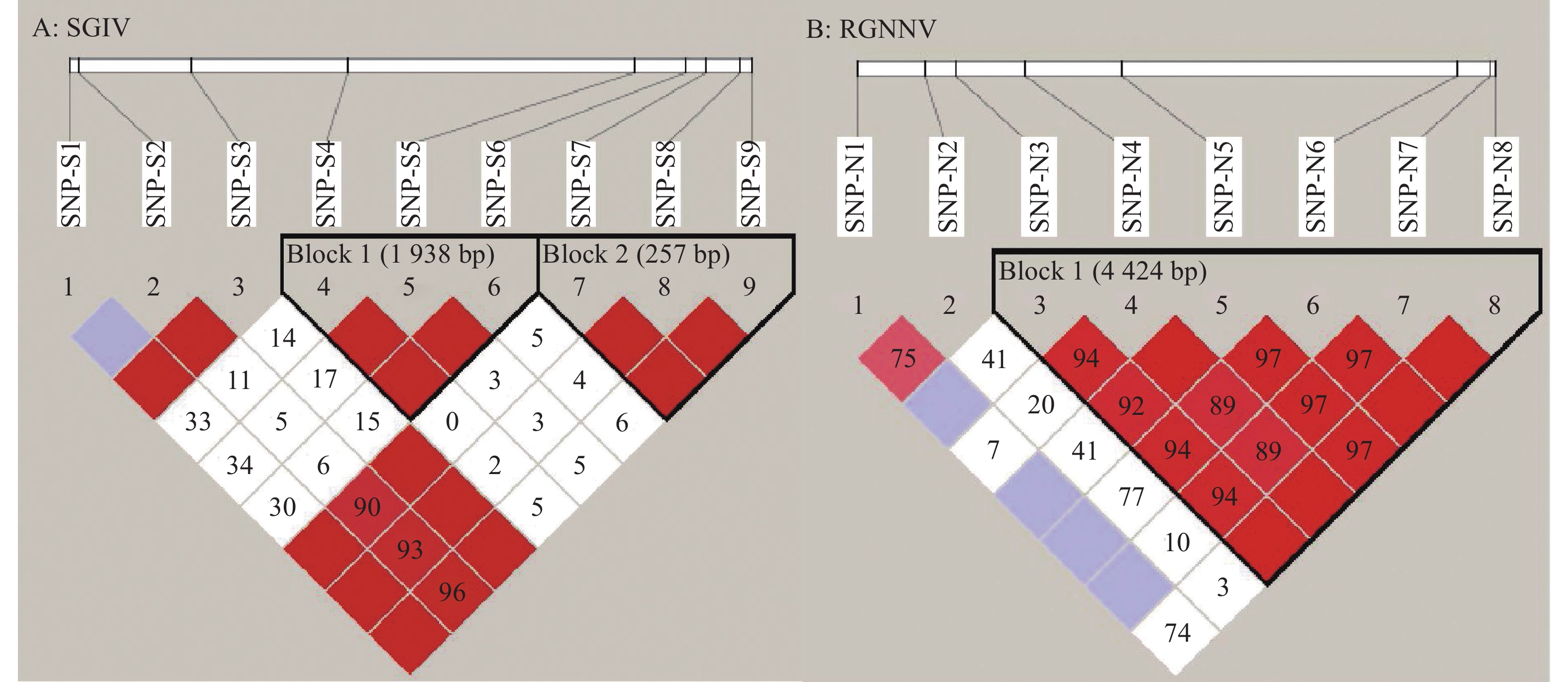
|
图 2 石斑鱼SGIV和RGNNV感染组中筛选出PPAR-δ基因SNP标记的连锁不平衡分析 Fig. 2 Linkage disequilibrium analysis of PPAR-δ SNP markers screened from SGIV and RGNNV infected groupers 图片顶部的白色长方形代表PPAR-δ基因组DNA序列,黑色细线对应标签代表各SNP位点;图片下半部分的正方形色块内的数值为连锁不平衡参数r2;正方形色块的颜色代表两两SNP位点间的连锁不平衡参数D的范围,白色表示D=0,红色和粉色表示0 < D < 1,蓝色表示D=1;黑框代表该区域内相邻的SNP位点处于连锁不平衡状态,形成单倍型块,用Block标记,括号内的数值表示单倍型块碱基的长度 The white rectangle at the top of image represents the PPAR-δ genome DNA sequence, and the black thin lines and corresponding tags represent the SNP sites; In the bottom half of the picture, the value in the square color block is the linkage unbalance parameter r2; The color of the square color block represents the range of linkage unbalance parameter D between two SNP sites, white represents D=0, red and pink represent 0 < D < 1, and blue represents D=1; The black box indicates that the adjacent SNP sites in this region are in the linkage unbalanced state and form a haplotype block, which is marked by Block, and the value in brackets indicates the length of haplotype block base |
|
|
表 5 石斑鱼PPAR-δ基因SNP连锁单倍型与2种病毒抗性的关联分析 Table 5 Association analysis between PPAR-δ SNPs linked haplotype and resistance of two viruses |
PPAR-δ基因在RGNNV抗感组和易感组的SNP统计分析结果(表4)显示:SNP-N5基因型频率在易感组和抗感组中存在显著差异分布(P<0.05);SNP-N5的CC基因型在易感组中分布频率为57.1%,在抗感组中分布频率为37.5%,表明CC基因型与RGNNV易感性状相关;而CT基因型在易感组中分布频率为38.8%,在抗感组中分布频率为60.4%,表明CT基因型与RGNNV抗性相关。
对8个SNP位点进行连锁不平衡分析,结果(表5)显示:SNP-N3、SNP-N4、SNP-N5、SNP-N6、SNP-N7和SNP-N8 这6个SNP位点高度连锁,构成单倍块1,可形成TACGGT、AGTCAC、TGCGGT、TACCAT、AGTCAT、AACGGT和TGTCAT共7个单倍型(图2)。将上述单倍型与石斑鱼抗神经坏死病毒抗性性状进行关联分析,结果显示7个单倍型与石斑鱼抗神经坏死病毒抗性性状均无显著的相关性(P>0.05)。
3 讨论与结论 3.1 PPAR-δ基因的SNP位点分布分析在本研究中,在石斑鱼对SGIV的易感和抗感群体中共筛查到9个SNP位点,全部位于内含子区域,在石斑鱼对RGNNV的易感和抗感群体中共筛查到8个SNP位点,除SNP-N1位于外显子区域外,其余均分布在内含子区域,且SNP-N1产生的突变为同义突变,对氨基酸编码并不造成影响。尽管对SNP的研究表明,SNP位点既可能发生在编码区也可能发生在非编码区,但其发生在非编码区的机率更大,这是由于相较于具有编码功能的外显子,内含子在进化上受到的压力更小一些,故内含子的核苷酸多态性和变异度会明显高于外显子[18]。尽管内含子不参与基因的编码功能,但其在机体中仍发挥着重要作用,内含子对机体的mRNA前体起着调节其剪接、运输和降解的作用,此外,近年来的研究还表明内含子对基因的表达也起着重要的调控作用[19]。因此我们推测,本研究中在PPAR-δ基因内含子中发现的SNP位点,也许与该基因的转录调控相关,其不同突变体通过影响PPAR-δ的转录调控来发挥免疫调控功能,从而影响宿主的抗病性状,在我们今后的研究中,会对该机制进一步深入分析。
3.2 SNP位点在不同群体中的多态性分析Botstein等[20]对SNP位点进行分类时提出,当PIC大于0.50时,该位点为高度多态性位点;PIC大于或等于0.25而小于0.50时为中度多态性位点;PIC小于0.25时为低度多态性位点。对本试验中发现的SNP位点进行多态性分析,结果显示石斑鱼PPAR-δ基因SNP位点在抗感组和易感组中均表现为低度或中度多态,并未出现高度多态位点。而在草鱼、罗非鱼等的免疫基因SNP位点分析中也发现,中度多态性位点较多,低度和高度多态性位点偏少,造成这一现象的原因可能是由于SNP位点为典型的二等位基因表达,本身多态性不高[21],此外,用于筛选抗病性状的群体会在养殖过程中经过自然选择和人工选择,淘汰掉抗病能力差的个体,导致群体多样性下降,因此SNP位点的整体多态性不高。
Hardy-Weinberg平衡分析表明,在不同感染组群体中均检测到不符合Hardy-Weinberg平衡的SNP位点,由于本研究中的试验群体均来自养殖场,而养殖场的亲本是经过了长期人工选育过程而获得的,因此群体遗传多态性在选育过程中会逐渐降低,从而导致部分SNP偏离Hardy-Weinberg平衡。
3.3 SNP位点与病毒抗性性状的关联分析SNP是第3代分子标记技术,其具有分布广泛、遗传稳定性高,易于分型等特点。近年来,SNP标记技术在水产动物育种中得到广泛应用[22]。在草鱼中,采用Snapshot法筛选与草鱼呼肠弧病毒(GCRV)抗感/易感性状关联的SNP位点,结果表明在RIG-I(视黄酸诱导基因蛋白I)、TLR3(Toll样受体3)和补体C6等许多重要免疫基因中均筛选到多个抗病相关SNP位点和单倍型,可以作为草鱼抗出血病品系的构建提供技术手段[23-25]。在罗非鱼中,同样利用Snapshot分型法,在LBP(脂多糖结合蛋白)、NOD1(核苷酸结合和寡聚化结构域1)和MCP-8(肥大细胞蛋白酶−8)等基因中筛选出多个与无乳链球菌抗性关联的SNP位点和单倍型[26-28]。此外,基于高通量测序,全基因组数据库分析等其他技术手段也分别在异育银鲫、牙鲆、虹鳟、鲤鱼等重要经济鱼类中分别筛选获得与鲫疱疹病毒(CaHV),鳗弧菌、传染性造血器官坏死病毒(IHNV)和鲤疱疹病毒(CyHV-3)等病原抗性关联的SNP位点。
在PPARs基因SNP分子标记研究方面,目前在人、鼠以及牦牛上已有相关研究,而在低等脊椎动物上的研究鲜有报道。有研究对黑线仓鼠繁殖相关基因PPAR-δ部分序列进行SNP分析,发现其外显子区域发生碱基突变的概率较大,与人类研究结果一致,猜测与其功能有密切关系[29]。PPARs基因作为调控牦牛肌内脂肪沉积的相关候选基因,分析其基因多态性,并与生长性状进行关联分析,为牦牛分子育种提供了科学依据[30]。
在本试验中,在SGIV感染组中筛选到9个SNP位点,与抗病性状的关联分析结果表明,SNP-S7基因型频率在易感组和抗感组中存在显著差异分布(P<0.05)。该位点的TT和AA 2种纯合子基因型与SGIV抗感性状相关,而AT杂合子基因型与SGIV易感性相关。对9个SNP位点进行连锁不平衡分析,结果显示:SNP-S4、SNP-S5和SNP-S6这3个位点高度连锁,构成单倍块1,可形成TTT、AAA、TAA和TAT共4个单倍型;SNP-S7、SNP-S8和SNP-S9这 3个SNP位点高度连锁,构成单倍块2,可形成CAG、TTA和CAA共3个单倍型。将上述单倍型与石斑鱼抗虹彩病毒抗性性状进行关联分析,结果显示上述单倍型与石斑鱼抗虹彩病毒抗性性状均无显著的相关性(P>0.05)。在RGNNV感染群体中筛选到8个SNP位点,与抗病性状进行关联分析结果表明,SNP-N5(g.2510C>T)基因型频率在抗感组和易感组中存在显著差异分布(P<0.05),该位点的CC基因型与RGNNV易感性状相关,CT基因型则与RGNNV抗性相关。连锁不平衡分析结果显示,SNP-N3、SNP-N4、SNP-N5、SNP-N6、SNP-N7和SNP-N8这6个SNP位点高度连锁,构成单倍块1,可形成TACGGT、AGTCAC、TGCGGT、TACCAT、AGTCAT、AACGGT和TGTCAT共7个单倍型。将上述单倍型与石斑鱼抗RGNNV性状进行关联分析,结果显示7个单倍型与石斑鱼抗神经坏死病毒抗性性状均无显著的相关性(P>0.05)。上述研究结果中筛选出来的与抗SGIV和RGNNV关联的SNP位点及其基因型可以作为石斑鱼抗病品种选育的候选分子标记。
在本试验中,虽然抗性关联SNP位点与其他位点形成了高度连锁的单倍块,但并没有单倍型与抗病性状显著关联,通常情况下,将位于1条染色体上某一区域的1组相关联的多个SNP等位位点称作为单倍型,其经常被用于基因组关联研究、复杂的致病基因或者重要经济性状基因的定位以及多态性筛选研究中,相较于单个SNP位点的分析,其效应分析更为可靠,基因型检测工作也更为高效[31]。因此本研究这些单倍块暂时无法用于石斑鱼抗病品系的构建工作中。
3.4 总结本研究分析了石斑鱼PPAR-δ基因组多态性及其与SGIV和RGNNV抗感/易感性状的相关性,研究发现SNP-N5(g.2510C>T)与石斑鱼RGNNV抗性显著相关,SNP-S7(g.4595T>A)与SGIV抗性显著相关(P<0.05),该研究结果为石斑鱼病毒性疾病的抗病选育工作提供了理论基础和技术支持。
| [1] |
罗鸣, 陈傅晓, 刘龙龙, 等. 我国石斑鱼养殖疾病的研究进展[J]. 水产科学, 2013, 32(9): 549-554. DOI:10.3969/j.issn.1003-1111.2013.09.010 (  0) 0) |
| [2] |
QIN Q W, CHANG S F, NGOH-LIM G H, et al. Characterization of a novel ranavirus isolated from grouper Epinephelus tauvina
[J]. Diseases of Aquatic Organisms, 2003, 53(1): 1-9. (  0) 0) |
| [3] |
COSTA J Z, THOMPSON K D. Understanding the interaction between Betanodavirus and its host for the development of prophylactic measures for viral encephalopathy and retinopathy[J]. Fish and Shellfish Immunology, 2016, 53: 35-49. DOI:10.1016/j.fsi.2016.03.033 (  0) 0) |
| [4] |
黄剑南, 郭志勋, 冯娟, 等. 鱼类诺达病毒及其所导致的疾病[J]. 水产学报, 2006, 30(6): 831-836. (  0) 0) |
| [5] |
VIGNAL A, MILAN D, SANCRISTOBAL M, et al. A review on SNP and other types of molecular markers and their use in animal genetics[J]. Genetics Selection Evolution, 2002, 34(3): 275-305. DOI:10.1186/1297-9686-34-3-275 (  0) 0) |
| [6] |
高焕, 赖晓芳, 孟宪红, 等. 中国明对虾抗菌肽基因应答WSSV侵染的表达及其SNP分析[J]. 中国水产科学, 2011, 18(3): 646-653. (  0) 0) |
| [7] |
BAO Y, LI L, ZHANG G. Polymorphism of the superoxide dismutase gene family in the bay scallop (Argopecten irradians) and its association with resistance/susceptibility to Vibrio anguillarum
[J]. Developmental and Comparative Immunology, 2010, 34(5): 553-561. DOI:10.1016/j.dci.2009.12.016 (  0) 0) |
| [8] |
FU G H, BAI Z Y, XIA J H, et al. Characterization of the LECT2 gene and its associations with resistance to the big belly disease in Asian seabass
[J]. Fish and Shellfish Immunology, 2014, 37(1): 131-138. DOI:10.1016/j.fsi.2014.01.019 (  0) 0) |
| [9] |
DUNN S E, BHAT R, STRAUS D S, et al. Peroxisome proliferator-activated receptor delta limits the expansion of pathogenic Th cells during central nervous system autoimmunity[J]. Journal of Experimental Medicine, 2010, 207(8): 1599-1608. DOI:10.1084/jem.20091663 (  0) 0) |
| [10] |
SUN L, SHI Y, WANG G, et al. PPAR-delta modulates membrane cholesterol and cytokine signaling in malignant B cells
[J]. Leukemia, 2018, 32(1): 184-193. DOI:10.1038/leu.2017.162 (  0) 0) |
| [11] |
NEEL J G, GRIMALDI P A. Physiological functions of peroxisome proliferator-activated receptor beta[J]. Physiological Reviews, 2014, 94(3): 795-858. DOI:10.1152/physrev.00027.2013 (  0) 0) |
| [12] |
JI J D, KIM H J, RHO Y H, et al. Inhibition of IL-10-induced STAT3 activation by 15-deoxy-Delta12, 14-prostaglandin J2[J]. Rheumatology, 2005, 44(8): 983-988. DOI:10.1093/rheumatology/keh657 (  0) 0) |
| [13] |
WANG Y, YU Y, WANG Q, et al. PPAR-δ of orange-spotted grouper exerts antiviral activity against fish virus and regulates interferon signaling and inflammatory factors
[J]. Fish & Shellfish Immunology, 2019, 94: 38-49. (  0) 0) |
| [14] |
HUANG X, HAUNG Y, SUN J, et al. Characterization of two grouper Epinephelus akaara cell lines: Application to studies of Singapore grouper iridovirus (SGIV) propagation and virus-host interaction
[J]. Aquaculture, 2009, 292(3/4): 172-179. (  0) 0) |
| [15] |
YANG M, WEI J, LI P, et al. MHC polymorphism and disease resistance to Singapore grouper iridovirus (SGIV) in the orange-spotted grouper, Epinephelus coioides
[J]. Science Bulletin, 2016, 61(9): 693-699. DOI:10.1007/s11434-016-1055-5 (  0) 0) |
| [16] |
YANG M, WANG Q, CHEN J, et al. Identification of candidate SNPs and genes associated with anti-RGNNV using GWAS in the red-spotted grouper, Epinephelus akaara
[J]. Fish & Shellfish Immunology, 2021, 112: 31-37. (  0) 0) |
| [17] |
NORTON N, WILLIAMS N M, WILLIAMS H J, et al. Universal, robust, highly quantitative SNP allele frequency measurement in DNA pools[J]. Human Genetics, 2002, 110(5): 471-478. DOI:10.1007/s00439-002-0706-6 (  0) 0) |
| [18] |
PARENTEAU J, ABOU ELELA S. Introns: Good day junk is bad day treasure[J]. Trends in Genetics, 2019, 35(12): 923-934. DOI:10.1016/j.tig.2019.09.010 (  0) 0) |
| [19] |
SHAUL O. How introns enhance gene expression[J]. International Journal of Biochemistry & Cell Biology, 2017, 91: 145-155. (  0) 0) |
| [20] |
BOTSTEIN D, WHITE R L, SKOLNICK M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics, 1980, 32(3): 314-331. (  0) 0) |
| [21] |
HUBERT S, BUSSEY J T, HIGGINS B, et al. Development of single nucleotide polymorphism markers for Atlantic cod (Gadus morhua) using expressed sequences
[J]. Aquaculture, 2009, 296(1/2): 7-14. (  0) 0) |
| [22] |
周欣, 高风英, 卢迈新. 鱼类抗病育种研究进展[J]. 大连海洋大学学报, 2021, 36(3): 510-523. DOI:10.16535/j.cnki.dlhyxb.2020-156 (  0) 0) |
| [23] |
WAN Q Y, SU J G, CHEN X H, et al. Genomic sequence comparison, promoter activity, SNP detection of RIG-I gene and association with resistance / susceptibility to grass carp reovirus in grass carp (Ctenopharyngodon idella)
[J]. Developmental & Comparative Immunology, 2013, 39(4): 333-342. (  0) 0) |
| [24] |
HENG J F, SU J G, HUANG T, et al. The polymorphism and haplotype of TLR3 gene in grass carp (Ctenopharyngodon idella) and their associations with susceptibility / resistance to grass carp reovirus
[J]. Fish & Shellfish Immunology, 2011, 30(1): 45-50. (  0) 0) |
| [25] |
SHEN Y B, ZHANG J B, XU X Y, et al. A new haplotype variability in complement C6 is marginally associated with resistance to Aeromonas hydrophila in grass carp
[J]. Fish & Shellfish Immunology, 2013, 34(5): 1360-1365. (  0) 0) |
| [26] |
高风英, 卢迈新, 曹建萌, 等. 尼罗罗非鱼 NOD1 基因 SNP 位点和单倍型与抗无乳链球菌感染的关联分析
[J]. 农业生物技术学报, 2018, 26(11): 1949-1961. (  0) 0) |
| [27] |
FU G H, LIU F, XIA J H, et al. The LBP gene and its association with resistance to Aeromonas hydrophila in tilapia
[J]. International Journal of Molecular Sciences, 2014, 15(12): 22028-22041. DOI:10.3390/ijms151222028 (  0) 0) |
| [28] |
FU G H, WAN Z Y, XIA J H, et al. The MCP-8 gene and its possible association with resistance to Streptococcus agalactiae in tilapia
[J]. Fish & Shellfish Immunology, 2014, 40(1): 331-336. (  0) 0) |
| [29] |
LATRUFFE N, VAMECQ J. Peroxisome proliferators and peroxisome proliferator activated receptors (PPARs) as regulators of lipid metabolism[J]. Biochimie, 1997, 79(2/3): 81-94. (  0) 0) |
| [30] |
纪永吉. 牦牛PPARα、PPARγ、PPARδ基因的多态性与生长性状相关性研究[D]. 兰州: 甘肃农业大学, 2016.
(  0) 0) |
| [31] |
丁克越. 不同单倍型块定义在单倍型块结构推断与htSNPs选择中的效应[D]. 北京: 中国协和医科大学, 2004.
(  0) 0) |
 2023, Vol. 44
2023, Vol. 44


