东方蜜蜂微孢子虫Nosema ceranae是一种专性侵染成年蜜蜂中肠上皮细胞的单细胞真菌病原,对蜂王、工蜂、雄蜂和幼虫均有感染性[1]。东方蜜蜂微孢子虫侵染能引起蜜蜂中肠上皮细胞结构破坏、细胞凋亡和免疫应答抑制,哺育力下降,寿命缩短,严重影响蜂群群势和生产力[2]。目前,东方蜜蜂微孢子虫的参考基因组[3]和全长转录组[4]均已公布,为深入开展相关分子生物学研究奠定了基础。
染色体结构维持(Structural maintenance of chromosome,SMC)蛋白在细菌、古生菌和真核生物中广泛存在,可直接参与染色体的形成与结构维持等动态变化及DNA的复制、重组和修复等过程,因而对染色质结构的组织、细胞分裂过程中遗传物质的准确分离等均具有关键作用[5]。在水稻中,SMC1和SMC3-1基因被证实参与DNA双链断裂损伤修复和有丝分裂等生物学过程[6]。作为SMC复合体蛋白的成员之一,凝聚蛋白复合体Ⅰ通过分级折叠启动早期的染色质凝聚,起到塑造、稳定染色体的作用[7]。敲除凝聚蛋白复合体Ⅰ或Ⅱ的特异亚基均能引起中期染色体结构的明显异常[8]。然而对于东方蜜蜂微孢子虫,SMC相关研究至今依然缺失。
近期,笔者团队测定了nce-miR-15325及其靶向的核凝聚复合体亚基(Nuclear condensin complex subunit,NCCS)基因在东方蜜蜂微孢子虫侵染意大利蜜蜂工蜂过程中的表达谱,并发现nce-miR-15325与NCCS具有相似的表达规律。本研究利用生物信息学方法解析东方蜜蜂微孢子虫NCCS编码的SMC的分子特性,预测和分析东方蜜蜂微孢子虫和其他物种SMC的保守基序和结构域,并进行系统进化分析,以期丰富东方蜜蜂微孢子虫SMC的基础信息,并为深入开展相关功能研究提供理论依据。
1 材料与方法 1.1 SMC的分子特性分析根据前期基于东方蜜蜂微孢子虫转录组数据[9]预测出的NCCS序列,利用NCBI网站( https://www.ncbi.nlm.nih.gov/)上的ORF工具预测相应的氨基酸序列。通过Expasy网站( https://www.espasy.org/resources)上的Protparam、ProtScale和SWISS-model等软件分析SMC的理化性质、亲水性和三级结构。使用SignalP 4.1 Server、NetPhos 3.1 Server、TMHMM及SOPMA等软件[10]预测SMC的信号肽、磷酸化位点、跨膜结构域和二级结构。采用PSORTⅡ软件( https://www.genscript.com/psort.html)[11]进行SMC的亚细胞定位预测。
1.2 SMC的保守基序和结构域预测使用MEME软件( https://meme-suite.org/)[12]预测东方蜜蜂微孢子虫、海伦脑炎微孢子虫Encephalitozoon hellem、菲尼斯毕罗酵母Piromycesc finnis、发廯菌Trichophyton tonsurans和黑曲霉Aspergillus melleus等10个物种SMC的保守基序。通过Pfam网站( http://pfam.xfam.org/search#tabview=tab1)查找东方蜜蜂微孢子虫等10个物种SMC保守结构域相关信息。采用TBtools软件预测上述10个物种SMC的结构域,选择默认参数。
1.3 SMC蛋白的系统进化分析利用Blast工具将东方蜜蜂微孢子虫SMC氨基酸序列比对到NCBI GenBank数据库( https://www.ncbi.nlm.nih.gov/genbank/),搜索序列相似性较高的其他物种的SMC。通过Mega 11.0软件[13]对东方蜜蜂微孢子虫和其他物种的SMC进行氨基酸序列多重比对,采用邻接法构建基于SMC的系统进化树,选择软件默认参数。
2 结果与分析 2.1 SMC的分子特性东方蜜蜂微孢子虫NCCS含有3 093个核苷酸,编码的SMC含有1 102个氨基酸。SMC的分子式为C5787H9418N1526O1771S41,相对分子质量约130 020,等电点为8.28;包含195个负电荷氨基酸,其中天冬氨酸和谷氨酸分别有67和128个;包含202个正电荷氨基酸,其中赖氨酸和精氨酸分别有169和33个;含量最高和最低的氨基酸分别为赖氨酸和色氨酸(表1)。
|
|
表 1 东方蜜蜂微孢子虫SMC的氨基酸组成 Table 1 Amino acid composition of SMC in Nosema ceranae |
SMC的脂溶系数为93.49,平均亲水系数为−0.740,亲水氨基酸数量比疏水氨基酸多(图1A),说明该蛋白为亲水性蛋白。SMC中不存在典型的信号肽,说明其为胞内蛋白(图1B)。另外,在SMC中预测到104个磷酸化位点,包含50个丝氨酸、26个酪氨酸和28个苏氨酸磷酸化位点(图1C)。
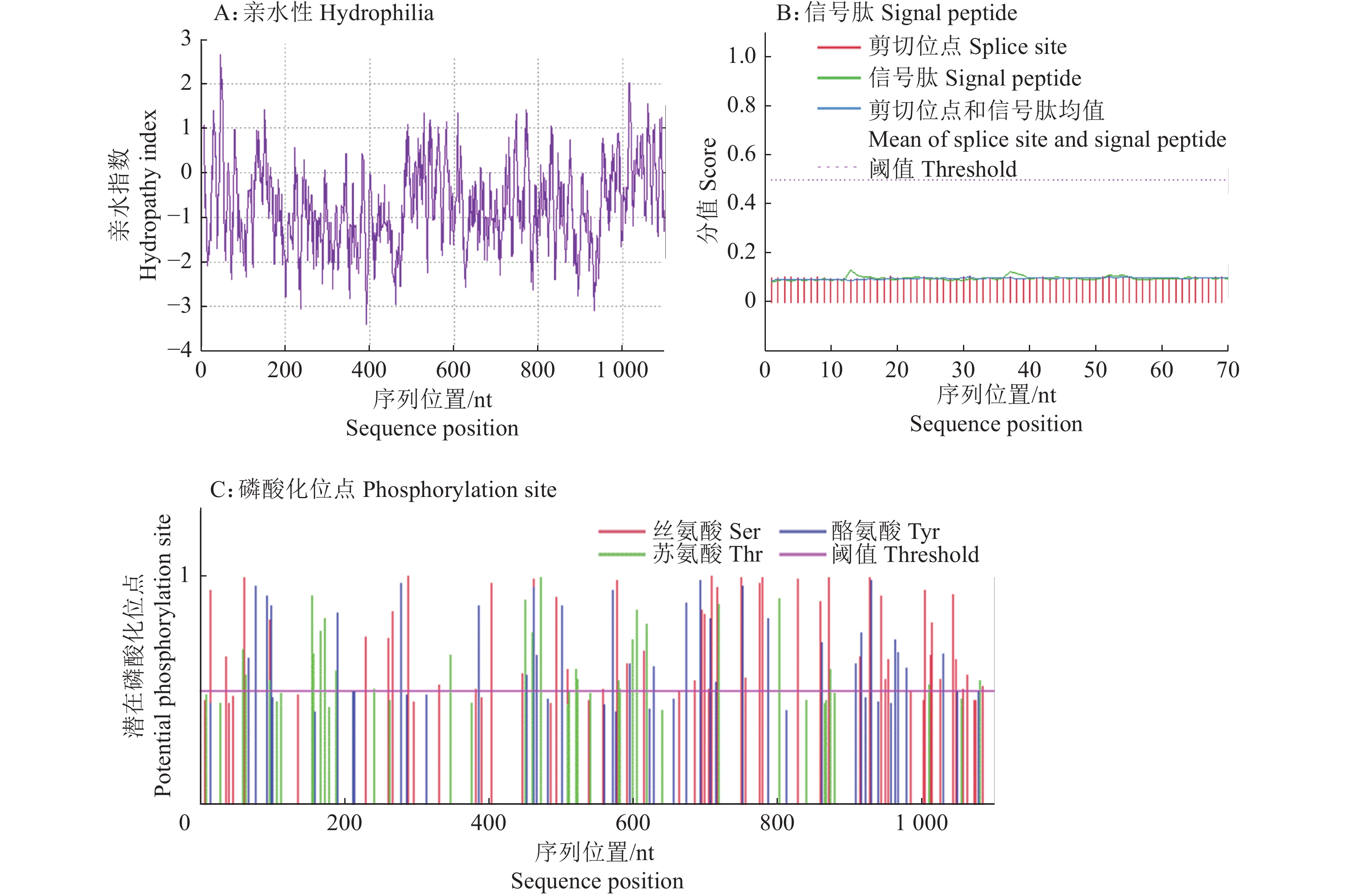
|
图 1 东方蜜蜂微孢子虫SMC的分子特性预测 Fig. 1 Molecular characteristic prediction of SMC in Nosema ceranae |
二级结构分析结果显示,SMC含有787个(71.42%)α−螺旋,106条(9.62%)β−折叠,49个(4.45%)β−转角和160个(14.52%)无规则卷曲(图2A)。三级结构分析结果显示,SMC的模板为6yvu.1.B,序列相似性为31.26%,其中61.00%的残基自信度达80%以上(图2B)。此外,SMC同时定位于细胞核、细胞质和线粒体,占比分别为78.30%、8.70%和13.00%。
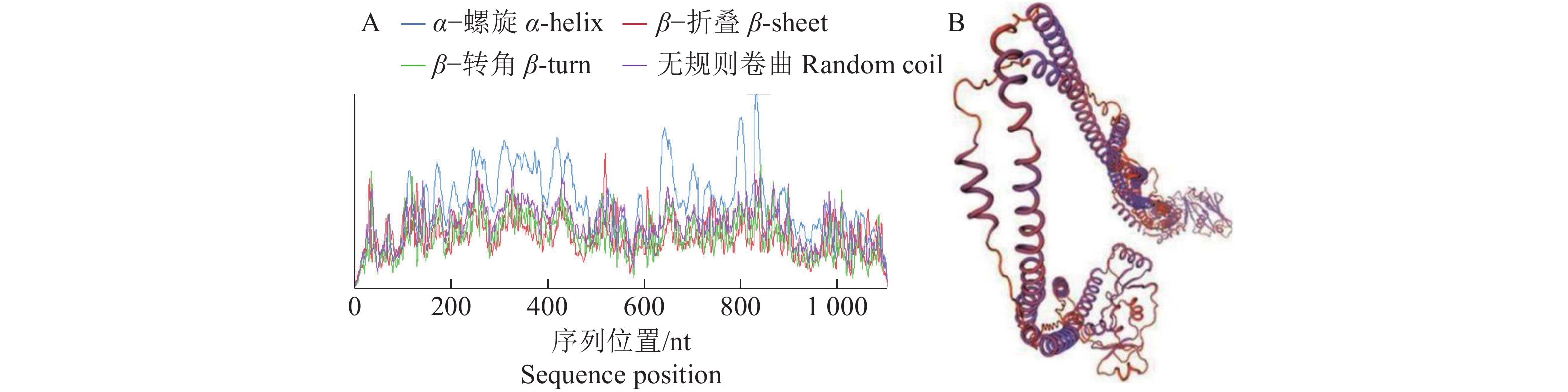
|
图 2 东方蜜蜂微孢子虫SMC的二级结构(A)和三级结构(B)预测 Fig. 2 Prediction of secondary structure (A) and tertiary structure (B) of Nosema ceranae SMC |
在东方蜜蜂微孢子虫SMC中预测到9个保守基序,分别为Motif 1、2、3、4、5、6、7、8、9;类似地,在海伦脑炎微孢子虫、颗粒病微孢子虫Nosema granulosis、麦格水蚤汉氏孢虫Hamiltosporidium magnivora、康氏泰罗汉孢虫Thelohania contejeani、菲尼斯毕罗酵母、指间毛廯菌Trichophyton interdigitale、紫色毛廯菌Trichophyton violaceum、发廯菌和黑曲霉的SMC中同样预测上述9个Motif(图3),说明SMC在东方蜜蜂微孢子虫和其他真菌物种中高度保守。

|
图 3 东方蜜蜂微孢子虫和其他9个物种SMC包含的保守基序 Fig. 3 Conserved motifs included in SMC of Nosema ceranae and other nine species |
在东方蜜蜂微孢子虫SMC中预测到4个结构域,包括1个SMC_N、1个SMC_hinge、1个AAA_29和1个AAA_23;在海伦脑炎微孢子虫、指间毛廯菌和黑曲霉SMC中同样预测到1个SMC_N、1个SMC_hinge、1个AAA_23和1个AAA_21;在颗粒病微孢子虫、麦格水蚤汉氏孢虫TBU02480.1和康氏泰罗汉孢虫SMC中预测到2个相同的结构域,包括1个SMC_N和1个SMC_hinge;在麦格水蚤汉氏孢虫TBU02444.1 SMC中预测到6个结构域,包括1个SMC_N、1个SMC_hinge、1个AAA_29、1个AAA_21、1个AAA_23和1个AAA_15;在菲尼斯毕罗酵母SMC中预测到5个结构域,包括1个SMC_N、1个SMC_hinge、1个AAA_29、1个AAA_21和1个AAA_23;在紫色毛廯菌SMC中预测到3个结构域,包括1个SMC_N、1个SMC_hinge和1个AAA_15;在发廯菌SMC中预测到3个结构域,包括1个AAA_21、1个SMC_N和1个SMC_hinge(图4)。进一步分析发现东方蜜蜂微孢子虫和其他9个物种的SMC中均含有1个SMC_N和1个SMC_hinge(图4)。以上结果进一步说明SMC在东方蜜蜂微孢子虫和其他真菌物种中高度保守。

|
图 4 东方蜜蜂微孢子虫和其他9个物种SMC的结构域比较 Fig. 4 Comparison of structural domains within SMC in Nosema ceranae and other nine species |
如表2所示,东方蜜蜂微孢子虫、海伦脑炎微孢子虫、颗粒病微孢子虫、康氏泰罗汉孢虫、菲尼斯毕罗酵母、指间毛廯菌、紫色毛廯菌、发廯菌和黑曲霉均仅含有1个SMC,而麦格水蚤汉氏孢虫含有2个SMC。
|
|
表 2 东方蜜蜂微孢子虫与其他9个物种的SMC蛋白概览 Table 2 Overview of SMC proteins in Nosema ceranae and other nine species |
氨基酸序列多重比对结果显示,东方蜜蜂微孢子虫与菲尼斯毕罗酵母的SMC序列相似性最高,达到61.96%,其次是与指间毛廯菌、发廯菌、紫色毛廯菌和黑曲霉,SMC序列相似性均为60.98%,与康氏泰罗汉孢虫的SMC序列相似性最低(34.73%)。
系统进化分析结果显示,东方蜜蜂微孢子虫、颗粒病微孢子虫、麦格水蚤汉氏孢虫、康氏泰罗汉孢虫和海伦脑炎微孢子虫的SMC聚为一个大支,而发廯菌、指间毛廯菌、紫色毛廯菌、黑曲霉和菲尼斯毕罗酵母的SMC聚为一个大支;东方蜜蜂微孢子虫与颗粒病微孢子虫的SMC聚为一支,且置信度达到99%,说明二者SMC的进化距离最近(图5)。

|
图 5 邻接法构建基于SMC的东方蜜蜂微孢子虫与其他9个物种的系统进化树 Fig. 5 Phelogenetic tree of Nosema ceranae and other nine species based on SMC by neighbor-joining method |
目前,由于缺乏成熟的转基因操作技术体系,东方蜜蜂微孢子虫绝大多数基因功能未明,相关信息匮乏。本研究通过生物信息学手段对东方蜜蜂微孢子虫NCCS基因编码的SMC进行分子特性解析,结果显示,SMC的分子式为C5787H9418N1526O1771S41,包含1 102个氨基酸,相对分子质量约130 020,等电点为8.28,脂溶系数为93.49,平均亲水系数为−0.740,亲水氨基酸数量多于疏水氨基酸,说明SMC可能是亲水性蛋白;不含信号肽和跨膜螺旋区,说明SMC可能为胞内蛋白和非跨膜蛋白。以上结果丰富了东方蜜蜂微孢子虫NCCS基因的基本信息,为进一步开展相关功能研究提供了有价值的参考信息。另外,预测SMC同时定位于细胞核、细胞质和线粒体,但主要定位于细胞核(占比78.30%),鉴于染色体主要存在于细胞核,上述结果符合客观实际;但预测到少量SMC分布于细胞质和线粒体,一方面需要通过分子生物学试验加以验证,另一方面暗示SMC功能的潜在多样性。
真核生物中存在6种SMC,这6种蛋白两两结合形成异二聚体,进而结合其他组分形成复合体,分别为黏结蛋白复合体、凝聚蛋白复合体和SMC5-SMC6复合体,这些复合体在DNA修复、重组与复制等方面发挥重要作用[14]。SMC在进化上较为保守,从微生物到哺乳动物的SMC蛋白都具有相似的结构[15]。本研究在东方蜜蜂微孢子虫、海伦脑炎微孢子虫、麦格水蚤汉氏孢虫、颗粒病微孢子虫、康氏泰罗汉孢虫、菲尼斯毕罗酵母、指间毛廯菌、紫色毛廯菌、发廯菌和黑曲霉的SMC中均预测到Motif 1~9共9个保守基序;此外,发现东方蜜蜂微孢子虫和上述其他物种的SMC均含有1个SMC_N和1个SMC_hinge。以上结果表明SMC在东方蜜蜂微孢子虫和其他真菌中具有高度保守性,推测SMC在东方蜜蜂微孢子虫和上述其他真菌中发挥类似功能。本研究发现,东方蜜蜂微孢子虫、颗粒病微孢子虫、海伦脑炎微孢子虫、麦格水蚤汉氏孢虫和康氏泰罗汉孢虫的SMC聚为一个大支,说明这些物种的SMC亲缘关系较近;东方蜜蜂微孢子虫与颗粒病微孢子虫的SMC聚为一支,置信度为99 %,说明二者的SMC进化距离最近。
前人研究发现SMC的C端和N端相互结合形成ATP酶功能域,因此SMC蛋白也属于ABC(ATP binding cassette)蛋白家族[16],ABC蛋白在东方蜜蜂微孢子虫的生命活动中起到重要作用[17]。因此,推测SMC在东方蜜蜂微孢子虫中发挥的功能类似于ABC蛋白。
通过建立东方蜜蜂微孢子虫侵染成年蜜蜂的模式进行东方蜜蜂微孢子虫的基因功能研究已见诸报道[18-19]。我们下一步拟通过体外转录合成NCCS的dsRNA,并通过饲喂法探究东方蜜蜂微孢子虫侵染蜜蜂宿主过程中NCCS的功能。
| [1] |
梁勤, 陈大福. 蜜蜂保护学[M]. 北京: 中国农业出版社, 2009: 5-7.
(  0) 0) |
| [2] |
CORNMAN R S, CHEN Y P, SCHATZ M C, et al. Genomic analyses of the microsporidian Nosema ceranae, an emergent pathogen of honey bees
[J]. PLoS Pathogens, 2009, 5(6): e1000466. DOI:10.1371/journal.ppat.1000466 (  0) 0) |
| [3] |
MARÍN-GARCÍA P J, PEYRE Y, AHUIR-BARAJA A E, et al. The role of Nosema ceranae (microsporidia: Nosematidae) in honey bee colony losses and current insights on treatment
[J]. Veterinary Sciences, 2022, 9(3): 130. DOI:10.3390/vetsci9030130 (  0) 0) |
| [4] |
陈华枝, 杜宇, 范小雪, 等. 基于第三代纳米孔测序技术的东方蜜蜂微孢子虫全长转录组构建及注释[J]. 昆虫学报, 2020, 63(12): 1461-1472. DOI:10.16380/j.kcxb.2020.12.004 (  0) 0) |
| [5] |
YOSHINAGA M, INAGAKI Y. Ubiquity and origins of structural maintenance of chromosomes (SMC) proteins in eukaryotes[J]. Genome Biology and Evolution, 2021, 13(12): evab256. DOI:10.1093/gbe/evab256 (  0) 0) |
| [6] |
张建祥. 水稻TOP6和SMC1/3蛋白以及miRNA在体细胞DSB修复中的功能研究[D]. 扬州: 扬州大学, 2021.
(  0) 0) |
| [7] |
KIM K D. Potential roles of condensin in genome organization and beyond in fission yeast[J]. Journal of Microbiology, 2021, 59(5): 449-459. DOI:10.1007/s12275-021-1039-2 (  0) 0) |
| [8] |
ONO T, LOSADA A, HIRANO M, et al. Differential contributions of condensin I and condensin II to mitotic chromosome architecture in vertebrate cells[J]. Cell, 2003, 115(1): 109-121. DOI:10.1016/S0092-8674(03)00724-4 (  0) 0) |
| [9] |
GUO R, CHEN D F, XIONG C L, et al. First identification of long non-coding RNAs in fungal parasite Nosema ceranae
[J]. Apidologie, 2018, 49(5): 660-670. DOI:10.1007/s13592-018-0593-z (  0) 0) |
| [10] |
ISON J, KALAS M, JONASSEN I, et al. EDAM: An ontology of bioinformatics operations, types of data and identifiers, topics and formats[J]. Bioinformatics, 2013, 29(10): 1325-1332. DOI:10.1093/bioinformatics/btt113 (  0) 0) |
| [11] |
SEDAGHAT-NEJAD E, FAKHARIAN M A, PI J, et al. P-sort: An open-source software for cerebellar neurophysiology[J]. Journal of Neurophysiology, 2021, 126(4): 1055-1075. DOI:10.1152/jn.00172.2021 (  0) 0) |
| [12] |
BAILEY T L, ELKAN C. Fitting a mixture model by expectation maximization to discover motifs in biopolymers[J]. Proceedings: International Conference on Intelligent Systems for Molecular Biology, 1994, 2: 28-36. (  0) 0) |
| [13] |
TAMURA K, STECHER G, KUMAR S. MEGA11: Molecular evolutionary genetics analysis version 11[J]. Molecular Biology and Evolution, 2021, 38(7): 3022-3027. DOI:10.1093/molbev/msab120 (  0) 0) |
| [14] |
HARVEY S H, KRIEN M J E, O'CONNELL M J. Structural maintenance of chromosomes (SMC) proteins, a family of conserved ATPases[J]. Genome Biology, 2002, 3(2): 3003.1. (  0) 0) |
| [15] |
STRUNNIKOV A V, JESSBERGER R. Structural maintenance of chromosomes (SMC) proteins: Conserved molecular properties for multiple biological functions[J]. European Journal of Biochemistry, 1999, 263(1): 6-13. DOI:10.1046/j.1432-1327.1999.00509.x (  0) 0) |
| [16] |
HIRANO T. The ABCs of SMC proteins: Two-armed ATPases for chromosome condensation, cohesion, and repair[J]. Genes & Development, 2002, 16(4): 399-414. (  0) 0) |
| [17] |
HUANG Q, LI W, CHEN Y, et al. Dicer regulates Nosema ceranae proliferation in honeybees
[J]. Insect Molecular Biology, 2019, 28(1): 74-85. DOI:10.1111/imb.12534 (  0) 0) |
| [18] |
王思懿, 胡颖, 刘小玉, 等. 东方蜜蜂微孢子虫LCFAL 2基因的分子特性及系统进化分析[J/OL]. 环境昆虫学报, 2023. https://kns.cnki.net/kcms/detail//44.1640.Q.20230215.1814.010.html
(  0) 0) |
| [19] |
FAN Y C, WANG J, YU K J, et al. Comparative transcriptome investigation of Nosema ceranae infecting eastern honey bee workers
[J]. Insects, 2022, 13(3): 241. DOI:10.3390/insects13030241 (  0) 0) |
 2023, Vol. 44
2023, Vol. 44


