水稻Oryza sativa是世界上重要的粮食作物,是全球主要的食物来源之一,其产量的高低直接影响粮食安全。每年由细菌、真菌和病毒侵染造成的水稻减产高达30%~50%。其中稻瘟病对水稻生产造成的危害最为严重[1],培育具有稻瘟病抗性的水稻品种是防治该病害最经济有效的方法[2]。然而,由于稻瘟病生理小种的迅速更替,许多抗病品种的抗病性在逐渐丧失[3]。目前已经被鉴定并克隆的稻瘟病抗性(R)基因有35个[4],其中Pi2基因来自对稻瘟病具有高度抗性的哥伦比亚籼稻品种5173[5],是一个广谱抗稻瘟病显性基因。该基因对13个国家的36个生理小种表现出高水平抗性[6]。针对我国华南籼稻区的稻瘟病优势生理小种,Pi2基因表现出良好的抗性[7- 8],利用Pi2基因选育具有广谱抗性的水稻品种具有重要的应用价值。
随着生物技术的发展,利用分子标记进行分子标记辅助选择(Maker-associated selection, MAS)可以大大提高育种的效率和进程[9]。目前针对Pi2基因开发并应用于育种的分子标记大多数属于第2代分子标记,包括简单重复序列标记(Simple sequence repeats,SSR)、插入/缺失标记(Insert/Deletion,InDel)等,这类标记需要电泳技术进行检测,其操作过程较为繁琐,主要应用于基因定位和遗传图谱的绘制[10],无法实现大批量的准确检测,严重妨碍了该基因在水稻育种中的应用。
第3代分子标记——单核苷酸多态性(Single nucleotide polymorphism,SNP)标记[11-13]的出现为快速检测相关基因开辟了一条新的途径。SNP是指分子水平上由单个核苷酸的改变即单个碱基的替换、插入或缺失,从而引起的DNA序列的多态性,被认为是水稻基因组中最丰富的标记类型[14]。SNP在生物基因组中数量多,分布广泛,遗传稳定性高,与性状表达密切相关,富有代表性。另外,SNP标记检测技术易于实现自动化高通量基因分型,可以有效缩短检测时间[15]。
近年来,竞争性等位基因聚合酶链式反应(Kompetitive allele specific PCR,KASP)技术已成为高效安全地检测SNP的新方法。KASP技术是一种基于SNP标记的高通量基因分型技术,通过识别相应的荧光共振能量转移,利用基因探针对广泛的基因组DNA样品,以及对目标SNPs和InDels进行精准的双等位基因分型[16]。该技术的检测过程无需电泳,可以有效地减少试验过程中有害物质的产生,现已广泛应用于各类作物的种质筛选工作[17-18]。针对Pi2基因,利用KASP技术开发的分子标记相较于普遍使用SSR标记具有稳定性好、检测效率高等优点,可以更加高效准确地鉴定抗病基因,为聚合多个优质基因、培育高产抗病水稻品种提供技术支持。
本研究通过对大量水稻种质资源的基因组测序数据分析,挖掘Pi2基因核心区域SNP并开发为KASP分子标记,进一步通过室内接种、病圃鉴定验证标记的可靠性,最终构建一套适合于Pi2基因的分型体系。研究结果将为利用稻瘟病抗性基因Pi2进行品种选育提供技术支撑。
1 材料与方法 1.1 供试材料本研究以华南农业大学国家植物航天育种工程技术研究中心收集的593份水稻自然种质作为供试材料,该群体分为122个粳稻材料和471个籼稻材料。通过该群体的基因组测序信息筛选SNP位点,并利用H-74和H-78开发KASP标记。以该群体田间2021年早造和晚造材料以及阳江市2020年晚造病圃材料作为验证材料。以R1179作为抗病对照,‘日本晴’作为感病对照。材料采用水培法[19]种植,营养液参照国际水稻研究所(International Rice Research Institute,IRRI)的培养配方配制。
1.2 试验方法 1.2.1 DNA提取方法本研究采用改良的十六烷基三甲基溴化铵(Cetyltrimethylammonium bromide,CTAB)法提取DNA。具体流程如下:取少量幼嫩的叶片,放入2 mL离心管中,加入2×CTAB提取液800 mL,用高通量组织研磨仪(Geno/Grinder)以1 500 r/min充分研磨3 min,65 ℃水浴30 min;冷却后加入三氯甲烷︰异戊醇体积比为24︰1的混合液至满管(约900 μL),混合均匀,8 000 r/min离心10 min,放入–20 ℃的冰箱中20 min,使DNA充分沉淀;10 000 r/min离心3 min,去除上清液,倒置1 min,加入φ为70%的乙醇溶液800 μL,10 000 r/min离心3 min,去除上清液,干燥后加入ddH2O100 μL,使DNA充分溶解,置于–20 ℃保存。
1.2.2 KASP标记开发在美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)下载Pi2序列(Genbank登录号:DQ352453)以及与Pi2互为等位基因的Pi9(Genbank登录号:DQ285630.1)和Pigm序列(Genbank登录号:KU904633.2)。分别利用程序VISTA tools( https://genome.lbl.gov/vista/index.shtml)与Softberry( http://www.softberry.com/berry.phtml)在线分析,获得序列组成和基因结构信息。对H-74和H-78材料进行单倍型分析,获取Pi2抗性位点核心区域和重要SNP差异位点,并根据样品材料的相似性判断目标基因是否存在。结合单倍型分析结果获得的Pi2基因抗性位点核心区域的物理区间,利用程序Primer-BLAST( https://www.ncbi.nlm.nih.gov/tools)和软件Premier5设计普通PCR引物。H-74和H-78这2个材料各设置2个重复,对目标物理位置进行扩增并测序。对上述获得的测序结果进行比对,分析差异SNP,优先选择单碱基突变、SNP突变位点位于编码区且周边没有其他的差异SNP位点,利用这些位点基于KASP技术设计成若干KASP分子标记。
1.2.3 KASP基因分型将待检测的DNA样品稀释至50 mg/L,加入96孔PCR板中。配制KASP扩增反应体系20 μL(DNA 5 μL、引物Mix 0.4 μL、2×KASP Master Mix 4.6 μL,矿物油10 μL)。KASP扩增反应程序:95 ℃预变性180 s,循环1次;95 ℃变性10 s,63.4 ~ 56.2 ℃退火/延伸30 s(–0.8 ℃/循环),循环9次;95 ℃变性10 s,55 ℃退火/延伸30 s,循环29次。待PCR扩增程序结束后,用荧光检测仪(FLUOstar Omega SNP)进行荧光检测,获得基因分型结果。
1.2.4 稻瘟病菌接种鉴定使用开发的KASP标记进行基因分型后,选择检测结果为阳性和阴性的材料各3份,以及已知具有Pi2基因的材料R1179作为阳性对照和无Pi2基因的材料‘日本晴’作为阴性对照。将种子浸种催芽处理,待种子浸种露白后,以8×8的规格种植于30 cm×20 cm的盆中,每个品种种植4株,重复2次,待水稻生长至3~4叶期,参照韩美丽等[20]的方法离体单菌接种稻瘟病菌GDYJ7,6~7 d后调查。
自然诱发病圃设置在广东省阳江市阳江农业科学研究所白沙试验场,该病圃常年处于高温高湿的气候环境中,具有发病的地理环境条件,是广东省具有代表性的稻瘟病区。试验材料按单本种植,每份材料种5行,每行6株,行距23.3 cm,株距16.7 cm,四周种植诱发品种‘Co39’。病圃全生育期防虫不防病。在成熟期,以‘Co39’充分发病为准,对供试材料进行穗颈瘟调查,按IRRI 0~9级的分级标准进行记录(0级:无病;1级:抗;3级:中抗;5级:中感;7级:感;9级:高感)。
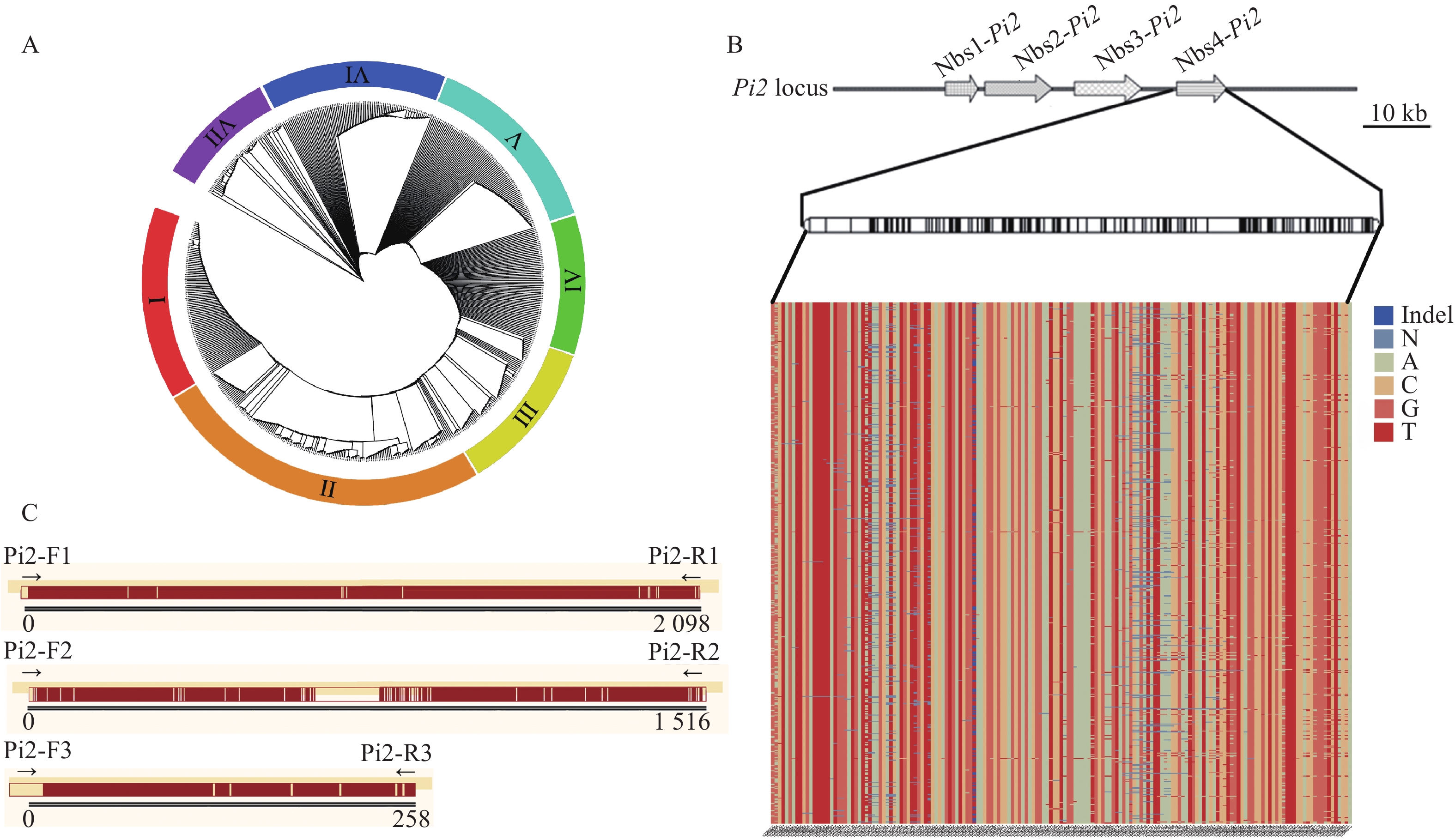
2 结果与分析 2.1 Pi2基因核心SNP筛选基于593份自然群体的全基因组重测序结果,结合Pi2基因序列,利用MEGA X64构建系统进化树,根据进化树分布密度,分为7个部分(图1A)。从中选择2个抗性不同且亲缘关系较远的材料H-74和H-78,其中H-74为抗病材料,与R1179等抗病材料在III区;H-78为感病材料,与‘日本晴’、‘丽江新团黑谷’等感病材料在II区。Pi2基因第2外显子上第787~788位密码子位置的核苷酸变异与稻瘟病抗性密切相关[21-24]。因此对H-74和H-78这2个材料,在Pi2基因区段以及基因上下游100 kb区域进行单倍型分析,确定Pi2基因抗性位点核心区域的物理位置在10386510~10389466 bp的范围内。单倍型分析结果中发现该区域内含有99个重要的SNP突变位点。根据材料测序结果与分析结果中位点突变情况的相似性判断H-74材料含有Pi2基因,H-78材料不含有Pi2基因。进一步利用593份自然群体的全基因组重测序结果,分析区域范围内的所有差异SNP位点,共筛选出175个SNP位点,并获得这些位点的碱基分布情况(图1B)。
|
图 1 系统进化树及差异SNP位点分析 Fig. 1 Phylogenetic tree and analysis of different SNP sites A:593份材料系统进化树(Ⅰ~Ⅶ:分布密度不同的7个部分);B:593份材料在Pi2抗性位点核心区域的差异SNP及其碱基分布情况;C:H-74和H-78材料在Pi2抗性位点核心区域差异SNP位点分布(黄色竖线区域表示两者差异SNP位点分布情况) A: Phylogenetic tree of 593 materials (Ⅰ−Ⅶ: Seven parts with different distribution density); B: Distributions of different SNPs and their bases in the core region of the Pi2 resistance locus for the 593 materials; C: Distribution of different SNP sites in the core region of the Pi2 resistance locus for H-74 and H-78 materials (The yellow vertical line area indicates the distribution of SNP sites that are different between them) |
根据物理位置10386510~10389466 bp,利用软件Primer5设计引物(表1),分别利用3对引物对H-74和H-78材料的目标片段进行扩增和测序,测序结果进行序列比对分析,以获得更加精确的SNP位点及其分布和碱基突变情况(图1C,图2)。结合593份材料中发现的175个SNP位点与单倍型分析获得的99个SNP位点,过滤数据,在10386510~10389466 bp的区间内共筛选出28个关键SNP位点,其中9个位于内含子,19个位于外显子。
|
|
表 1 测序引物 Table 1 Sequencing primers |

|
图 2 Pi2基因差异位点序列比对 Fig. 2 Sequence alignment analysis of the different sites of Pi2 gene a:H-74材料,b:包含Pi2基因的材料,c:H-78材料,d、e:确定没有Pi2基因的材料;1和2表示材料的2个重复 a: Material H-74, b: Material containing Pi2 gene, c: Material H-78, d and e: Material without Pi2 gene; 1 and 2 represent two repetitions of the material |
以不含有Pi2基因的‘日本晴’基因组作为参考序列,将H-74材料与‘日本晴’基因组序列进行比对,对上述的28个关键SNP位点进一步筛选,剔除相同的突变SNP位点。最后获得5个关键的SNP位点,分别在10388566、10388841、10389007、10389256和10389341 bp发生SNP变异,利用这5个SNP位点设计KASP引物(表2)。
|
|
表 2 KASP引物 Table 2 KASP primers |
以含有Pi2基因的水稻材料R1179为阳性对照,不含Pi2基因的‘日本晴’为阴性对照,ddH2O为空白对照,在同一批593份自然群体材料中随机选取84份,利用上述引物对其进行KASP基因分型检测。待测样品扩增产物的荧光信号显示为蓝色圆点,且靠近X轴,则表明该材料包含Pi2基因,即T/T基因型;如果待测的扩增产物荧光信号显示为红色圆点,且靠近Y轴,则表明该材料不包含Pi2基因,即A/A基因型。
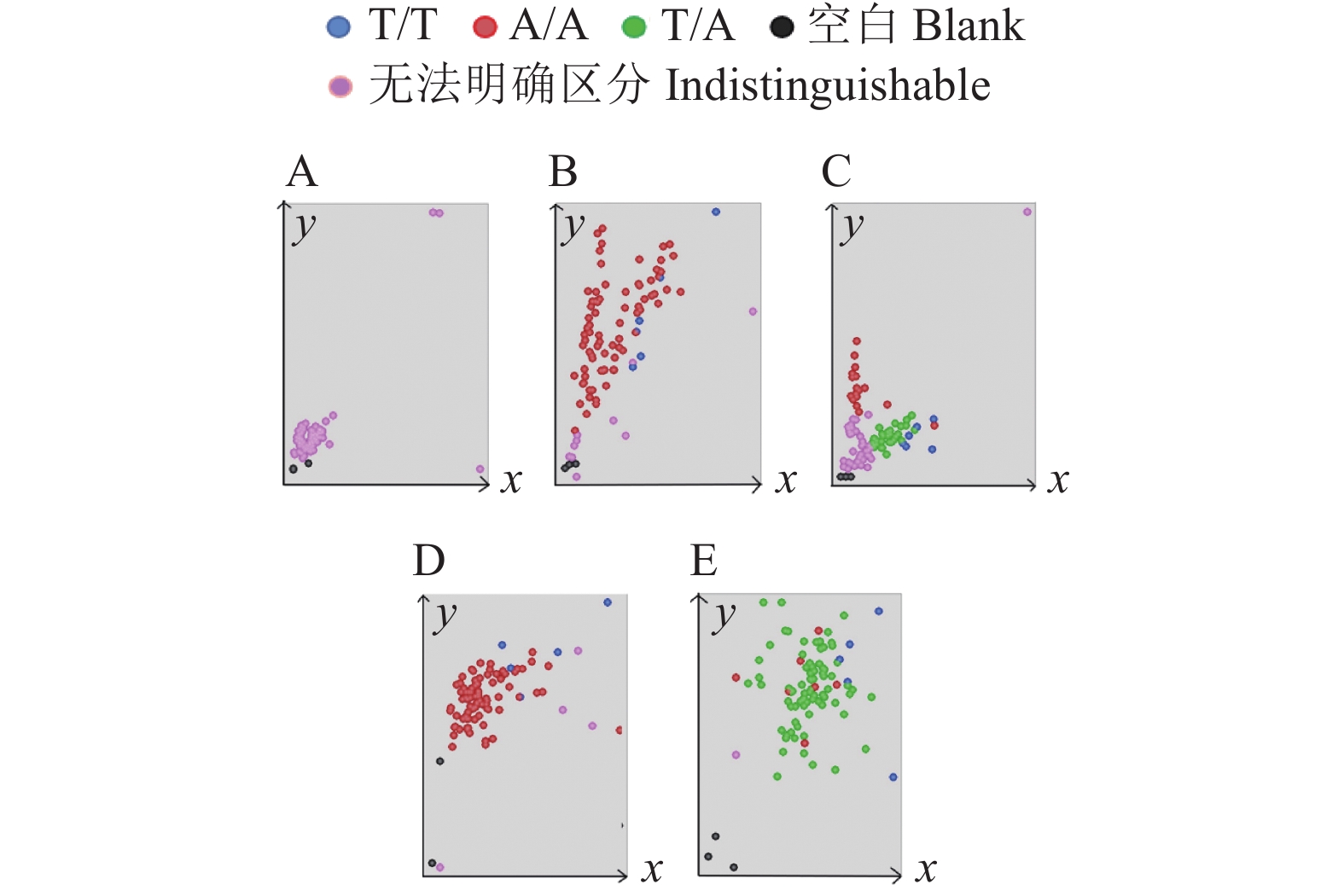
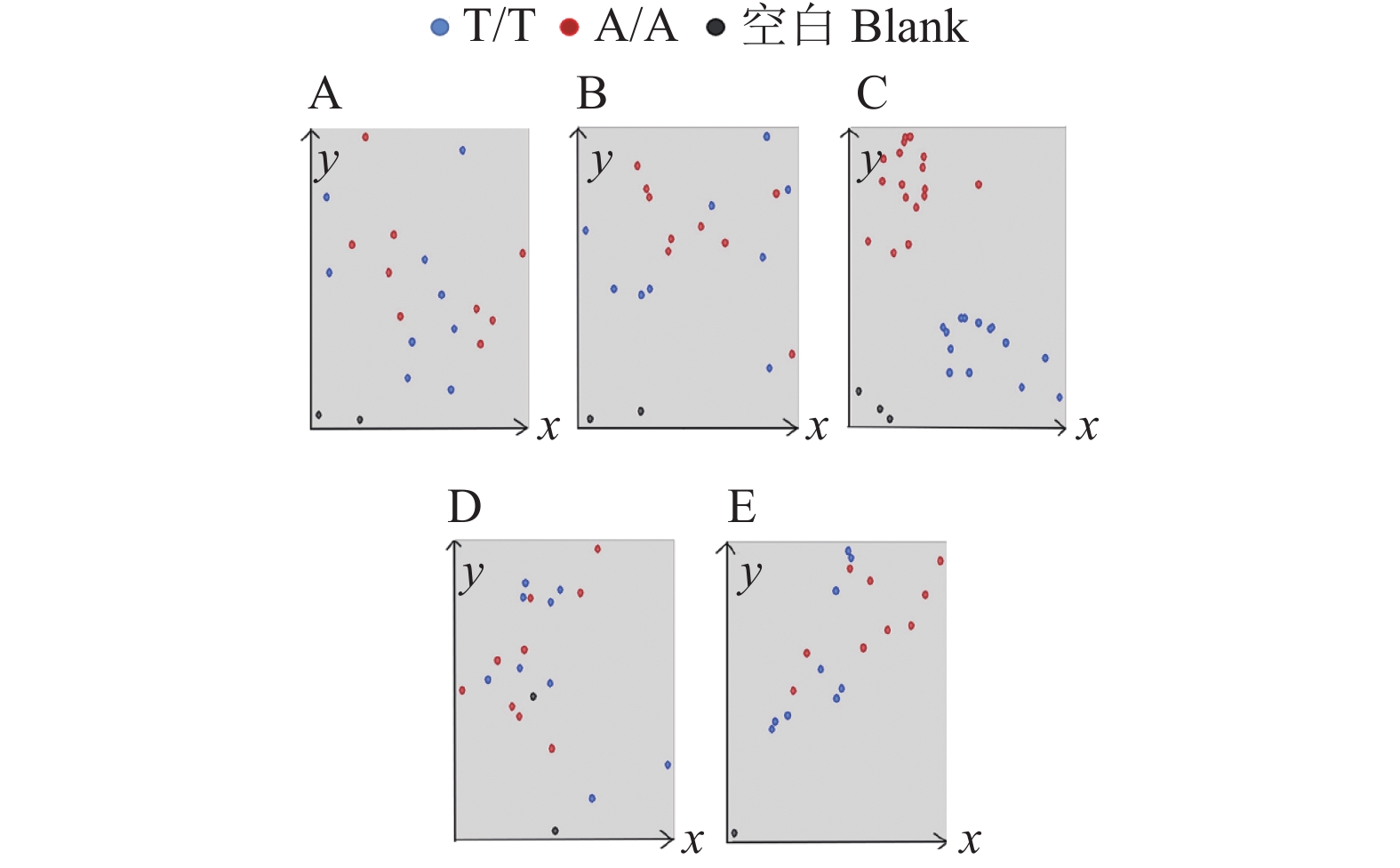
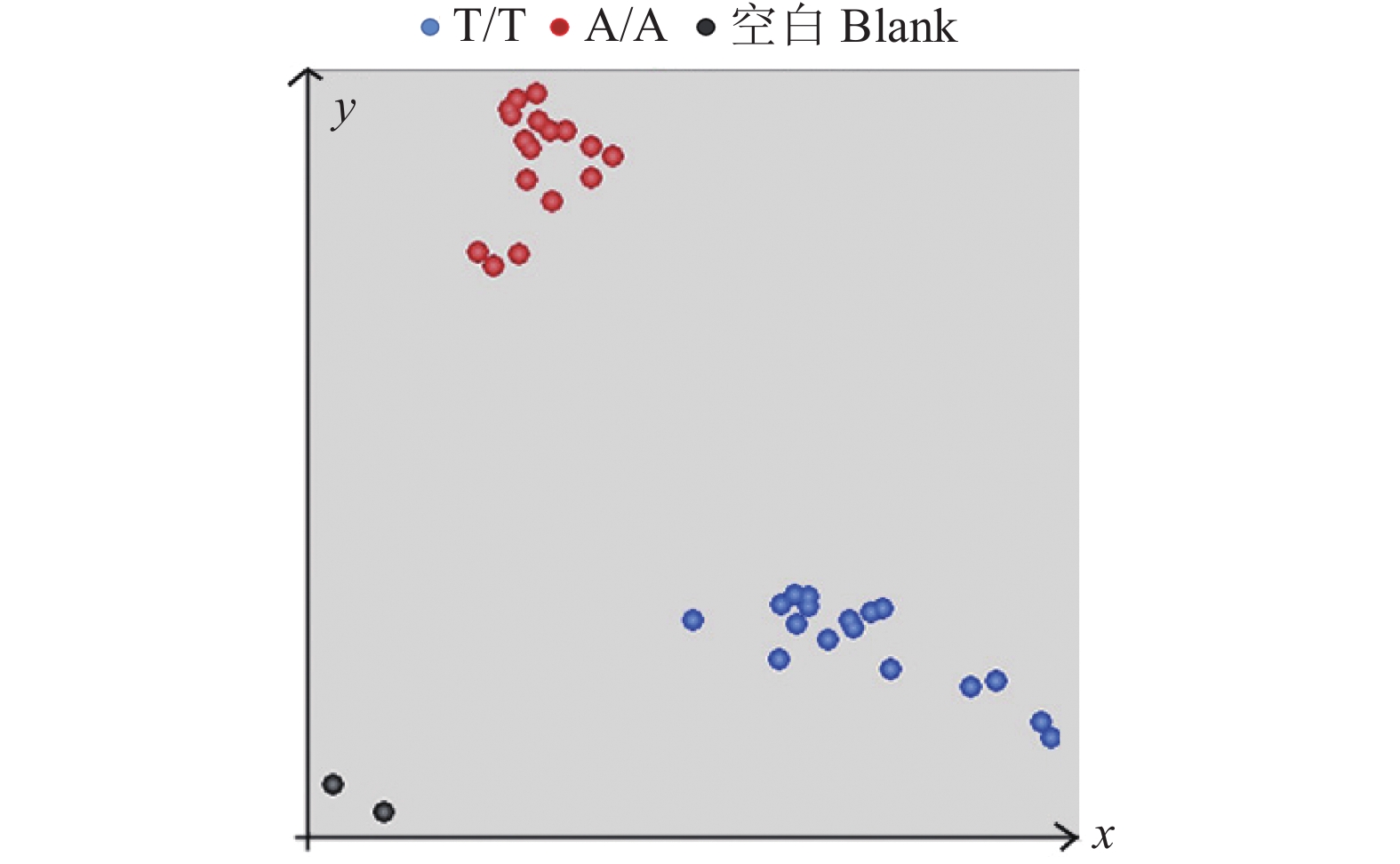
如图3所示,引物Pi2-C1、Pi2-C2和Pi2-C3的基因分型中有着分群聚集的趋势,引物Pi2-C4和Pi2-C5分型结果不够明确(图3)。为进一步确认这5个引物的可行性,利用3个已知包含Pi2基因的样品材料和3个已知不包含Pi2基因的样品材料作为阴、阳对照,同样以ddH2O作为空白对照,对其进行KASP基因分型检测,结果如图4所示。引物Pi2-C3可以成功将阴、阳对照及空白对照和检测样品材料分开。基因分型结果和预期结果相匹配,初步判断引物Pi2-C3的可行性。然而利用引物Pi2-C3的分型结果较为分散,为获得较为直观和清晰的图片,在原有的检测PCR程序基础上加4个循环,获得新的KASP基因分型结果(图5)。
|
图 3 KASP引物基因分型效果 Fig. 3 Genotyping effects of KASP primers A~E对应的引物分别为Pi2-C1、Pi2-C2、Pi2-C3、Pi2-C4、Pi2-C5 Primers corresponding to A−E are Pi2-C1, Pi2-C2, Pi2-C3, Pi2-C4 and Pi2-C5 respectively |
|
图 4 对照KASP基因分型结果 Fig. 4 KASP genotyping results for control samples A~E对应的引物分别为Pi2-C1、Pi2-C2、Pi2-C3、Pi2-C4、Pi2-C5 Primers corresponding to A−E are Pi2-C1, Pi2-C2, Pi2-C3, Pi2-C4 and Pi2-C5 respectively |
|
图 5 利用引物Pi2-C3增加PCR循环数后KASP基因分型结果 Fig. 5 KASP genotyping results after increasing PCR cycles by primer Pi2-C3 |
为评估KASP标记Pi2-C3的检测效果,本研究基于全基因组重测序结果选取了6个基因型为T/T的材料作为阳性对照、4个基因型为A/A的材料作为阴性对照,ddH2O作为空白对照,对84个田间水稻材料,分别利用设计的KASP分子标记Pi2-C3和已报道的SSR标记M-Pi2(Pi2-F:5′-CAGCGATGGTATGAGCACAA-3′,Pi2-R:5′-CGTTCCTATACTGCCACATCG-3′)进行检测,并对两者的检测结果进行比较。SSR标记扩增后,含有Pi2基因的片段大小为450 bp,不含Pi2基因的片段大小为282 bp[25]。结果如图6A 所示,KASP标记的基因分型结果中,有50个材料的荧光信号点为蓝色,38个材料的荧光信号点为红色,4个材料的荧光信号点为绿色,前后进行2次检测,分型结果一致,表明KASP标记Pi2-C3具有较好的重复性和稳定性。将上述结果与SSR标记的电泳结果(图6B)相比较,两者有95%的相似性,并且在两者的结果中,10个对照材料的检测结果与前期测序结果一致。因此可初步判断,KASP标记Pi2-C3可以成功区分纯合型T/T、A/A和杂合型T/A这3种基因型。
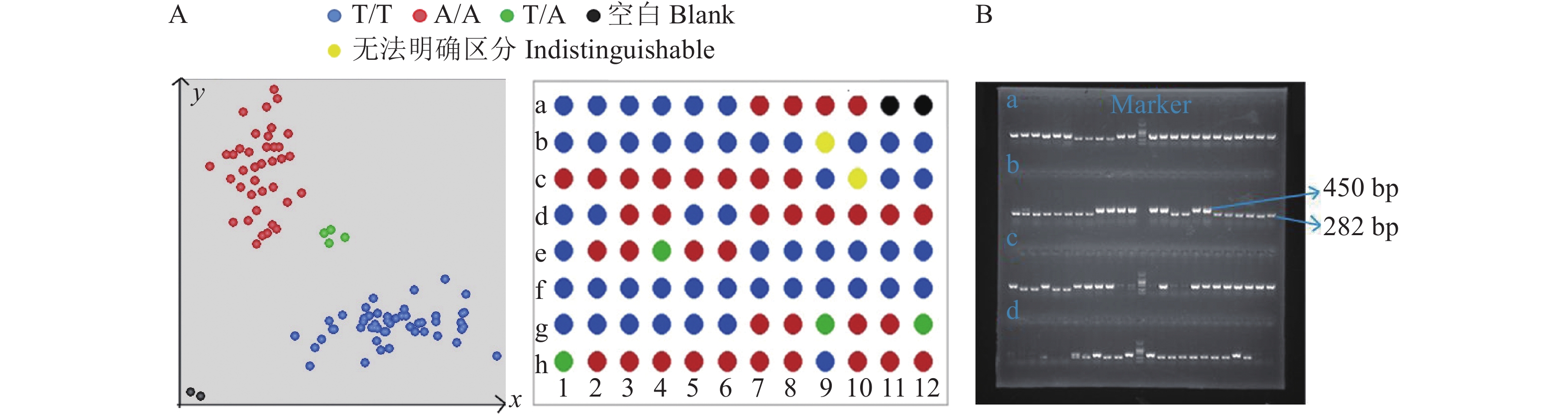
|
图 6 84个田间材料Pi2-C3 KASP分子标记的验证 Fig. 6 Verification of Pi2-C3 KASP molecular marker for 84 field samples A:Pi2 KASP基因分型检测结果;B:SSR标记检测结果(Marker位于胶图中央,大小为2 000 bp,a、c、d区域中相应位置片段大小与b区域标示相同) A: KASP genotype detection results for Pi2; B: SSR marker detection results (marker is located in the center of the rubber map, with the size of 2 000 bp, and the corresponding fragment sizes in areas a, c and d are the same as that in area b) |
从593份材料中对该位点进行筛选,挑选出基因型为T/T的2份材料607-557和RT1031-69;基因型为A/T的22份材料,包括京农大‘W030’、‘早粳占’、CT15672-3-2-3-2-2-M、‘华粳籼74’、白水糯、雪华新占、海南糯、1641选、远野4号、‘黄广华占1号’、黑糯47、Getu、海南黑、本地糯(阳春三甲)、黑节早、广粳籼165、观音稻、IRRI129、黄广软占、‘大丰糯’、‘鹏稻2号’、‘凤新丝苗-1’;其余材料基因型为A/A。从基因型为A/A的材料中随机挑选出60个材料,再加上挑选出的基因型为T/T和A/A的24份材料,对这84个材料进行KASP基因分型检测,进一步验证KASP标记的可靠性。如图7A所示,a-1和a-2对应基因型为T/T的2个材料,与检测结果保持一致。a-3至b-12对应基因型为A/T的22个材料,基因型数量比T/T︰A/A=13︰6,初步判断这22个材料后代发生性状分离。c-1至g-12对应基因型为A/A的60个材料,54个检测为A/A,6个检测为T/T,准确率为93%(图7B)。
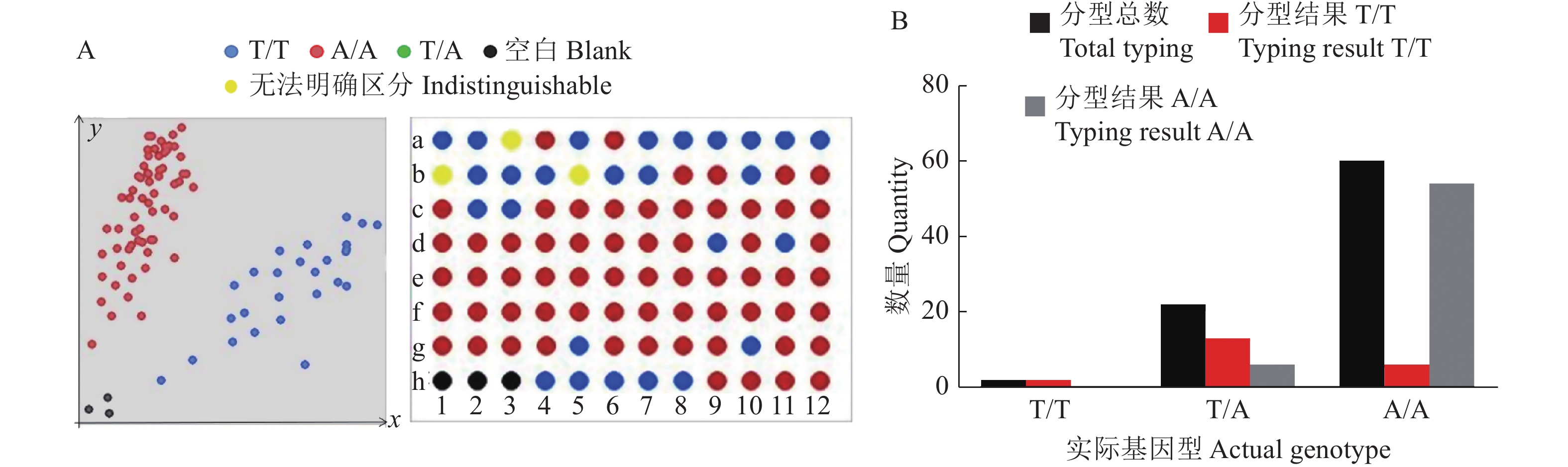
|
图 7 593份材料中部分材料KASP基因分型结果 Fig. 7 KASP genotyping results of partial materials of the 593 materials A:Pi2基因KASP基因分型结果;B:Pi2基因KASP基因分型与实际基因型比对分析 A: KASP genotyping results for Pi2 gene; B: Comparison analysis between Pi2 gene KASP genotyping and actual genotype |
为进一步验证基因分型结果的准确性,以R1179与‘日本晴’分别作为阳性对照与阴性对照,对基因分型中确定含有Pi2基因的种质(H-11、H-12和H-94)与基因分型中确定不含有Pi2基因的种质(H-14、H-15和H-151)进行稻瘟病离体打孔接种试验,5~7 d后调查接种表型,根据叶片病斑大小分析材料的抗病性。结果(图8)表明,H-11、H-12和H-94对稻瘟病具有抗性而H-14、H-15和H-151对稻瘟病表现高感。表明抗性表型与基因分型结果一致,Pi2基因KASP标记Pi2-C3具有较高的可靠性。
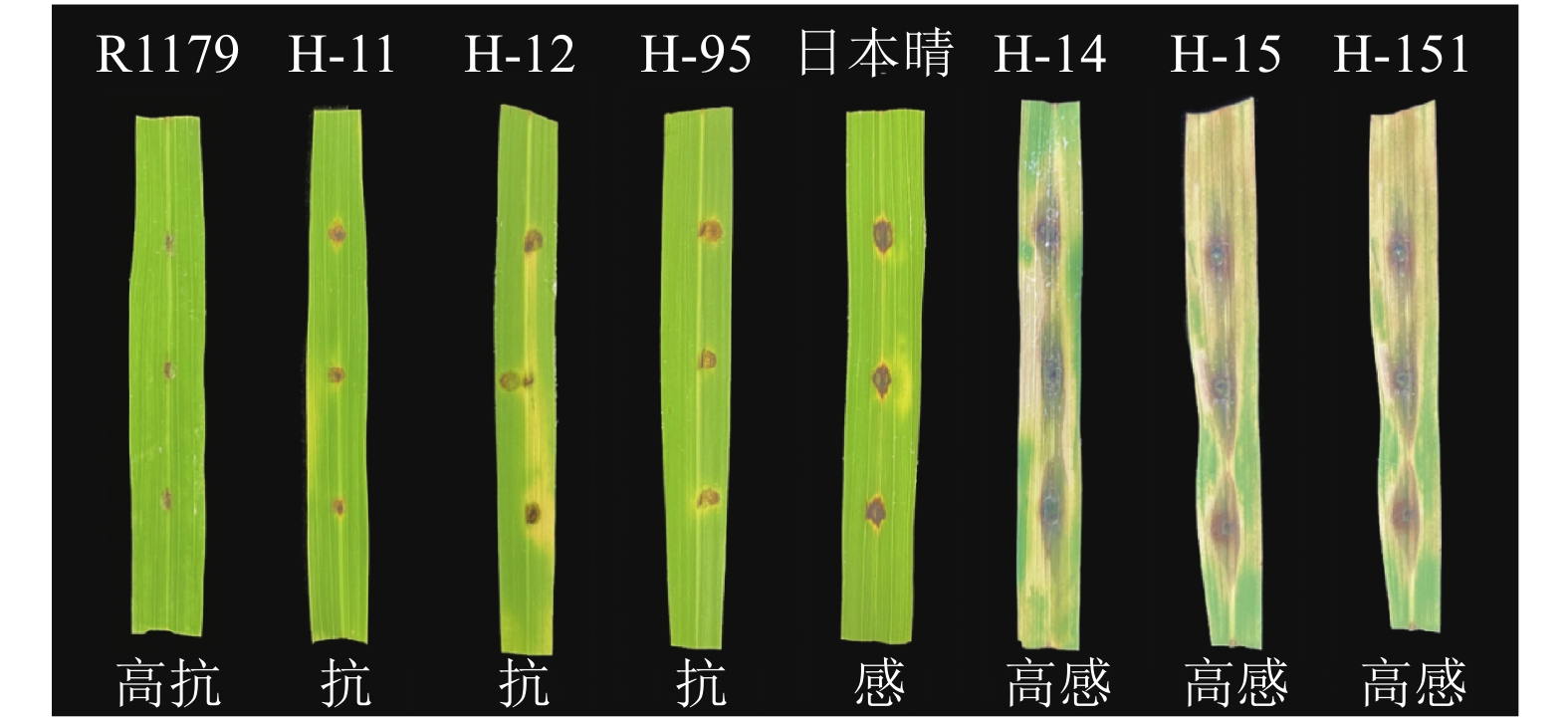
|
图 8 叶片接种表型 Fig. 8 Leaf inoculation phenotype |
利用开发的Pi2-C3抗性标记对2020年晚造中阳江市病圃的84份材料进行KASP基因分型,确定供试材料中Pi2基因的存在情况,同时进行田间病圃抗性评价(表3)。从阳江市病圃材料中,检测到携带Pi2抗性基因的材料46份,占54.76%,未携带Pi2抗性基因的材料38份,占45.24%。结合病圃表型观察,抗性材料68份,包括高抗材料16份、中抗材料25份;感病材料16份,包括高感材料5份、中感材料5份。在后续对病圃检测结果的验证中表明,携带Pi2基因的材料对稻瘟病表现为抗性,其中5份材料表现为高抗,7份表现为中抗。
|
|
表 3 广东阳江病圃材料Pi2基因检测结果及抗性评价 Table 3 Pi2 gene detection results and resistance evaluation of materials from the Yangjiang disease nursery in Guangdong |
培育和种植抗稻瘟病水稻品种可以有效减少稻瘟病造成的损失,利用与抗稻瘟病基因连锁的分子标记进行辅助选择可以提高品种选育的效率。针对抗稻瘟病基因Pi2,先后有不同的研究人员开发了不同类型的分子标记用于该基因的分型。Wu等[26]利用Pi2基因的SSR标记AP22,对277份优质中国水稻亲本材料进行了检测;高利军等[25]基于Pi2基因与日本晴基因序列的差异开发共显性分子标记M-Pi2;李潜龙[27]基于Pi2基因1543~1545处3个碱基的缺失,开发Indel功能标记Pi2-1,使用功能标记Pi2-1辅助选择育种改良文香1号的稻瘟病抗性,抗性表型的选择准确率在90%以上;李冰等[28]利用Pi2位点Nbs4-Pi2及其复等位基因Pi9、Pigm位点Pigm-R2 DNA序列比对结果,开发了Indel标记Pi2-CM1,该标记可有效区分Pi2与其等位基因Pi9和Pigm。除了传统的二代标记,已有研究人员针对Pi2基因开发了一系列SNP标记。华丽霞等[23]开发的dCAPS标记,在PCR扩增后需要通过酶切和电泳进行检测,操作复杂,而金名捺等[24]开发的HRM标记,对PCR扩增体系要求较高,不适合进行大批量筛选。基于KASP的基因分型技术无需电泳,稳定性好,可以有效解决以上问题,杨义强等[29]利用高抗稻瘟病品种‘黄广油占’与高感品种‘广陆矮4号’Pi2基因第787和788密码子上的突变,开发了Pi2基因特异性KASP标记W-Pi2,通过对40个品种和部分F2分离群体的检测发现该标记可以实现对Pi2基因的准确分型。邓钊等[30]利用基于KASP技术开发的针对16个稻瘟病抗性基因的标记组合对隆两优和晶两优系列杂交稻品种及其亲本进行基因型检测,Pia、Pita、Pi2、Pi5和Piz基因在品种中的检出频率相对较高,均达到50%以上,其中Pi2在父本中的检出率达到了71.2%。
本研究在593份自然群体材料中筛选出2个表型不同、亲缘关系较远的材料H-74和H-78,进一步对这2个材料进行单倍型分析,获得Pi2基因抗性位点的核心区域和99个重要SNP位点。结合593份材料的全基因组重测序结果,筛选出关键SNP位点,最后成功开发出KASP特异性分子标记Pi2-C3。该标记被应用于已知基因型的84个自然群体材料的检测中,显示出较高的准确度。同时,从阳江市病圃材料中,选出84份材料进行KASP基因分型和抗性评价,发现46份材料携带Pi2抗性基因,占总体的54.76%,而38份材料未携带Pi2抗性基因,占45.24%。本研究开发的KASP标记专门针对Pi2基因进行检测。然而,在被检测的病圃材料中未携带Pi2抗性基因的材料可能由于携带Pi1、Pita及Piz-t等其他抗性基因而表现出稻瘟病抗性。与以往的研究相比,本研究开发的KASP标记Pi2-C3不需要进行电泳,准确度较高,并且易于实现高通量和自动化。另外,相较于其他KASP分子标记,本研究基于Pi2抗性位点的核心区域进行标记的开发,确保了标记的真实性和可靠性。
3.2 KASP基因分型技术的应用前景目前,SSR标记鉴定体系已被广泛应用,显著提高了品种鉴定的效率和准确性。然而,在水稻基因组中,具有多个等位基因的位点增加了构建DNA指纹库的难度,需要频繁更新数据以避免转换错误。一些通量化的SSR标记检测技术缺乏稳定性。相比之下,SNP标记仅有3个基因型,不受扩增片段大小的影响,可广泛用于DNA指纹图谱的构建,推动了检测技术的发展。基于PCR原理的KASP基因分型技术具有直观的检测结果,适用于高通量检测。与同为SNP检测技术的高分辨率熔解曲线(High resolution melting,HRM)相比,KASP技术成本更低,操作更简便。本研究开发的KASP标记Pi2-C3和SSR标记M-Pi2分别对包含阴阳对照的96个材料进行检测,2种标记检测结果高达95%的一致性。由于KASP和SSR标记是基于不同的原理和标记位点设计的,SSR不是基于基因的标记,其多态性不能完全反映基因的存在,而KASP是基于SNP差异开发的,具有更高的准确性。此外,Pi2区域的某些位点可能在个别个体中呈现差异,这也可能导致KASP和SSR标记的检测结果不一致。另一方面,利用KASP标记进行基因分型可以减少电泳点样的步骤,从而降低试验误差。此外,KASP标记的应用避免了使用有毒化学物质如核酸染料,减少了对人体和环境的潜在危害。KASP的结果更加直观,可以通过颜色区分不同基因型,直接定位到相应的材料。
3.3 结论本研究针对稻瘟病抗性基因Pi2的核心区域,开发了基于KASP基因分型技术的特异性分子标记Pi2-C3,并建立了一套水稻Pi2基因的KASP基因分型体系。该标记能够准确检测水稻材料中Pi2基因,将不同水稻材料的Pi2位点分为T/T、T/A和A/A基因型,其中T/T为抗病基因型。相较于其他分子标记,操作流程更加简便,检测结果更加高效。KASP基因分型检测技术为水稻抗稻瘟病种质资源的鉴定和抗性群体的高通量筛选提供了技术支持,为水稻新时代抗性育种奠定了基础。
| [1] |
向聪, 雷东阳, 任西明, 等. 水稻抗稻瘟病遗传育种研究进展[J]. 作物研究, 2017, 31(5): 547-552. (  0) 0) |
| [2] |
权水清. 水稻稻瘟病菌无毒基因AVR-Pik的共显性标记开发与应用[D]. 武汉: 武汉大学, 2017.
(  0) 0) |
| [3] |
温小红, 谢明杰, 姜健, 等. 水稻稻瘟病防治方法研究进展[J]. 中国农学通报, 2013, 29(3): 190-195. (  0) 0) |
| [4] |
MENG X L, XIAO G, TELEBANCO-YANORIA M J, et al. The broad-spectrum rice blast resistance (R) gene Pita2 encodes a novel R protein unique from Pita
[J]. Rice, 2020, 13(1): 1-15. DOI:10.1186/s12284-019-0361-3 (  0) 0) |
| [5] |
ZHOU B, QU S H, LIU G F, et al. The eight amino-acid differences within three leucine-rich repeats between Pi2 and Piz-t resistance proteins determine the resistance specificity to Magnaporthe grisea
[J]. Molecular Plant-Microbe Interactions, 2006, 19(11): 1216-1228. DOI:10.1094/MPMI-19-1216 (  0) 0) |
| [6] |
LIU G, LU G, ZENG L, et al. Two broad-spectrum blast resistance genes, Pi9(t) and Pi2(t), are physically linked on rice chromosome 6
[J]. Molecular Genetics and Genomics, 2002, 267(4): 472-480. DOI:10.1007/s00438-002-0677-2 (  0) 0) |
| [7] |
朱业宝, 方珊茹, 沈伟峰, 等. 国外引进水稻种质资源的稻瘟病抗性基因检测与评价[J]. 植物遗传资源学报, 2020, 21(2): 418-430. (  0) 0) |
| [8] |
陆展华, 付魏魏, 刘维, 等. 广东省主栽水稻品种稻瘟病主效抗性基因的鉴定及分析[J]. 植物病理学报, 2020, 50(6): 711-722. (  0) 0) |
| [9] |
沈新莲, 张天真. 作物分子标记辅助选择育种研究的进展与展望[J]. 高技术通讯, 2003, 13(2): 105-110. (  0) 0) |
| [10] |
SHABIR G, ASLAM K, KHAN A R, et al. Rice molecular markers and genetic mapping: Current status and prospects[J]. Journal of Integrative Agriculture, 2017, 16(9): 1879-1891. (  0) 0) |
| [11] |
何元浩, 胡凤荣. 基于AFLP分子标记的30个风信子品种遗传多样性分析[J]. 分子植物育种, 2023, 21(9): 2955-2963. (  0) 0) |
| [12] |
李建武. 马铃薯(Solanum tuberosum L. )块茎淀粉含量及植株熟性性状的QTL定位与遗传分析[D]. 武汉: 华中农业大学, 2019.
(  0) 0) |
| [13] |
刘传光, 张桂权. 水稻单核苷酸多态性及其应用[J]. 遗传, 2006, 28(6): 737-744. (  0) 0) |
| [14] |
BEGUM H, SPINDEL J E, LALUSIN A, et al. Genome-wide association mapping for yield and other agronomic traits in an elite breeding population of tropical rice (Oryza sativa)
[J]. PLoS One, 2015, 10(3): e0119873. DOI:10.1371/journal.pone.0119873 (  0) 0) |
| [15] |
王彦云. 基于SNP标记的黄河鲤鱼溯源研究与应用[D]. 北京: 中国农业科学院, 2021.
(  0) 0) |
| [16] |
SEMAGN K, BABU R, HEARNE S, et al. Single nucleotide polymorphism genotyping using Kompetitive Allele Specific PCR (KASP): Overview of the technology and its application in crop improvement[J]. Molecular Breeding, 2014, 33(1): 1-14. DOI:10.1007/s11032-013-9917-x (  0) 0) |
| [17] |
练云, 李海朝, 李金英, 等. 利用KASP标记筛选含rhg1和Rhg4位点的大豆抗病资源
[J]. 植物遗传资源学报, 2021, 22(2): 399-406. (  0) 0) |
| [18] |
王蕊, 施龙建, 田红丽, 等. 玉米杂交种纯度鉴定SNP核心引物的确定及高通量检测方案的建立[J]. 作物学报, 2021, 47(4): 770-779. (  0) 0) |
| [19] |
张瑞, 王洋, HUSSAIN S, 等. 水培条件下水稻全生育期耐盐筛选鉴定[J]. 植物遗传资源学报, 2021, 22(6): 1567-1581. (  0) 0) |
| [20] |
韩美丽, 陆荣生, 霍秀娟, 等. 水稻离体叶抗稻瘟病鉴定方法研究[J]. 安徽农业科学, 2009, 37(13): 6037-6039. (  0) 0) |
| [21] |
SU J, WANG W, HAN J, et al. Functional divergence of duplicated genes results in a novel blast resistance gene Pi50 at the Pi2/9 locus
[J]. Theoretical and Applied Genetics, 2015, 128: 2213-2225. (  0) 0) |
| [22] |
汪文娟, 周继勇, 汪聪颖, 等. 八个抗稻瘟病基因在华南籼型杂交水稻中的分布[J]. 中国水稻科学, 2017, 31(3): 299-306. (  0) 0) |
| [23] |
华丽霞, 汪文娟, 陈深, 等. 抗稻瘟病Pi2/9/z-t基因特异性分子标记的开发
[J]. 中国水稻科学, 2015, 29(3): 305-310. (  0) 0) |
| [24] |
金名捺, 陈竹锋, 丘式浚, 等. 基于HRM体系的稻瘟病抗性基因Pi2特异性分子标记的开发及应用
[J]. 农业生物技术学报, 2018, 26(3): 365-373. (  0) 0) |
| [25] |
高利军, 高汉亮, 颜群, 等. 4个抗稻瘟病基因分子标记的建立及在水稻亲本中的分布[J]. 杂交水稻, 2010, 25(S1): 294-298. (  0) 0) |
| [26] |
WU Y, XIAO N, YU L, et al. Combination patterns of major R genes determine the level of resistance to the M. oryzae in Rice (Oryza sativa L. )
[J]. PLoS One, 2015, 10(6): e0126130. DOI:10.1371/journal.pone.0126130 (  0) 0) |
| [27] |
李潜龙. 水稻抗稻瘟病基因Pi2功能标记的开发及育种应用[D]. 长沙: 湖南农业大学, 2018.
(  0) 0) |
| [28] |
李冰, 李永聪, 刘芝妤, 等. 水稻抗稻瘟病基因Pi2特异分子标记的开发与应用
[J]. 分子植物育种, 2021, 19(8): 2638-2643. (  0) 0) |
| [29] |
杨义强, 朱林峰, 李晓芳, 等. 抗稻瘟病基因Pi2的基因特异性KASP标记开发与应用
[J]. 植物遗传资源学报, 2021, 22(5): 1314-1321. (  0) 0) |
| [30] |
邓钊, 江南, 符辰建, 等. 隆两优与晶两优系列杂交稻的稻瘟病抗性基因分析[J]. 作物学报, 2022, 48(5): 1071-1080. (  0) 0) |
 2023, Vol. 44
2023, Vol. 44


