水稻是我国的主要粮食作物,对保障我国粮食安全做出了重要贡献[1]。水稻的生产起始于种子的播种和萌发,结束于种子的成熟和收获。因此,种子的发育和成熟是影响水稻产量和品质形成的关键阶段。近年来,有多项研究利用蛋白质组学技术对水稻种子性状进行了探究,包括种子发育[2-5]、种子萌发[6-7]、种子休眠[8]等。但是,关注种子成熟干燥阶段的蛋白质组学报道较少,对于水稻种子成熟期蛋白水平上的变化规律和调控机制尚需进一步研究。
蛋白质组学是一个快速发展的生物学研究领域,广泛应用于研究作物的生长、发育、生物和非生物胁迫。传统的凝胶双向电泳技术分离的蛋白数量少,近年来逐渐发展出了一些无凝胶的新方法,分为基于标签(Label)和无标签(Label-free)两种类型[9] 。Label-free无需使用同位素标记,而是通过液−质联用技术对酶解肽段进行质谱鉴定,基于肽段的信号强度对肽段对应的蛋白质进行相对定量。4D label-free是传统无标签技术的升级,具有更好的灵敏度和覆盖深度。
本研究,以授粉后30 d的水稻成熟种子为材料进行4D label-free蛋白组学研究,获得了蛋白的表达谱,分析了蛋白的亚细胞定位、结构域、GO功能注释和KEGG通路,对揭示水稻种子成熟脱水期的蛋白积累模式和挖掘种子成熟的关键蛋白具有参考意义。
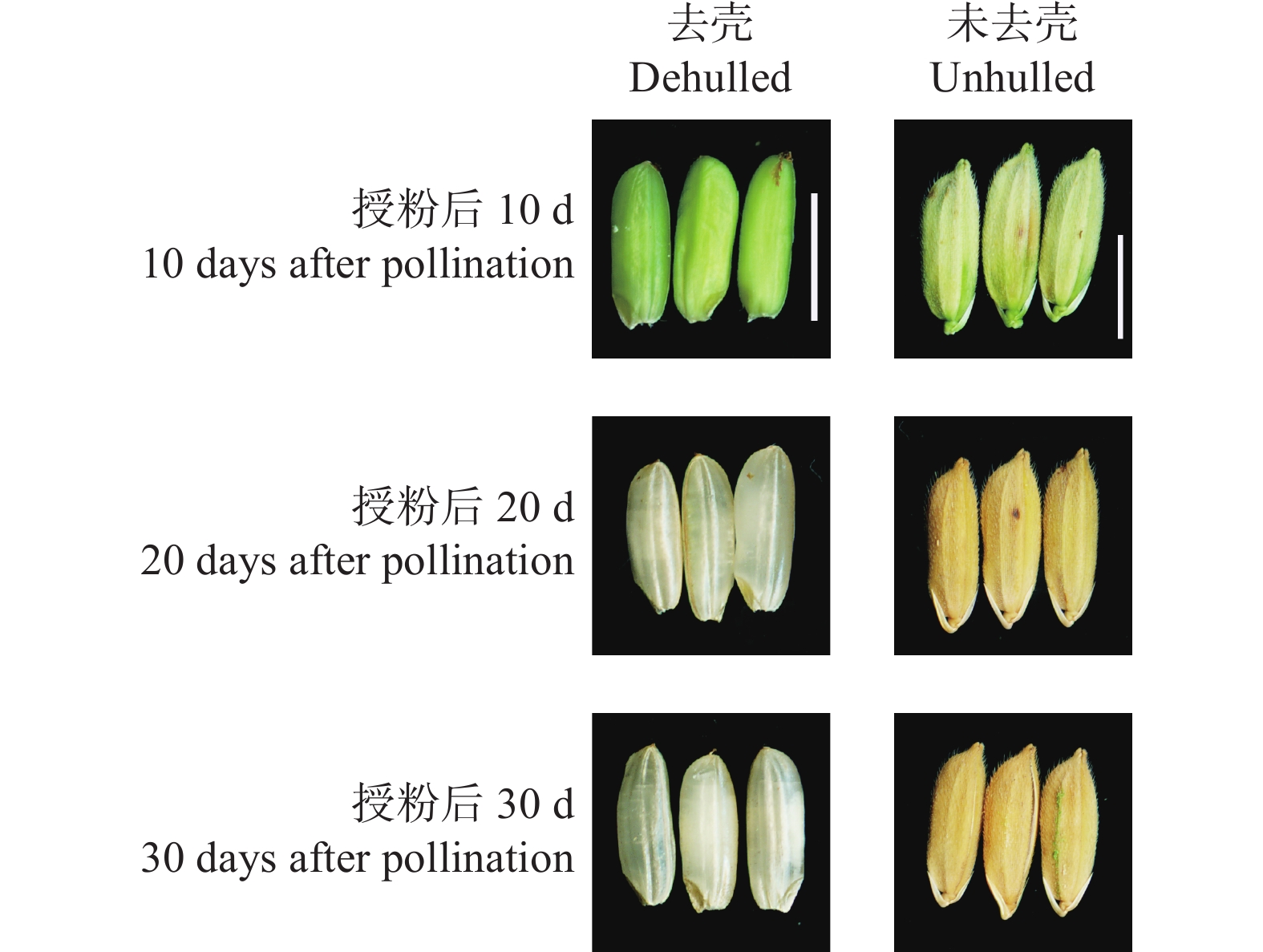
1 材料与方法 1.1 试验材料本研究所用的水稻材料是染色体单片段代换系,受体亲本是‘华粳籼74’,供体亲本是‘Khazar’,代换片段的分子标记是PSM141--RM436-PSM142--PSM143,“--”表示发生交换的位置。染色体单片段代换系与受体‘华粳籼74’的种子发育和成熟过程相似,但染色体单片段代换系具有更强的休眠特性。在授粉后30 d,收集健康、饱满的成熟种子,去壳后,液氮保存,委托上海中科新生命生物科技有限公司用于4D label-free蛋白质组学分析(图1)。设置 3个生物学重复。
|
图 1 不同发育阶段水稻种子的形态 Fig. 1 Morphology of rice seeds at different development stages 图中比例尺长度为0.5 cm The scale bars in figure are 0.5 cm |
样品用SDT(40 g·L−1 SDS,1 mmol/L DTT,100 mmol/L Tris-HCl,pH7.6)缓冲液提取蛋白质,用BCA蛋白测定试剂盒(Bio-Rad)定量蛋白质。每个样品取适量蛋白质用过滤器辅助样品制备法(Filter-aided sample preparation,FASP)进行胰蛋白酶(Promega)消化蛋白质,采用C18 Cartridge (Sigma)进行肽段脱盐,冻干后加入体积分数为0.1%的甲酸溶液40 μL复溶,在280 nm的紫外光下测定肽段溶液的光密度(Optical density,OD),对肽段进行定量。
1.3 SDS-PAGE每个样品取 20 µg 蛋白质,分别与 5X 上样缓冲液混合后煮沸 5 min,在 125 g·L−1 SDS-PAGE 凝胶上进行电泳(恒流 14 mA,90 min),最后通过考马斯亮蓝 R-250 染色观察蛋白质条带。
1.4 LC-MS/MS数据采集每份样品采用纳升流速的HPLC液相系统Easy nLC (Thermo Scientific)进行分离。缓冲液A液是体积分数为0.1%的甲酸水溶液,B液是体积分数为0.1%的甲酸乙腈水溶液。色谱柱以95%的A液平衡,样品由自动进样器进到Acclaim PepMap100上样柱(Thermo Scientific)中,经过EASY column分析柱(Thermo scientific)分离,流速为300 nL/min。色谱分离后的样品用timsTOF Pro质谱仪(Bruker)进行正离子模式检测。质谱扫描范围m/z设置为100~1700。数据采集采用平行累积串行碎裂(Parallel accumulation-serial fragmentation,PASEF)模式,一个循环窗口时间为1.17 s。电荷数在0~5范围内的二级谱图,串联质谱扫描的动态排除时间设置为24 s,避免母离子的重复扫描。
1.5 蛋白质鉴定和定量分析对质谱分析的原始数据采用 MaxQuant 软件(版本号 1.6.14)进行查库鉴定及定量分析。可信肽段和可信蛋白质的筛选标准是FDR≤0.01,采用唯一肽段和Razor肽段的定量强度值进行蛋白质定量,定量方法是LFQ (Label free quantitation)法。
1.6 生物信息学分析亚细胞定位分析使用CELLO( http://cello.life.nctu.edu.tw/),在网站输入候选蛋白FASTA格式的氨基酸序列进行预测;蛋白结构域分析使用Pfam数据库( http://pfam.xfam.org/),通过InterProScan软件包对目标蛋白序列进行功能表征,以获得结构域注释信息;对目标蛋白质集合利用Blast2GO进行GO(Gene ontology,http://geneontology.org/)注释,包括序列比对、GO条目提取、GO注释和InterProScan补充注释等4个步骤;利用KAAS (KEGG automatic annotation server) 软件进行KEGG(Kyoto encyclopedia of genes and genomes,https://www.genome.jp/kegg/)通路注释;采用Fisher精确检验,比较目标蛋白质集合和总体蛋白质集合中各个GO分类(或KEGG通路、或Domain)的分布情况,以进行富集分析。
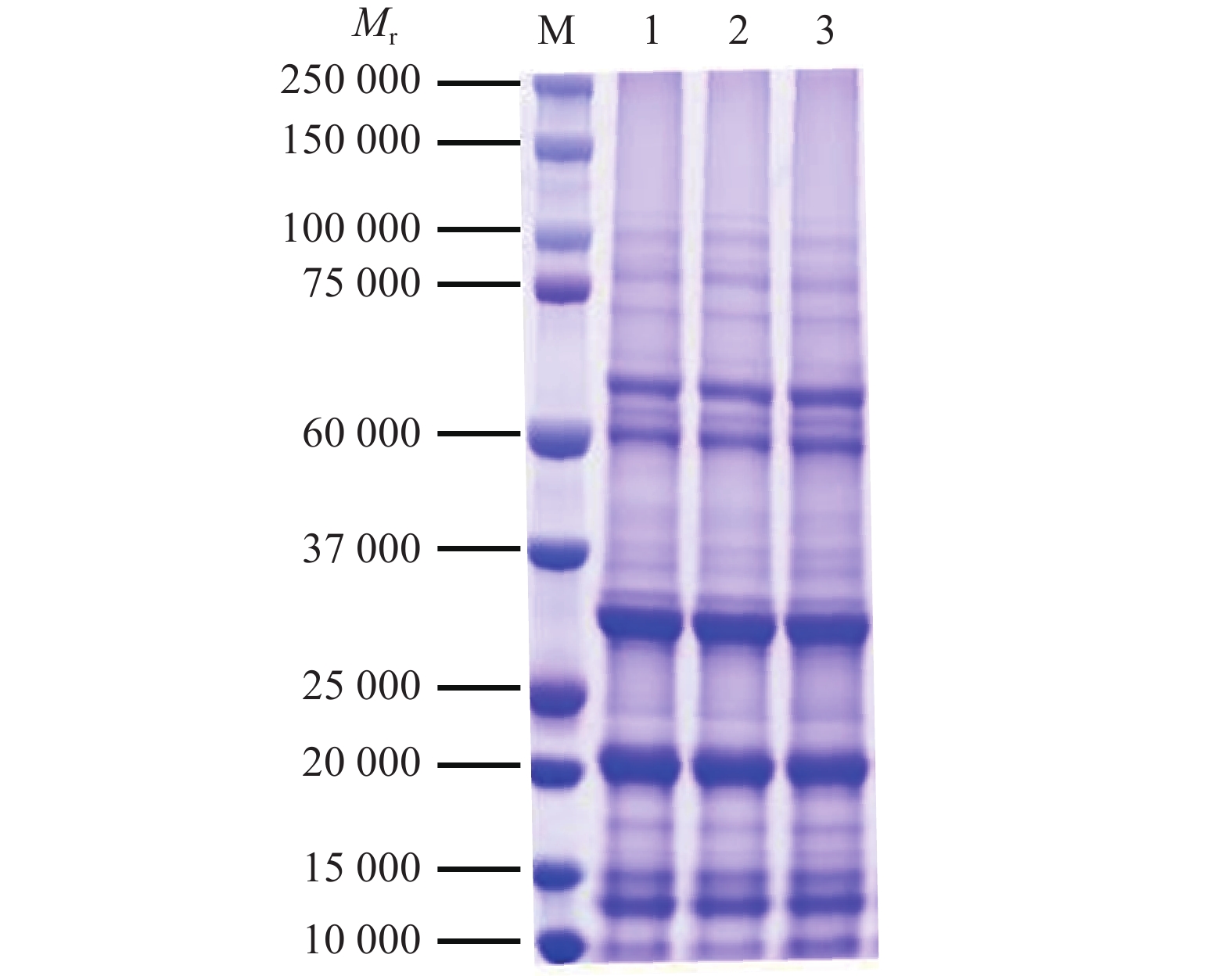
2 结果与分析 2.1 蛋白SDS-PAGE电泳提取水稻去壳种子的总蛋白后进行SDS-PAGE检测。结果表明,所提取的3个生物学重复样品的蛋白含量丰富,条带清晰,没有发生降解,蛋白的数量和质量均满足后续试验的要求(图2)。
|
图 2 总蛋白SDS-PAGE电泳图 Fig. 2 SDS-PAGE electrophoresis of total protein M:蛋白Marker;1~3:蛋白样品 M: Protein marker; 1−3: Protein samples |
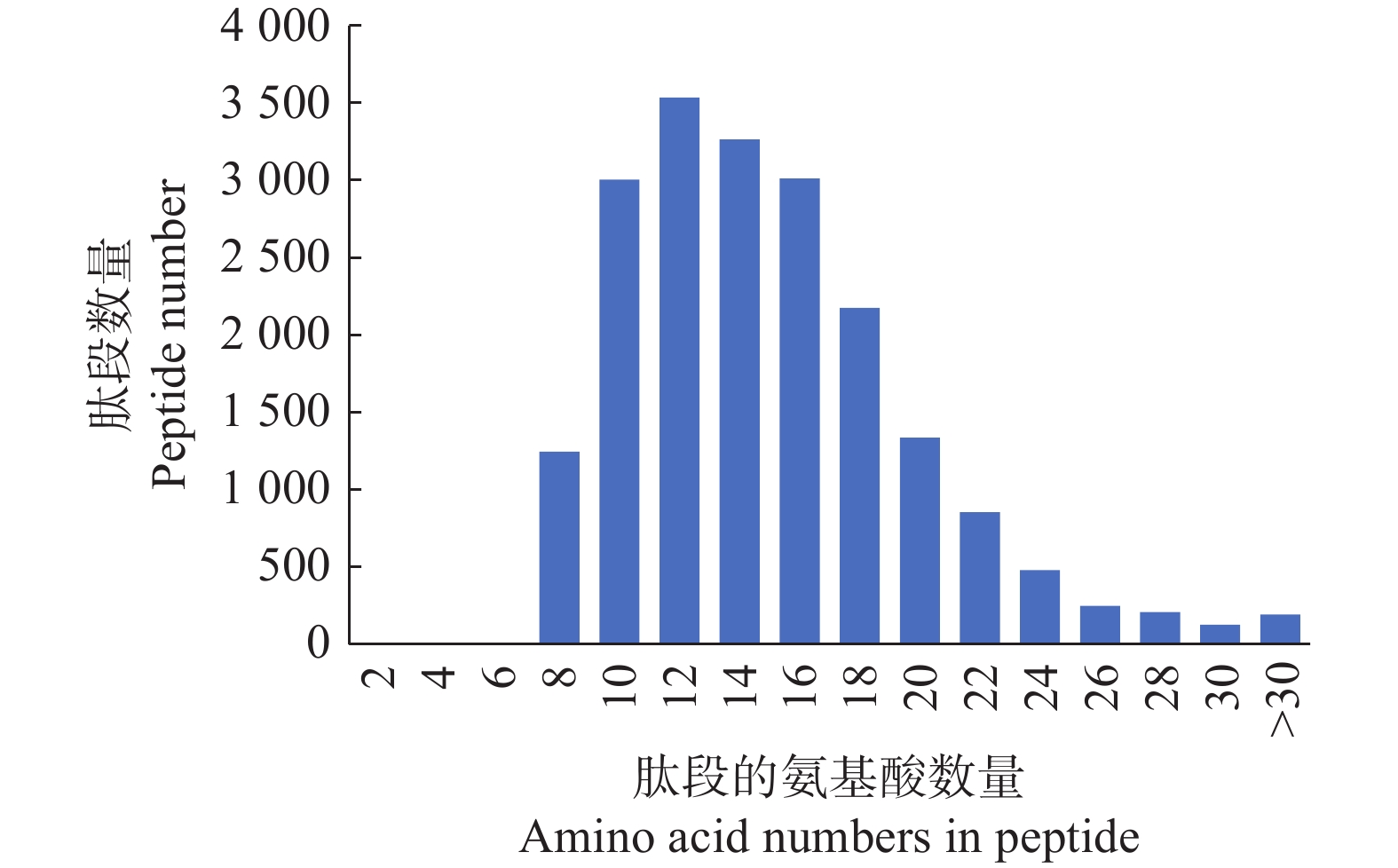
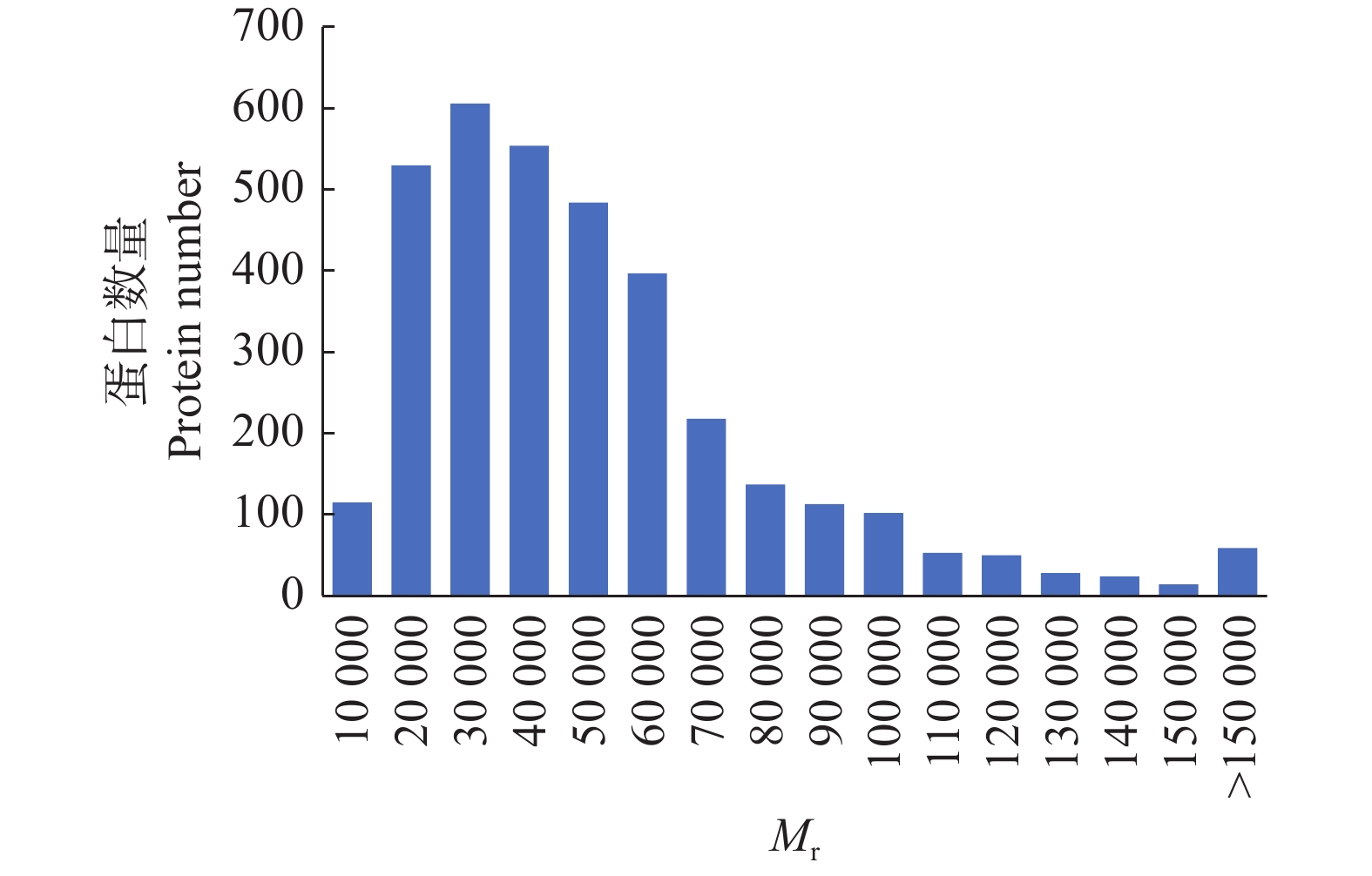
对酶解后的总蛋白进行质谱鉴定,总共鉴定到19 688个可信度高的肽段,肽段的氨基酸数量主要集中在8~20个之间,占比89.3%(图3)。上述肽段对应3 484个蛋白,蛋白数量丰富,蛋白质的相对分子质量主要在0~100 000 之间,占比93.5% (图4)。
|
图 3 肽段的氨基酸数量分布图 Fig. 3 Distribution of amino acid numbers in peptide |
|
图 4 蛋白相对分子质量分布图 Fig. 4 Distribution of protein relative molecular mass |
蛋白质定位的亚细胞器是蛋白发挥功能的重要场所,分析蛋白质的亚细胞定位有助于进一步探究蛋白质在细胞中的功能。如图5所示,水稻成熟种子中的蛋白质几乎存在于细胞内所有的亚细胞器中,其中细胞质、细胞核、叶绿体、线粒体、质膜和胞外是蛋白定位最多的场所,分别有1 381、949、807、571、447和433个蛋白。
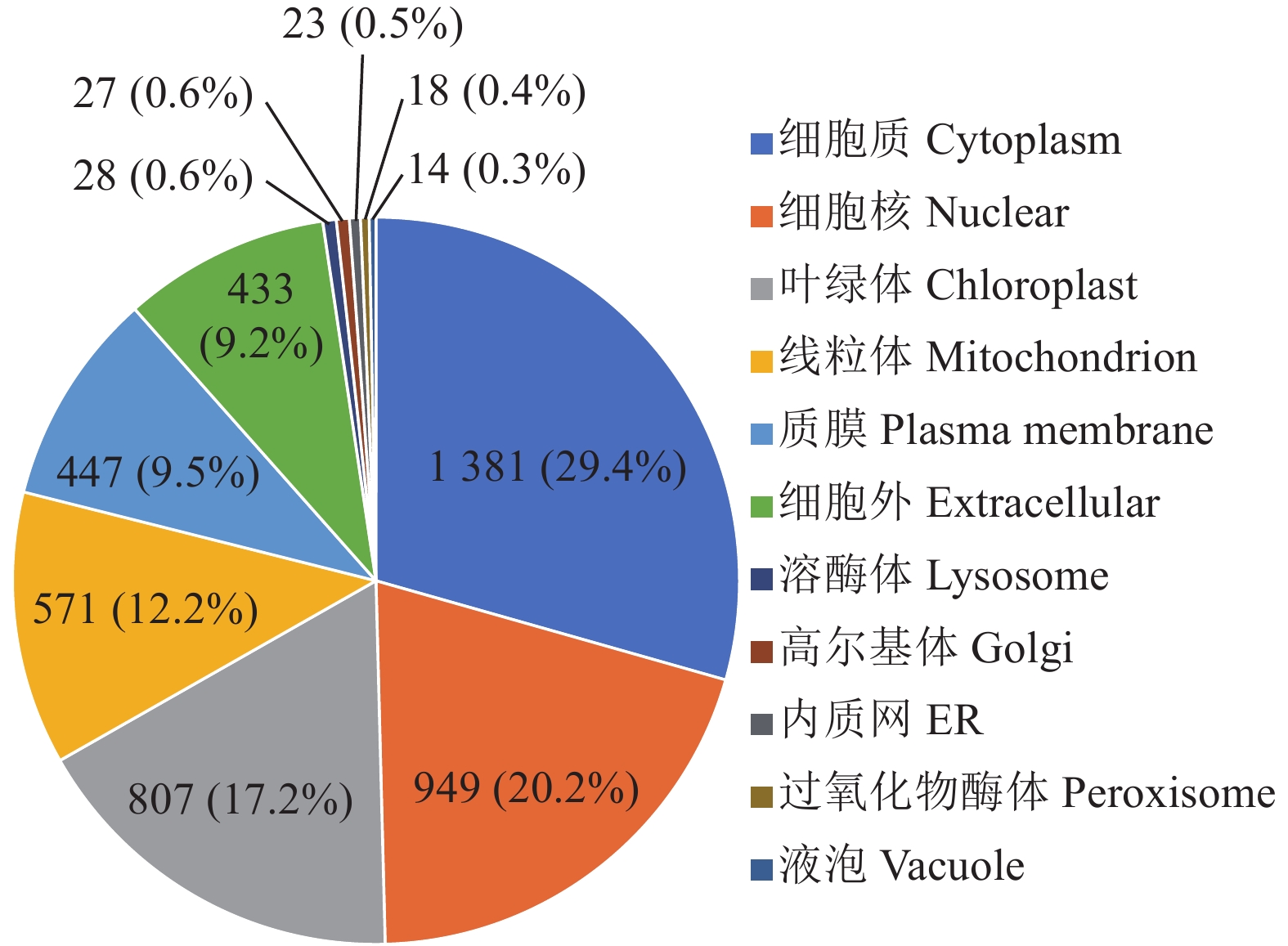
|
图 5 亚细胞定位分析 Fig. 5 Subcellular localization analysis 括号中数据为占比 The data in brackets are the proportion |
蛋白质结构域是一个蛋白中可以进行独立折叠和发挥功能的基本单位,预测蛋白的结构域对了解蛋白的生物学功能具有重要意义。对水稻成熟种子中鉴定的蛋白质进行结构域分析,结果发现排名前20的结构域分别是RNA识别基序、蛋白激酶结构域、WD结构域/G-beta重复、Cupin结构域、Ras家族、ATP酶家族、蛋白酶抑制剂/种子贮藏/LTP家族、EF-hand结构域、蛋白酪氨酸激酶、热激蛋白70、钙调磷酸酶样磷酸酯酶、硫氧还蛋白、TCP-1/cpn60伴侣蛋白家族、蛋白酶体亚基、PCI结构域、延伸因子Tu GTP结合域、过氧化物酶、糖基转移酶组1、通用应激蛋白家族、谷胱甘肽S−转移酶N端结构域(图6)。
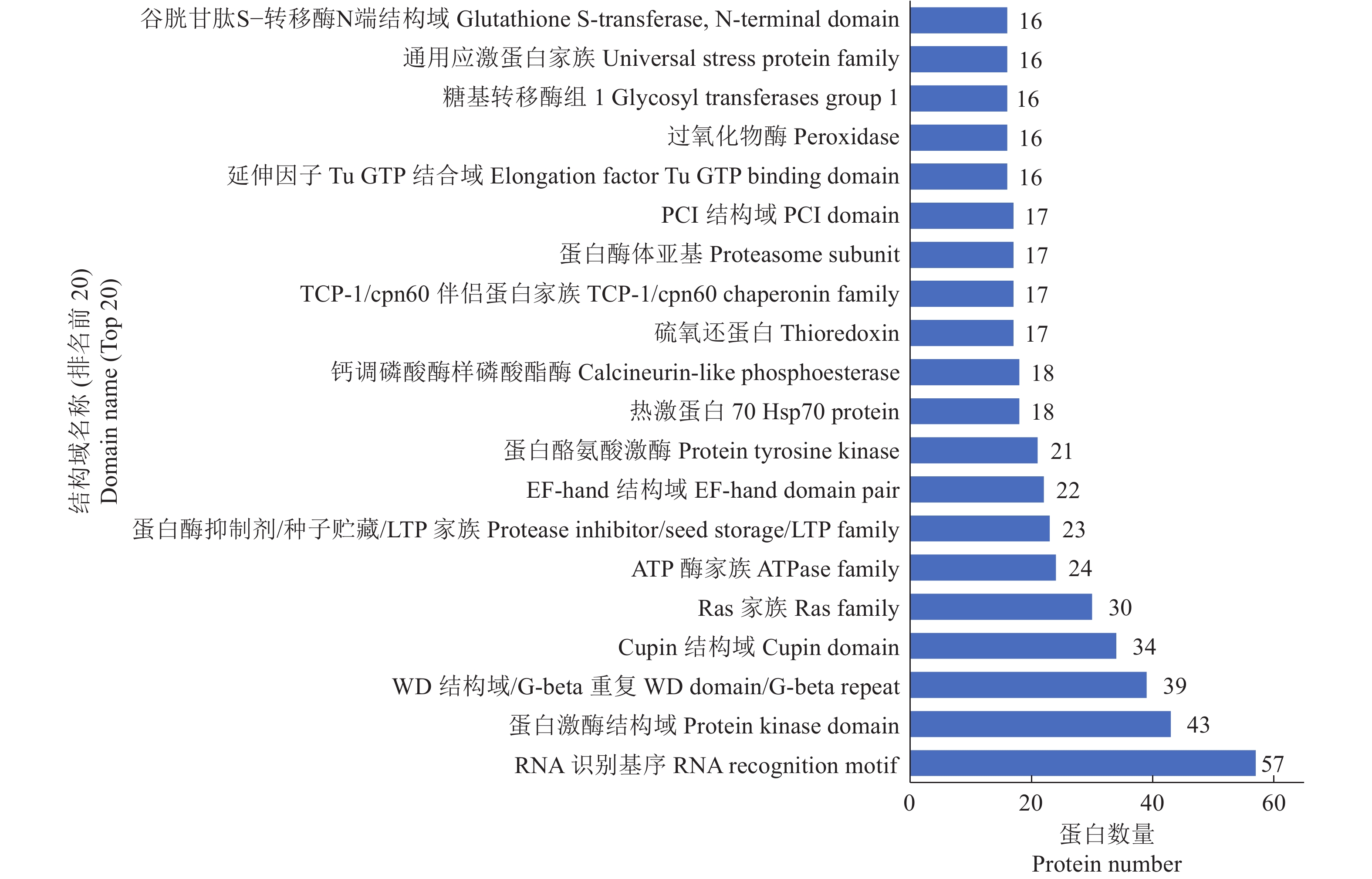
|
图 6 蛋白结构域分析 Fig. 6 Protein domain analysis |
为了全面了解蛋白在水稻种子发育成熟过程中参与的生物学过程、功能和定位,通过基因本体GO对鉴定到的蛋白质进行注释(图7)。在生物学过程(Biological process)中,共注释到16个子类,主要参与了细胞过程、代谢过程、定位、生物调节、生物发生、响应刺激、生物过程的调节、发育过程和信号过程等。在分子功能(Molecular function)上,共注释到8个子类,主要发挥催化活性、结合、结构分子活性、转运活性、分子功能调节剂、营养库活动、抗氧化活性和分子传感器活性。细胞组分(Cellular component)共注释到11个子类,主要构成了细胞、细胞组分、细胞器、细胞膜和蛋白质复合物等结构。
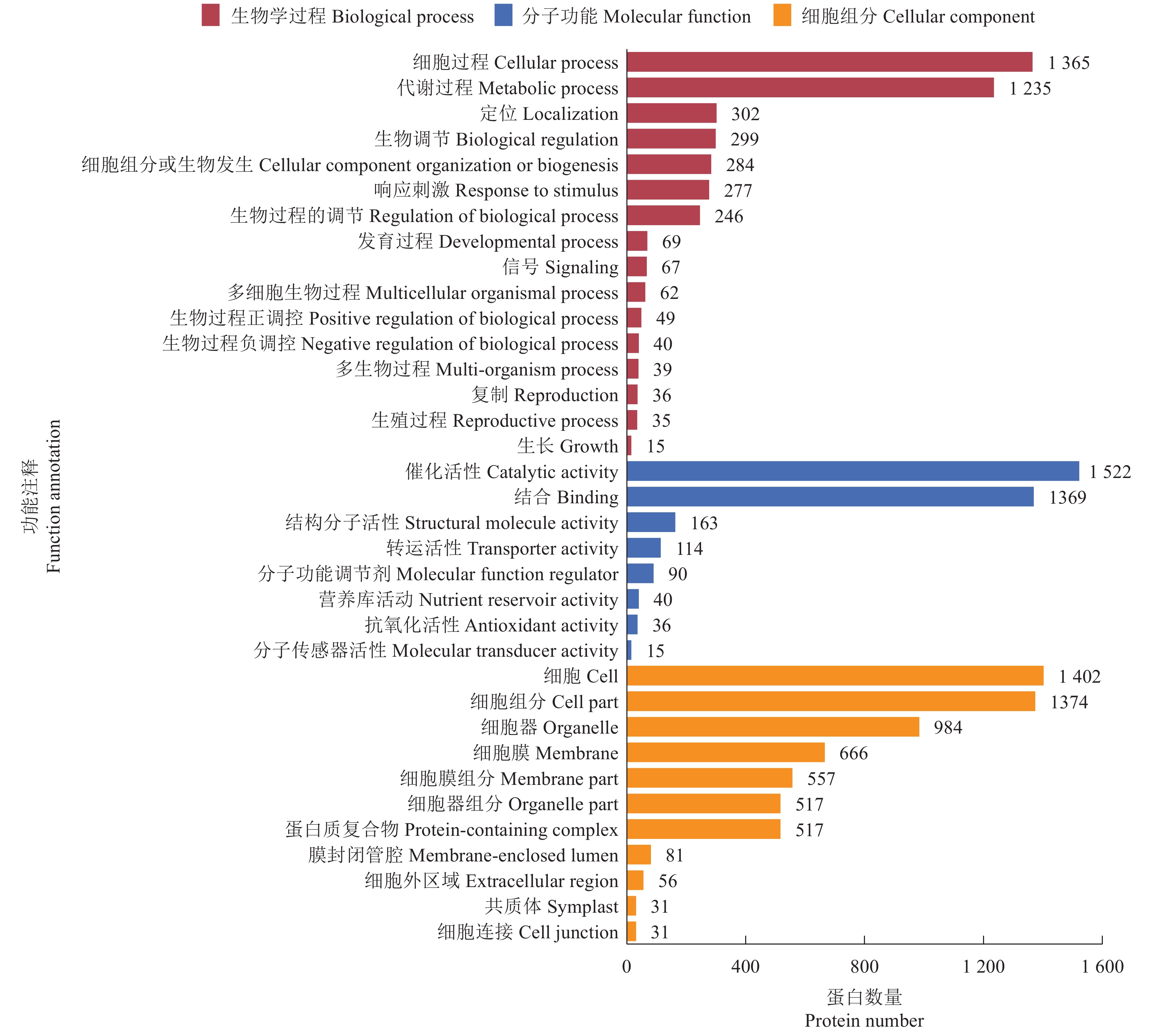
|
图 7 GO功能注释 Fig. 7 GO function annotation |
为了更系统地解析水稻种子成熟的生物学事件,对鉴定到的蛋白进行KEGG代谢通路和归属关系注释。如图8所示,排名前20的代谢通路可以分为8个大类,其中2个通路归属于运输和分解代谢,2个通路归属于翻译,1个通路归属于转录,1个通路归属于全局和概览图,2个通路归属于折叠、分类和降解,2个通路归属于能量代谢,8个通路归属于碳水化合物代谢,1个通路归属于氨基酸代谢。所有通路中,核糖体通路、内质网中的蛋白质加工通路、糖酵解通路和氧化磷酸化通路中注释到的蛋白数量最多,表明这些通路对水稻种子的成熟非常重要。
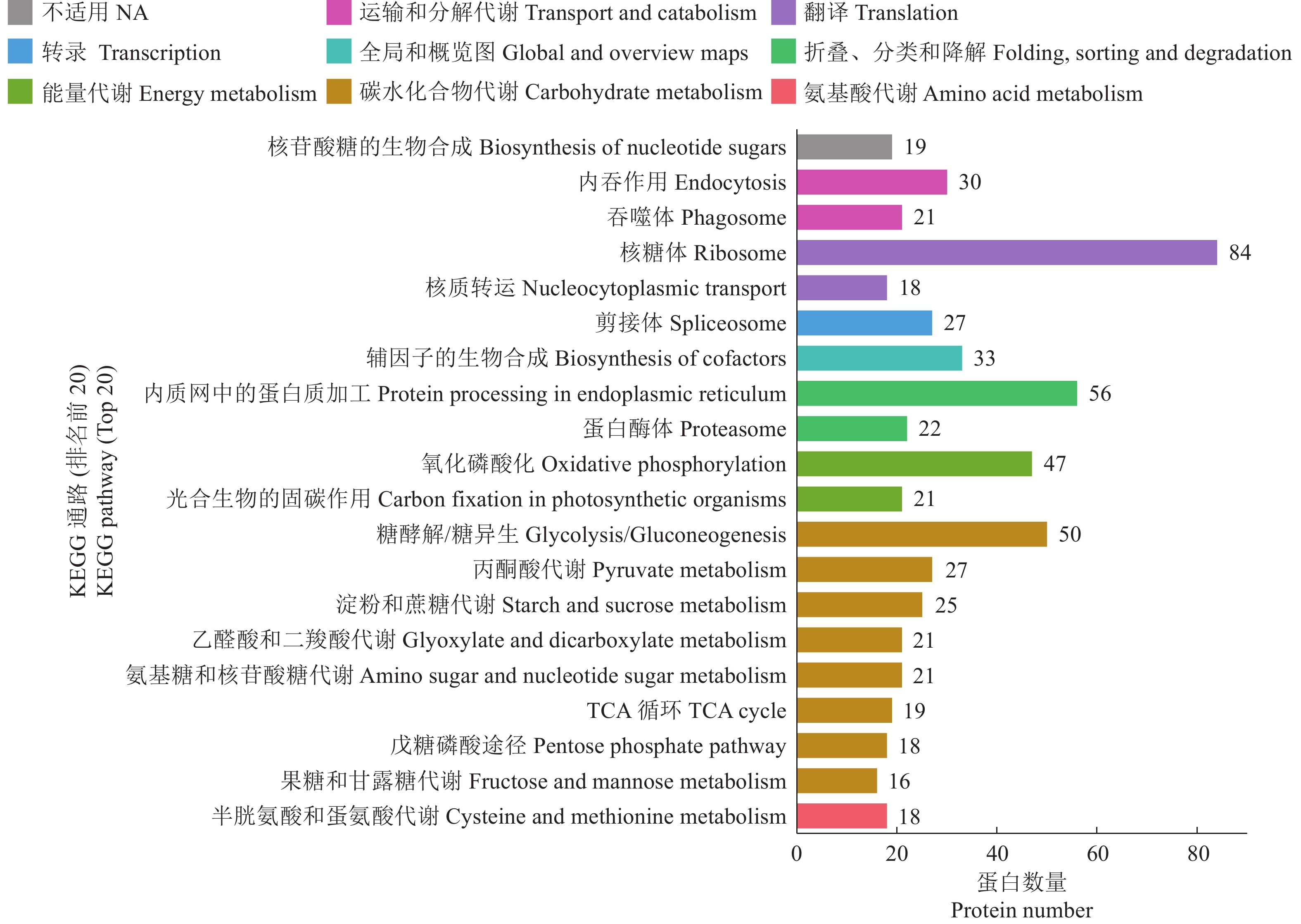
|
图 8 KEGG通路注释 Fig. 8 KEGG pathway annotation |
植物激素和转录因子在种子发育成熟中扮演了重要角色。通过分析水稻成熟种子中的蛋白质,鉴定了十几个植物激素和转录因子相关的蛋白(表1)。在脱落酸途径中,鉴定了2个脱落酸(Abscisic acid,ABA)受体OsRCAR10和OsRCAR7和1个蛋白激酶OsSAPK7。在生长素途径中,鉴定了2个吲哚乙酸(Indoleacetic acid ,IAA)−氨基酸水解酶OsILL8 和OsILL1、1个生长素抑制蛋白OsARP1、1个生长素氧化双加氧酶DAO。在赤霉素途径中,鉴定了2个α−淀粉酶OsAmy3E和OsAmy4A和1个β−淀粉酶OsBmy7。转录因子方面则主要鉴定了5个NAC (NAM、ATAF1/2和CUC2)家族的转录因子,说明其在水稻种子成熟时发挥重要作用。
|
|
表 1 成熟水稻种子中的植物激素和转录因子相关蛋白 Table 1 Proteins related to plant hormone and transcription factor in mature rice seeds |
水稻种子的发育可以划分为胚胎发生、籽粒灌浆和种子成熟3个阶段,在最后的种子成熟阶段水分大量流失,最终形成干燥、休眠状态的种子[10]。淀粉是水稻产量和品质的主要决定因素,我们发现淀粉合成酶(A0A076FRI5)在所有鉴定的蛋白质中质谱信号最强,高达379673333,表明种子成熟时淀粉的合成非常旺盛。另外,有意思的是我们在成熟种子中鉴定了2个α−淀粉酶OsAmy3E和OsAmy4A和1个β−淀粉酶OsBmy7,同时也鉴定了许多的α−淀粉酶抑制剂。有研究表明水稻种子发育阶段的热应激诱导了α−淀粉酶基因的表达,包括Amy1A、Amy1C、Amy3A、Amy3D和Amy3E[11]。但是,一般认为α−淀粉酶是在种子萌发过程中被植物激素赤霉素(Gibberellic acid, GA)诱导产生。本研究的结果证明,种子成熟过程中也存在一定水平的α−淀粉酶积累。在胚胎成熟的早期阶段,α−淀粉酶基因的高水平表达有助于将暂时储存的淀粉转化为糖,并快速满足能量和底物的需求[12]。但是,目前暂不清楚α−淀粉酶在成熟种子中的作用,其产生的原因和作用有待进一步深入研究。
贮藏蛋白也是水稻成熟种子的主要化学组分。结构域注释发现,排名第1的是RNA识别基序,含有这类基序的蛋白主要参与RNA的加工和蛋白的翻译,KEGG中富集蛋白数量最多的也是参与翻译的核糖体通路和蛋白质加工通路。结构域排名第4的是主要存在于植物种子贮藏蛋白中的Cupin,质谱信号排名前10的蛋白中有8个是Cupin蛋白。近期的研究还发现,编码Cupin结构域的种子贮藏蛋白基因与种子活力之间也存在密切的关系[13]。此外,蛋白激酶结构域也广泛存在,暗示蛋白磷酸化是种子成熟期的重要事件。淀粉合成、贮藏蛋白合成和蛋白修饰都要消耗能量,这需要大量的ATP供应。与此吻合的是,我们注意到在KEGG通路分析中,有大量关于能量代谢和碳水化合物代谢(呼吸作用)的通路,这可以为种子的成熟提供能量保障。
除了淀粉和蛋白质的合成,种子成熟后期另一个重要事件是脱水干燥。前人的研究表明,LEA蛋白(Late embryogenesis abundant protein)、热激蛋白(Heat shock protein, HSP)和抗氧化物质的积累与脱水密切相关[14-16],这些蛋白也大量出现在我们的质谱鉴定结果中。ABA被认为是种子成熟脱水的主要调控因子。ABA信号的核心通路包括ABA受体蛋白PYR/PYL/RCAR、PP2C和SnRK2三大组件。我们鉴定了OsRCAR10、OsRCAR7和OsSAPK7等3个ABA信号途径的正调控蛋白,说明ABA信号在水稻种子成熟中发挥重要作用,这与前人的研究结果[17]相吻合。此外,维持一定的ABA信号强度也有利于种子初级休眠性的建立。生长素IAA被认为是ABA之外另一个影响种子成熟的重要植物激素,IAA的积累会诱导种子的初级休眠[18-19]。我们的研究鉴定了2个IAA−氨基酸水解酶OsILL8和OsILL1以及IAA氧化酶DAO,前者可以提高游离IAA的浓度,而后者通过氧化作用降低IAA水平,说明种子成熟过程需要精准调节IAA的水平。高水平IAA所在的区域对同化产物具有“拉力”,从而调节贮藏物质在种子不同部位的积累[20]。
综上,水稻种子成熟期蛋白质组学的研究对于理解水稻种子生物学特性和提高水稻产量和品质具有重要意义。本研究利用4D label-free蛋白组学鉴定了水稻种子在成熟期蛋白的表达谱,获得了高深度、高精度的质谱数据,丰富了水稻种子成熟期的蛋白研究,为深入开展水稻种子成熟调控提供了理论依据。
| [1] |
余泓, 王冰, 陈明江, 等. 水稻分子设计育种发展与展望[J]. 生命科学, 2018, 30(10): 1032-1037. (  0) 0) |
| [2] |
SERA Y, HANAMATA S, SAKAMOTO S, et al. Essential roles of autophagy in metabolic regulation in endosperm development during rice seed maturation[J]. Scientific Reports, 2019, 9: 18544. DOI:10.1038/s41598-019-54361-1 (  0) 0) |
| [3] |
YOU C C, CHEN L, HE H B, et al. iTRAQ-based proteome profile analysis of superior and inferior spikelets at early grain filling stage in japonica rice
[J]. BMC Plant Biology, 2017, 17: 100. DOI:10.1186/s12870-017-1050-2 (  0) 0) |
| [4] |
ZHANG H D, CHEN J N, SHAN S L, et al. Proteomic profiling reveals differentially expressed proteins associated with amylose accumulation during rice grain filling[J]. BMC Genomics, 2020, 21: 714. DOI:10.1186/s12864-020-07105-9 (  0) 0) |
| [5] |
TAPPIBAN P, YING Y N, XU F F, et al. Proteomics and post-translational modifications of starch biosynthesis-related proteins in developing seeds of rice[J]. International Journal of Molecular Sciences, 2021, 22(11): 5901. DOI:10.3390/ijms22115901 (  0) 0) |
| [6] |
XU E S, CHEN M M, HE H, et al. Proteomic analysis reveals proteins involved in seed imbibition under salt stress in rice[J]. Frontiers in Plant Science, 2017, 7: 2006. DOI:10.3389/fpls.2016.02006 (  0) 0) |
| [7] |
CHU C, YAN N, DU Y M, et al. iTRAQ-based proteomic analysis reveals the accumulation of bioactive compounds in Chinese wild rice (Zizania latifolia) during germination
[J]. Food Chemistry, 2019, 289: 635-644. DOI:10.1016/j.foodchem.2019.03.092 (  0) 0) |
| [8] |
XU H B, LIAN L, JIANG M R, et al. Proteomic analysis reveals different involvement of proteins during the maintenance and release of rice seed dormancy[J]. Molecular Breeding, 2019, 39: 60. DOI:10.1007/s11032-019-0963-x (  0) 0) |
| [9] |
TAN B C, LIM Y S, LAU S. Proteomics in commercial crops: An overview[J]. Journal of Proteomics, 2017, 169: 176-188. DOI:10.1016/j.jprot.2017.05.018 (  0) 0) |
| [10] |
AN L, TAO Y, CHEN H, et al. Embryo-endosperm interaction and its agronomic relevance to rice quality[J]. Frontiers in Plant Science, 2020, 11: 587641. DOI:10.3389/fpls.2020.587641 (  0) 0) |
| [11] |
HAKATA M, KURODA M, MIYASHITA T, et al. Suppression of α-amylase genes improves quality of rice grain ripened under high temperature
[J]. Plant Biotechnology Journal, 2012, 10(9): 1110-1117. DOI:10.1111/j.1467-7652.2012.00741.x (  0) 0) |
| [12] |
DAMARIS R N, LIN Z Y, YANG P F, et al. The rice alpha-amylase, conserved regulator of seed maturation and germination[J]. International Journal of Molecular Sciences, 2019, 20(2): 450. DOI:10.3390/ijms20020450 (  0) 0) |
| [13] |
PENG L L, SUN S, YANG B, et al. Genome-wide association study reveals that the cupin domain protein OsCDP3.10 regulates seed vigour in rice[J]. Plant Biotechnology Journal, 2022, 20(3): 485-498. DOI:10.1111/pbi.13731 (  0) 0) |
| [14] |
宋松泉, 刘军, 唐翠芳, 等. 种子耐脱水性的生理及分子机制研究进展[J]. 中国农业科学, 2022, 55(6): 1047-1063. (  0) 0) |
| [15] |
SMOLIKOVA G, LEONOVA T, VASHURINA N, et al. Desiccation tolerance as the basis of long-term seed viability[J]. International Journal of Molecular Sciences, 2021, 22(1): 101. DOI:10.3390/ijms22010101 (  0) 0) |
| [16] |
ALI F, QANMBER G, LI F G, et al. Updated role of ABA in seed maturation, dormancy, and germination[J]. Journal of Advanced Research, 2022, 35: 199-214. DOI:10.1016/j.jare.2021.03.011 (  0) 0) |
| [17] |
宋松泉, 唐翠芳, 雷华平, 等. ABA调控种子发育的研究进展[J/OL]. 广西植物, 2023 [2023-07-29]. https://kns.cnki.net/kcms2/detail/45.1134.Q.20230620.1121.004.html.
(  0) 0) |
| [18] |
MATILLA A J. Auxin: hormonal signal required for seed development and dormancy[J]. Plants, 2020, 9(6): 705. DOI:10.3390/plants9060705 (  0) 0) |
| [19] |
LIU X D, ZHANG H, ZHAO Y, et al. Auxin controls seed dormancy through stimulation of abscisic acid signaling by inducing ARF-mediated ABI3 activation in Arabidopsis
[J]. Proceedings of the National Academy of Sciences, 2013, 110(38): 15485-15490. DOI:10.1073/pnas.1304651110 (  0) 0) |
| [20] |
田淑兰, 王熹. IAA与亚种间杂交稻籽粒发育的关系及烯效唑的调节[J]. 中国水稻科学, 1998, 12(2): 99-104. (  0) 0) |
 2023, Vol. 44
2023, Vol. 44


