植物固着生长在不断变化的环境中,不能通过移动来规避周围不良环境,需要根据环境灵活地调整自身生长状况进行应对。植物通过多种复杂的机制感知各种环境因素,并做出相应的反应,已经确定多种转录因子、转运蛋白等分子成分参与植物的胁迫响应[1-2]。在响应环境变化时,植物可以通过染色质结构的变化(包括DNA甲基化、组蛋白修饰、组蛋白变体、染色质重塑、非编码RNA等)来调控不同的生物学进程,从而适应外界环境的变化[3-4]。
H3K27me3是一种重要的组蛋白修饰,是一种抑制性的表观遗传标记,由Polycomb repressive complex 2(PRC2)复合物催化建立[5]。PRC2属于多梳家族蛋白(Polycomb group,PcG),PcG在表观遗传的调控方面起重要的作用,其最先在果蝇中被发现,参与许多重要的细胞活动和发育过程,具体包括干细胞多能性和可塑性、基因组印迹、X染色体失活等多种表观遗传过程[6-8]。在果蝇中PRC2的核心组分是一个含有SET结构域的甲基转移酶E[z](Enhancer of zeste),具有催化H3K27me3建立的活性[5]。
PRC2在果蝇、哺乳动物和植物中高度保守[9],拟南芥中存在和果蝇PRC2复合物核心亚基高度相似的蛋白,其中CURLY LEAF(CLF)、MEDEA (MEA)、SWINGER(SWN)是E[z]的同源蛋白[10-12]。在拟南芥生长发育过程中PRC2对调控基因的表达至关重要,涉及从种子萌发到胚形成的整个生命周期[13]。CLF和SWN主要在营养期起作用,而MEA主要在生殖期起作用[9, 13]。
拟南芥中CLF蛋白是PRC2蛋白复合物的重要组分,是H3K27me3甲基转移酶,CLF的突变会使H3K27me3的水平降低[14]。CLF影响植物叶片、花序、种子和根的发育,具体表现为:clf突变体叶片窄小而卷曲,叶片的细胞伸长长度和细胞数量显著减少[10, 15];萼片弯曲不足,不能包裹花蕾,导致早花表型,在短日照条件下开花时间比野生型早约3周,在长日照条件下开花时间比野生型早约2 d[10];种子更大,含油量更高[16-17];主根长度伸长,侧根的数目增加[18]。CLF影响拟南芥生长发育过程的多个方面,调控植物的多种性状。
温度是控制植物生长发育、分布和季节行为的关键因素,植物的整个生命周期都受到环境温度的影响,植物通过优化生长发育,不断适应自然季节温度的变化[19]。植物对温度非常敏感,甚至能感知到1 ℃的温差[20]。极端高温和低温都会对拟南芥的生长造成胁迫,导致生长缓慢、形态异常、种子产量降低[21]。在温和高温条件下,拟南芥表现出早花、下胚轴伸长、叶柄伸长、叶片变薄等性状[22];而在温和低温条件下,拟南芥表现出晚花、下胚轴变短等表型[20, 23]。本研究通过在不同温度条件(16、22 ℃)下,对野生型与clf-29突变体进行表型分析和差异表达基因分析,探索拟南芥表观遗传调控因子CLF在响应环境温度和温度形态建成中的作用。
1 材料与方法 1.1 供试材料与引物设计本研究使用的拟南芥植株为哥伦比亚(Col-0)生态型,其中clf-29(SALK_021003)为T-DNA插入突变体,鉴定突变体和RT-qPCR验证所需的引物见表1。
|
|
表 1 本研究用到的引物 Table 1 Primers used in this study |
拟南芥种子先用75%(φ)乙醇溶液消毒12 min,再用95%(φ)乙醇溶液继续消毒10 min后,在4 ℃冰箱避光放置2 d,然后在22或16 ℃长日照(16 h光∶8 h暗)条件下生长。22 ℃条件下,取用生长18 d的植株拍摄表型;16 ℃条件下,取用生长36 d的植株拍摄表型。
1.3 RNA-seq文库构建取0.1~0.2 g植物材料迅速用液氮预冷,于研钵中研磨成粉末后使用植物总RNA提取试剂盒(Tiangen Biotech,Beijing,China)进行RNA提取,每个样品3次重复。链特异性RNA-seq文库的构建参照KAPA stranded mRNA-seq kit(Illumina® Platforms,KR0960-v5.17)进行。构建好的文库进行琼脂糖凝胶电泳,选取250~500 bp的片段进行回收纯化。文库送到上海逆耳生物科技有限公司用Illumina Hiseq 6000测序仪测序,测序模式为双端150 bp。
1.4 RNA-seq数据分析使用cutadapt(v3.5)去除接头和测序质量低于Q20的碱基[24],再用HISAT2(v2.1.0)将筛选后的reads比对到拟南芥基因组(tair10)[25],通过SAMtools(v1.9)筛选出高比对质量的reads进行后续分析[26]。每个基因的reads数目通过featureCounts(v1.6.2)进行计算[27],并使用R包DESeq2(v1.34.0)以|log2(Fold change,FC)|≥log21.5和P<0.05的条件鉴定差异基因[28]。此外使用deeptools(v3.5.1)的bamCoverage工具将reads进行标准化处理生成bigwig文件[29],并使用IGV(v2.11.2)进行可视化[30]。分析基因的GO功能注释时使用了R包clusterProfiler(v4.2.2)[31]。
1.5 RT-qPCR取0.1~0.2 g植物材料迅速用液氮预冷,于研钵中研磨成粉末后使用TRIzol(Invitrogen,15596026)进行RNA提取,每个样品3次重复。利用反转录试剂盒(TaKaRa,RR047A)将提取的RNA反转录成cDNA,之后利用设计好的引物(表1)进行RT-qPCR,以ACTIN 2作为内参基因,根据3次生物学重复计算平均值和标准差。
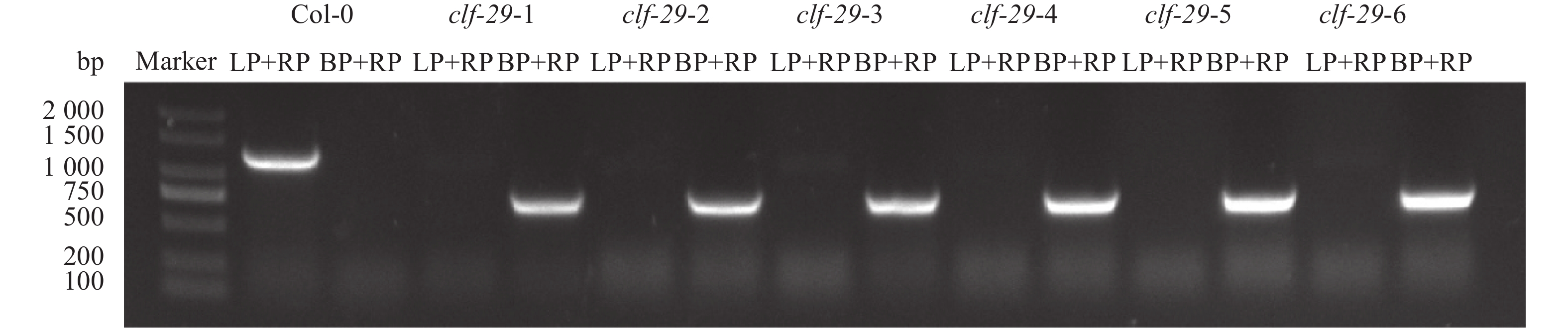
2 结果与分析 2.1 clf-29突变体在不同温度条件下的表型分析我们分别在22和16 ℃条件下种植拟南芥Col-0和clf-29突变体,使用三引物法对T-DNA插入材料进行鉴定,PCR结果显示,clf-29仅在T-DNA边界引物+右侧基因组引物(BP+RP)组合中才出现特异性条带,并且未出现Col-0中左侧基因组引物+右侧基因组引物(LP+RP)组合的特异性条带(图1),说明所用植物材料为纯合突变体。
|
图 1 Col-0和clf-29基因型鉴定 Fig. 1 Genotype identification of Col-0 and clf-29 LP:左侧基因组引物,RP:右侧基因组引物;BP:T-DNA边界引物 LP: Left genomic primer, RP: Right genomic primer, BP: T-DNA border primer |
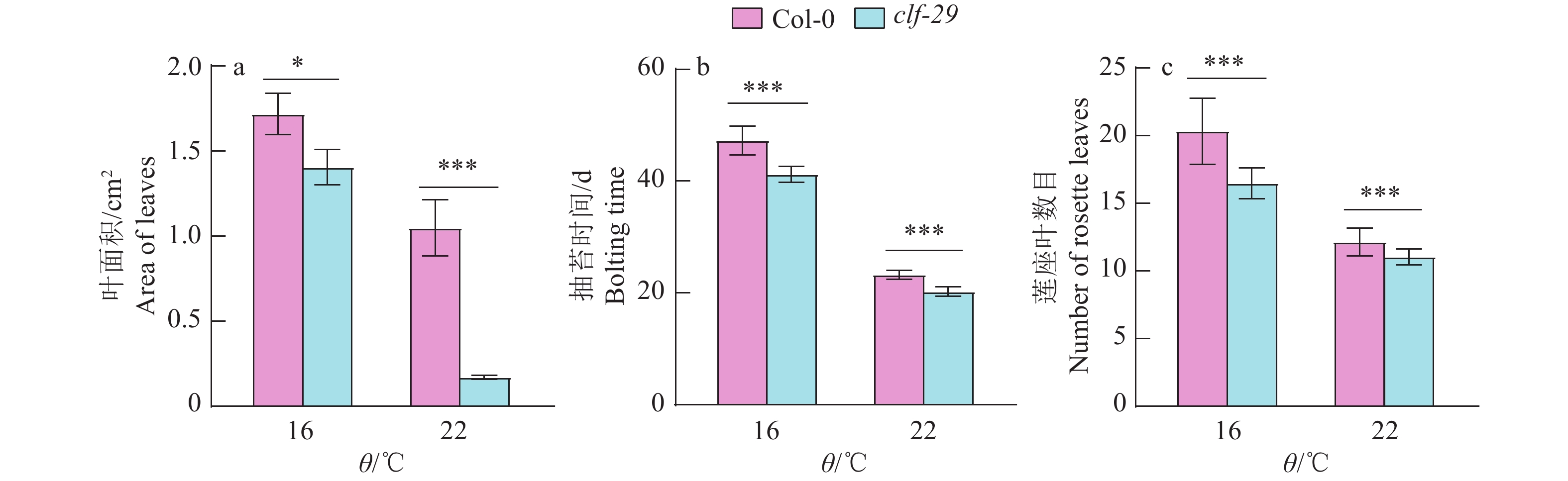
16 ℃条件下,clf-29突变体植株比Col-0稍小,但整体差异不大,叶片正常伸展,略小;而在22 ℃条件下,clf-29突变体与Col-0差异明显,植株更小,叶片卷曲而且显著减小(图2、3a)。无论在22 ℃还是16 ℃,相比Col-0,clf-29突变体都表现为抽苔时间提前和莲座叶数目减少,在16和22 ℃条件下,抽苔时间分别提前6和3 d,叶片数目分别减少4和1片(图3b、3c)。clf-29突变体在不同温度条件下的形态差异,说明CLF参与叶片形态、植株大小等发育进程,暗示CLF参与调控了拟南芥的温度形态建成。

|
图 2 Col-0和clf-29植株(a、b)和叶片(c、d)表型展示 Fig. 2 Phenotypic display of plant (a, b) and leaf (c, d) for Col-0 and clf-29 |
|
图 3 Col-0和clf-29在不同温度条件的表型统计 Fig. 3 Phenotypic statistics of Col-0 and clf-29 at different temperatures 图a统计的叶片数量为3片,图b、c统计的植株数量为25株;“*”和“***”分别表示Col-0和clf-29在P < 0.05和P < 0.001水平差异显著(t检验) The number of leaves counted in figure a is 3, the number of plants counted in figure b, c is 25; “*” and “***” indicate significant differences at P < 0.05 and P < 0.001 levels between Col-0 and clf-29 respectively (t test) |
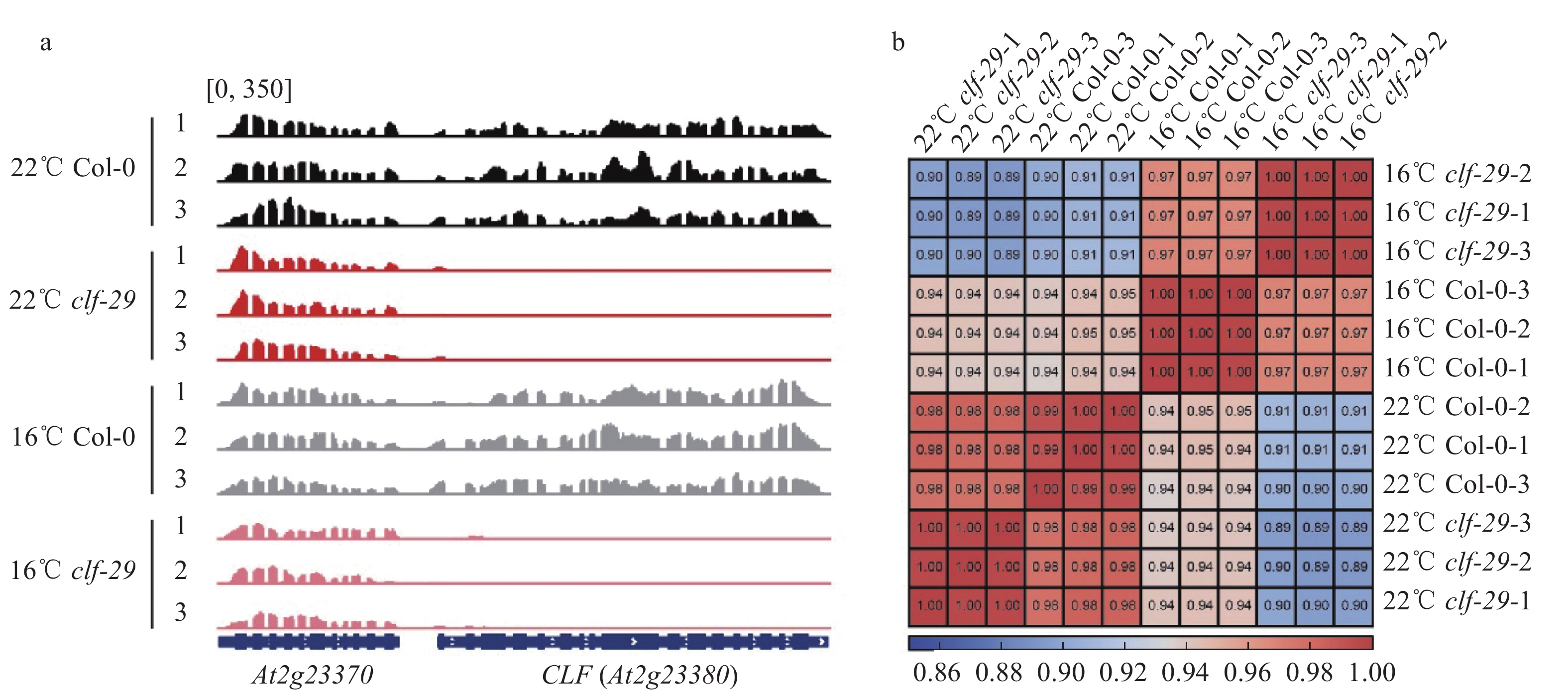
为了更深刻及全面地了解温度对clf-29突变体形态建成的调控机制,我们进行了高通量RNA-seq测序分析。使用在22 ℃培养箱生长18 d和在16 ℃培养箱生长36 d的整株植物材料进行取样、建库、测序,每个样品各有3次重复。筛选出碱基质量高的reads比对到拟南芥基因组,保留仅比对到染色质且比对质量较高的reads进行后续分析。结果显示,所有样品的原始Reads都在2 000万条以上,比对率都在97%以上,表明所建的RNA-seq文库的数据质量较高(表2)。为确定样品材料无误,查看了CLF基因的转录水平,在clf-29突变体中,CLF的表达量显著下降,而其邻近基因AT2G23370的表达量未受影响,说明试验材料是正确无误的(图4a)。此外转录组数据之间的相关性显示,每个样品的3次重复各自聚在一起,数据有良好的重复性(图4b)。
|
|
表 2 RNA-seq数据统计1) Table 2 Statistical analyses of RNA-seq data |
|
图 4 RNA-seq数据的准确性(a)和相关性分析(b) Fig. 4 Accuracy (a) and correlation analyses (b) of RNA-seq data |
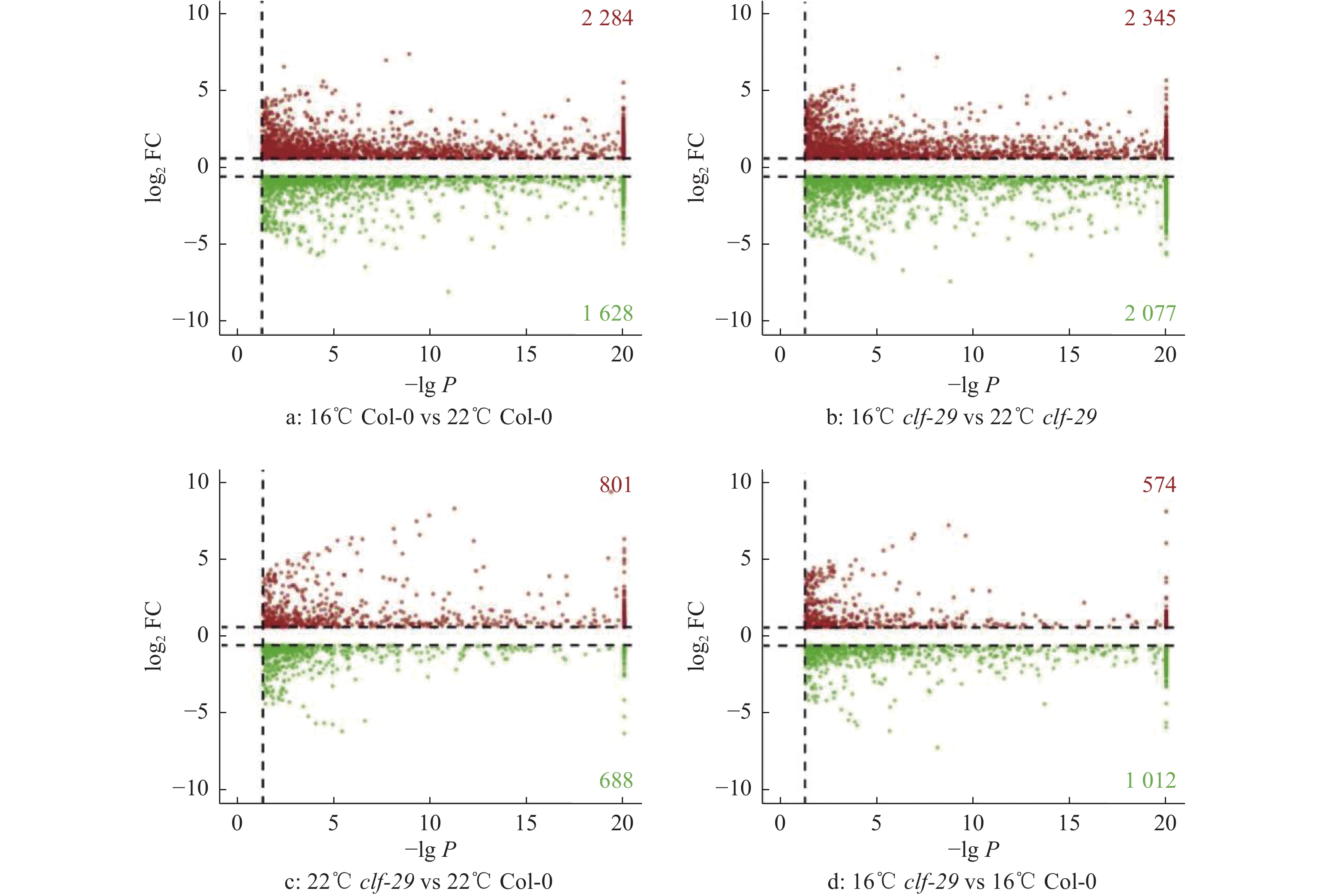
为了探究温度对CLF的影响,以|log2FC|≥log21.5和P<0.05为筛选条件,对转录组数据进行差异基因分析。我们鉴定到了Col-0、clf-29各自在16 ℃相较于22 ℃的差异基因,其中Col-0表达上调和下调的基因分别有2 284和1 628个,clf-29突变体分别是2 345和2 077个(图5a、5b),Col-0和clf-29在响应温度变化时改变的基因数目相近。我们还鉴定到了相比于Col-0,clf-29突变体分别在22和16 ℃的差异基因,22 ℃条件下clf-29突变体相比野生型上调801个基因、下调688个基因,而在16 ℃条件下clf-29突变体相比野生型上调574个基因、下调1 012个基因(图5c、5d),说明CLF的缺失会导致大量基因异常表达。
|
图 5 不同温度条件下Col-0和clf-29的差异表达基因 Fig. 5 Differentially expressed genes of Col-0 and clf-29 at different temperatures |
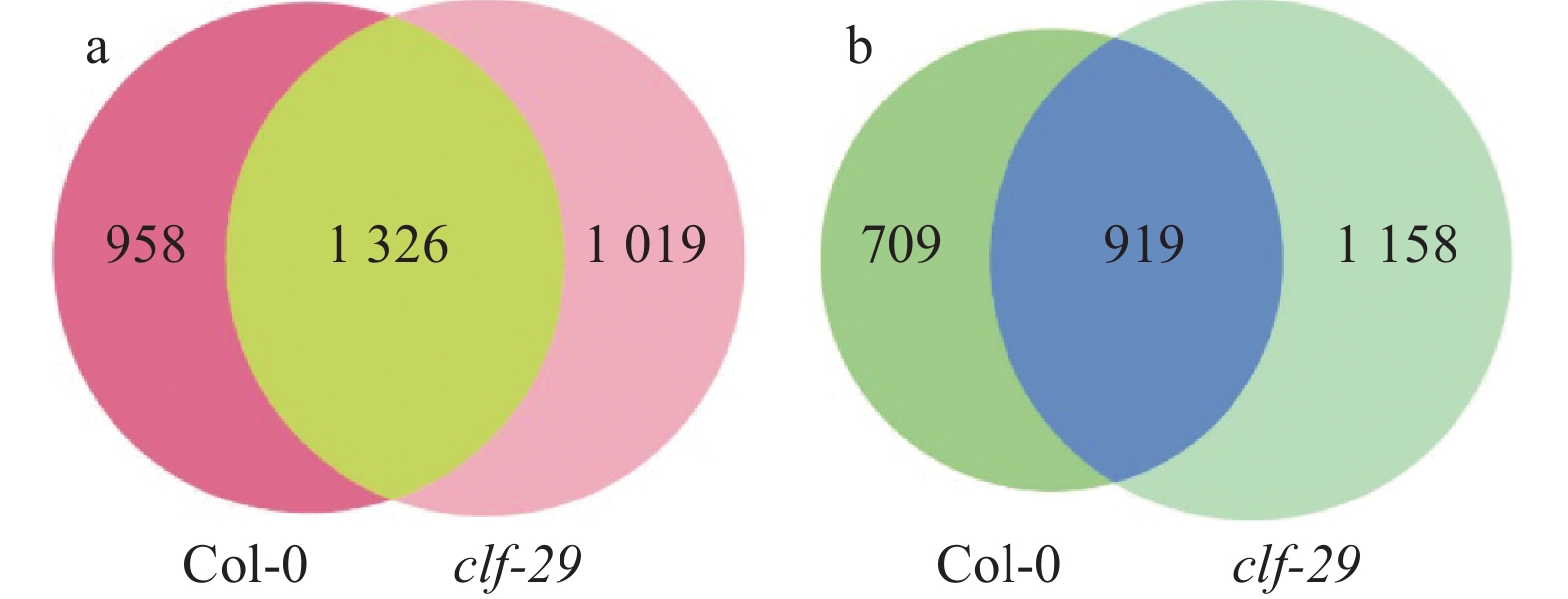
将Col-0在16、22 ℃的差异基因和clf-29在16、22 ℃的差异基因做交集。结果发现,对于上调基因,两者的交集是1 326个基因,仅在Col-0的是958个基因,仅在clf-29的是1 019个基因;对于下调基因,两者的交集是919个基因,仅在Col-0的是709个基因,仅在clf-29的是1 158个基因(图6a、6b)。在不同温度条件下,Col-0和clf-29有很多基因是共同改变的(图7),而那些仅在Col-0或者仅在clf-29变化的基因更为重要。我们对这些基因进行GO富集分析,发现仅在Col-0上调的基因更多地富集在环境响应、物质转运等途径(图8a);仅在clf-29上调的基因主要富集在响应刺激、激素运输、细胞壁生长等途径(图8b);仅在Col-0下调的基因富集在物质合成、物质代谢等途径(图8c);仅在clf-29下调的基因富集在刺激响应、合成代谢调控等途径(图8d)。
|
图 6 相比于22 ℃,16 ℃条件下Col-0和clf-29的差异上调(a)和差异下调(b)基因的交集 Fig. 6 Intersection of differentially up-regulated (a) and down-regualted (b) genes at 16 ℃ compared to 22 ℃ |

|
图 7 相比于22 ℃,16 ℃条件下Col-0和clf-29差异表达基因的相关性 Fig. 7 Correlation of differentially expressed genes between Col-0 and clf-29 at 16 ℃ compared to 22 ℃ |

|
图 8 相比于22 ℃,16 ℃条件下Col-0和clf-29各自差异表达基因的GO分析 Fig. 8 GO analyses of differentially expressed genes in Col-0 and clf-29 respectively at 16 ℃ compared to 22 ℃ |
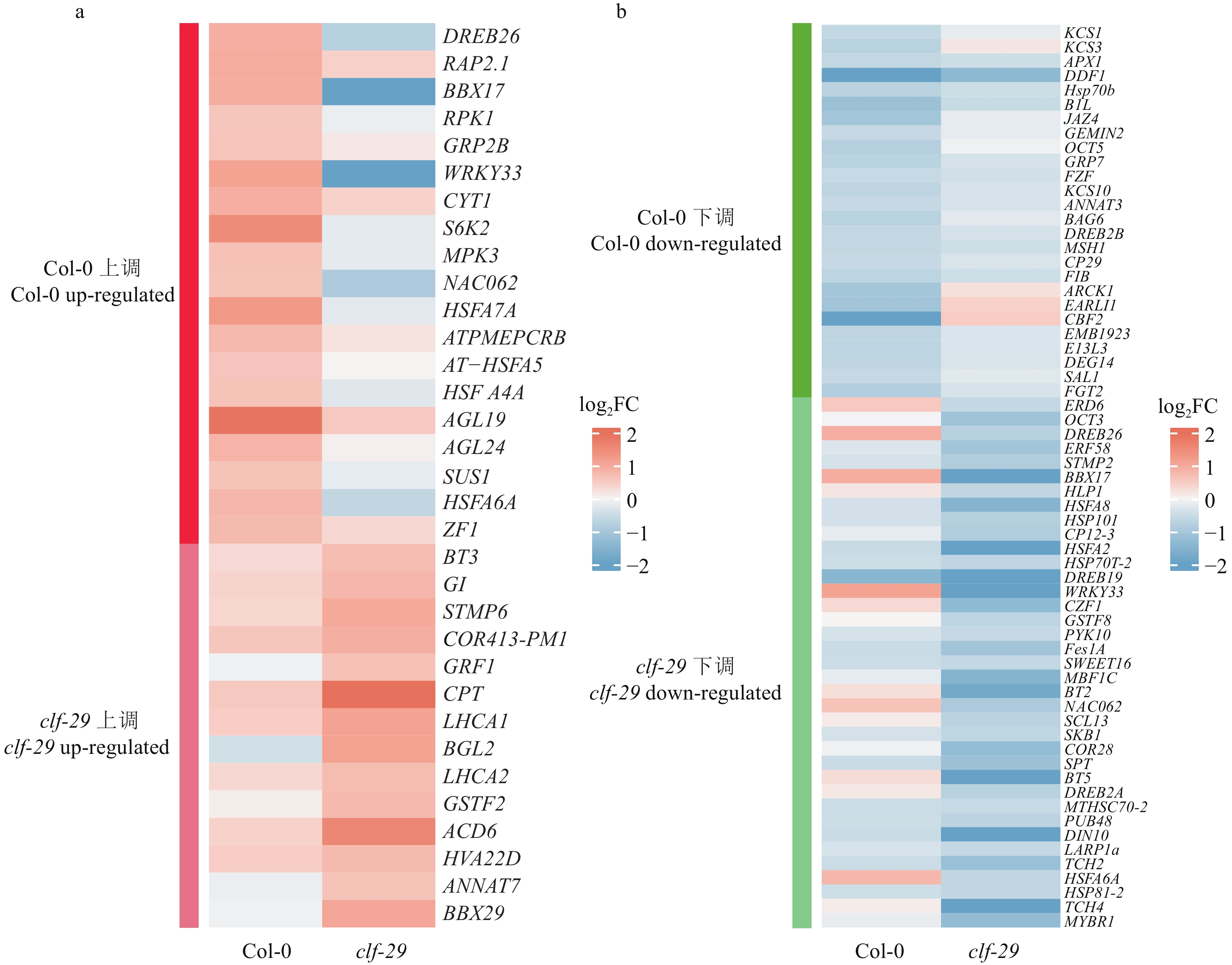
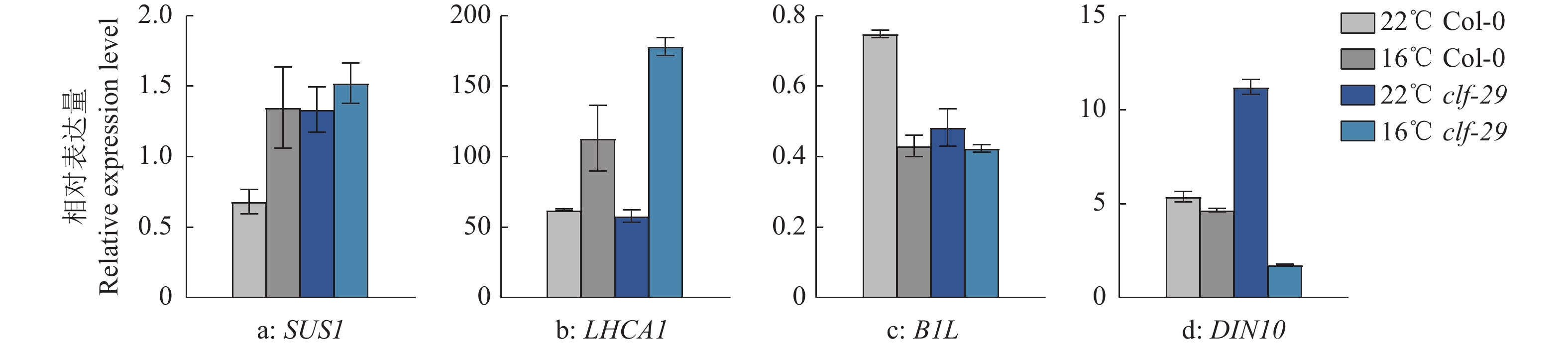
温度的变化造成了clf-29突变体的不同表型,因此我们找到了上述4类差异表达基因(图6),相比于22 ℃,在16 ℃条件下在Col-0显著上调而在clf-29中未显著上调的,称之为Col-0上调,同样得到了clf-29上调、Col-0下调、clf-29下调,通过GO注释在这4类基因中寻找与温度响应相关的基因进行分析。有19个基因仅在Col-0上调表达,有14个基因仅在clf-29上调表达(图9a);仅在Col-0下调的是26个基因,仅在clf-29下调的是37个基因(图9b)。在这4类基因中各选取特定基因SUCROSE SYNTHASE 1(SUS1)、PHOTOSYSTEM Ⅰ LIGHT HARVESTING COMPLEX GENE 1(LHCA1)、BYPASS1-LIKE(B1L)、DARK INDUCIBLE 10 (DIN10)进行RT-qPCR验证(材料情况与构建RNA-seq文库一致),相比于clf-29,SUS1在Col-0上调更加显著,与之类似,LHCA1在clf-29上调更加显著,B1L在Col-0、DIN10在clf-29的下调程度更高(图10),这与热图结果基本一致。Col-0材料在12 ℃处理1、3、5、10 h后LHCA1表达量上升,处理24 h后表达量下降[32],我们的结果与短时间处理一致;Col-0材料在4 ℃处理1、3、6、12 h后B1L表达量上升[33],处理24 h后表达量下降,我们的结果与处理24 h一致。我们的结果与其他已发表文章结果的差异,可能是处理温度与处理时间都存在差别而共同导致的。综上,CLF的缺失造成了大量温度响应基因表达改变,造成一系列的表型差异,证明了CLF在不同温度条件下调控生长发育的重要作用。
|
图 9 相比于22 ℃,16 ℃条件下Col-0和clf-29各自温度响应基因的热图 Fig. 9 Heatmap of temperature-responsive genes in Col-0 and clf-29 respectively at 16 ℃ compared to 22 ℃ |
|
图 10 特定温度响应基因的RT-qPCR验证 Fig. 10 RT-qPCR verifiction of specific temperature-responsive genes |
拟南芥中的CLF是PRC2复合物的重要亚基,是H3K27me3甲基转移酶,CLF蛋白结构保守,在多种植物如水稻、番茄中都报道过CLF的同源蛋白[34-35]。CLF调控植物生长发育的多个阶段,在拟南芥中鉴定到6 854个H3K27me3富集的基因,这些基因都是潜在的CLF可以调控的靶基因,涉及多种生长发育、响应刺激相关的通路,暗示CLF对植物的正常生长起至关重要的作用[14]。本研究发现不同温度条件下clf-29突变体的形态建成存在差异,并且找到了多类可能导致表型差异的重要基因,其中仅在Col-0响应而在clf-29突变体未响应的基因说明CLF的缺失使突变体无法正常发挥功能,仅在clf-29响应的差异基因说明缺失CLF使其产生了更多的变化,并找到了多个和温度相关的基因,其发生明显的表达变化,这些结果说明CLF是参与拟南芥温度形态建成的重要表观调控因子。
不同温度条件下clf-29突变体的形态差异,极有可能是因为在不同温度下CLF介导的H3K27甲基化在基因组上的不同丰度造成的。温度是一种重要的环境影响因子,低温会影响拟南芥H3K27me3的富集。在低温条件(4 ℃)下,低温响应基因COR15A和ATGOLS3的H3K27me3水平会降低从而导致基因表达的变化[36-37]。温度变化也可以通过改变开花抑制基因FLOWERING LOCUS C(FLC)的H3K27me3水平来调控FLC的活性,从而调控植物开花时间[38-39]。然而温度如何影响甲基转移酶CLF介导的H3K27甲基化的分布,是需要进一步探索的问题。
除了H3K27甲基化以外,其他的表观遗传标记也参与植物对环境温度的适应性调控,当植物生长环境的温度较低时,组蛋白变体H2A.Z会富集在DNA上,抑制基因的表达,当温度升高时,H2A.Z富集程度降低,使基因正常表达[20]。因此,在不同温度条件下,H3K27me3与其他的表观遗传标记会产生怎样的偶联反应也是值得深思和深入探究的问题。
| [1] |
CHANG Y N, ZHU C, JIANG J, et al. Epigenetic regulation in plant abiotic stress responses[J]. Journal of Integrative Plant Biology, 2020, 62(5): 563-580. DOI:10.1111/jipb.12901 (  0) 0) |
| [2] |
LAMERS J, VAN DER MEER T, TESTERINK C. How plants sense and respond to stressful environments[J]. Plant Physiology, 2020, 182(4): 1624-1635. DOI:10.1104/pp.19.01464 (  0) 0) |
| [3] |
ASHAPKIN V V, KUTUEVA L I, ALEKSANDRUSHKINA N I, et al. Epigenetic mechanisms of plant adaptation to biotic and abiotic stresses[J]. International Journal of Molecular Sciences, 2020, 21(20): 7457. DOI:10.3390/ijms21207457 (  0) 0) |
| [4] |
GALLUSCI P, DAI Z, GENARD M, et al. Epigenetics for plant improvement: Current knowledge and modeling avenues[J]. Trends in Plant Science, 2017, 22(7): 610-623. DOI:10.1016/j.tplants.2017.04.009 (  0) 0) |
| [5] |
KASSIS J A, KENNISON J A, TAMKUN J W. Polycomb and trithorax group genes in Drosophila
[J]. Genetics, 2017, 206(4): 1699-1725. DOI:10.1534/genetics.115.185116 (  0) 0) |
| [6] |
LEWIS E B. A gene complex controlling segmentation in Drosophila
[J]. Nature, 1978, 276(5688): 565-570. DOI:10.1038/276565a0 (  0) 0) |
| [7] |
SCHUETTENGRUBER B, GANAPATHI M, LEBLANC B, et al. Functional anatomy of Polycomb and trithorax chromatin landscapes in Drosophila embryos
[J]. PLoS Biology, 2009, 7(1): 146-163. (  0) 0) |
| [8] |
SPARMANN A, VAN LOHUIZEN M. Polycomb silencers control cell fate, development and cancer[J]. Nature Reviews Cancer, 2006, 6(11): 846-856. DOI:10.1038/nrc1991 (  0) 0) |
| [9] |
PIEN S, GROSSNIKLAUS U. Polycomb group and trithorax group proteins in Arabidopsis
[J]. Biochimica et Biophysica Acta: Gene Structure and Expression, 2007, 1769(5/6): 375-382. (  0) 0) |
| [10] |
GOODRICH J, PUANGSOMLEE P, MARTIN M, et al. A Polycomb-group gene regulates homeotic gene expression in Arabidopsis
[J]. Nature, 1997, 386(6620): 44-51. DOI:10.1038/386044a0 (  0) 0) |
| [11] |
GROSSNIKLAUS U, VIELLE-CALZADA J P, HOEPPNER M A, et al. Maternal control of embryogenesis by MEDEA, a Polycomb group gene in Arabidopsis
[J]. Science, 1998, 280(5362): 446-450. DOI:10.1126/science.280.5362.446 (  0) 0) |
| [12] |
CHANVIVATTANA Y, BISHOPP A, SCHUBERT D, et al. Interaction of Polycomb-group proteins controlling flowering in Arabidopsis
[J]. Development, 2004, 131(21): 5263-5276. DOI:10.1242/dev.01400 (  0) 0) |
| [13] |
XIAO J, WAGNER D. Polycomb repression in the regulation of growth and development in Arabidopsis
[J]. Current Opinion in Plant Biology, 2015, 23: 15-24. DOI:10.1016/j.pbi.2014.10.003 (  0) 0) |
| [14] |
SHU J, CHEN C, THAPA R K, et al. Genome-wide occupancy of histone H3K27 methyltransferases CURLY LEAF and SWINGER in Arabidopsis seedlings
[J]. Plant Direct, 2019, 3(1): 100. DOI:10.1002/pld3.100 (  0) 0) |
| [15] |
KIM G T, TSUKAYA H, UCHIMIYA H. The CURLY LEAF gene controls both division and elongation of cells during the expansion of the leaf blade in Arabidopsis thaliana
[J]. Planta, 1998, 206(2): 175-183. DOI:10.1007/s004250050389 (  0) 0) |
| [16] |
LAFOS M, KROLL P, HOHENSTATT M L, et al. Dynamic regulation of H3K27 trimethylation during Arabidopsis differentiation
[J]. PLoS Genetics, 2011, 7(4): e1002040. DOI:10.1371/journal.pgen.1002040 (  0) 0) |
| [17] |
LIU J, DENG S, WANG H, et al. CURLY LEAF regulates gene sets coordinating seed size and lipid biosynthesis
[J]. Plant Physiology, 2016, 171(1): 424-436. DOI:10.1104/pp.15.01335 (  0) 0) |
| [18] |
GU X, XU T, HE Y. A histone H3 lysine-27 methyltransferase complex represses lateral root formation in Arabidopsis thaliana
[J]. Molecular Plant, 2014, 7(6): 977-988. DOI:10.1093/mp/ssu035 (  0) 0) |
| [19] |
DING Y, SHI Y, YANG S. Molecular regulation of plant responses to environmental temperatures[J]. Molecular Plant, 2020, 13(4): 544-564. DOI:10.1016/j.molp.2020.02.004 (  0) 0) |
| [20] |
KUMAR S V, WIGGE P A. H2A.Z-containing nucleosomes mediate the thermosensory response in Arabidopsis
[J]. Cell, 2010, 140(1): 136-147. DOI:10.1016/j.cell.2009.11.006 (  0) 0) |
| [21] |
GIL K E, PARK C M. Thermal adaptation and plasticity of the plant circadian clock[J]. New Phytologist, 2019, 221(3): 1215-1229. DOI:10.1111/nph.15518 (  0) 0) |
| [22] |
QUINT M, DELKER C, FRANKLIN K A, et al. Molecular and genetic control of plant thermomorphogenesis[J]. Nature Plants, 2016, 2(1): 15190. DOI:10.1038/NPLANTS.2015.190 (  0) 0) |
| [23] |
BLAZQUEZ M A, AHN J H, WEIGEL D. A thermosensory pathway controlling flowering time in Arabidopsis thaliana
[J]. Nature Genetics, 2003, 33(2): 168-171. DOI:10.1038/ng1085 (  0) 0) |
| [24] |
MARTIN M. Cutadapt removes adapter sequences from high-throughput sequencing reads[J]. EMBnet Journal, 2011, 17(1): 10-12. DOI:10.14806/ej.17.1.200 (  0) 0) |
| [25] |
KIM D, LANGMEAD B, SALZBERG S L. HISAT: A fast spliced aligner with low memory requirements[J]. Nature Methods, 2015, 12(4): 357-360. DOI:10.1038/nmeth.3317 (  0) 0) |
| [26] |
LI H, HANDSAKER B, WYSOKER A, et al. The sequence alignment/map format and SAMtools[J]. Bioinformatics, 2009, 25(16): 2078-2079. DOI:10.1093/bioinformatics/btp352 (  0) 0) |
| [27] |
LIAO Y, SMYTH G K, SHI W. FeatureCounts: An efficient general purpose program for assigning sequence reads to genomic features[J]. Bioinformatics, 2014, 30(7): 923-930. DOI:10.1093/bioinformatics/btt656 (  0) 0) |
| [28] |
LOVE M I, HUBER W, ANDERS S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2[J]. Genome Biology, 2014, 15(12): 550. DOI:10.1186/s13059-014-0550-8 (  0) 0) |
| [29] |
RAMIREZ F, RYAN D P, GRUNING B, et al. DeepTools2: A next generation web server for deep-sequencing data analysis[J]. Nucleic Acids Research, 2016, 44(W1): W160-W165. DOI:10.1093/nar/gkw257 (  0) 0) |
| [30] |
THORVALDSDOTTIR H, ROBINSON J T, MESIROV J P. Integrative Genomics Viewer (IGV): High-performance genomics data visualization and exploration[J]. Briefings in Bioinformatics, 2013, 14(2): 178-192. DOI:10.1093/bib/bbs017 (  0) 0) |
| [31] |
YU G, WANG L G, HAN Y, et al. ClusterProfiler: An R package for comparing biological themes among gene clusters[J]. OMICS: A Journal of Integrative Biology, 2012, 16(5): 284-287. DOI:10.1089/omi.2011.0118 (  0) 0) |
| [32] |
JUNG C G, HWANG S G, PARK Y C, et al. Molecular characterization of the cold- and heat-induced Arabidopsis PXL1 gene and its potential role in transduction pathways under temperature fluctuations
[J]. Journal of Plant Physiology, 2015, 176: 138-146. DOI:10.1016/j.jplph.2015.01.001 (  0) 0) |
| [33] |
CHEN T, CHEN J H, ZHANG W, et al. BYPASS1-LIKE, a DUF793 family protein, participates in freezing tolerance via the CBF pathway in Arabidopsis
[J]. Frontiers in Plant Science, 2019, 10: 807. DOI:10.3389/fpls.2019.00807 (  0) 0) |
| [34] |
BOUREAU L, HOW-KIT A, TEYSSIER E, et al. A CURLY LEAF homologue controls both vegetative and reproductive development of tomato plants[J]. Plant Molecular Biology, 2016, 90(4/5): 485-501. (  0) 0) |
| [35] |
LUO M, PLATTEN D, CHAUDHURY A, et al. Expression, imprinting, and evolution of rice homologs of the polycomb group genes[J]. Molecular Plant, 2009, 2(4): 711-723. DOI:10.1093/mp/ssp036 (  0) 0) |
| [36] |
KWON C S, LEE D, CHOI G, et al. Histone occupancy-dependent and -independent removal of H3K27 trimethylation at cold-responsive genes in Arabidopsis[J]. Plant Journal, 2009, 60(1): 112-121. DOI:10.1111/j.1365-313X.2009.03938.x (  0) 0) |
| [37] |
RAMAKRISHNAN M, ZHANG Z, MULLASSERI S, et al. Epigenetic stress memory: A new approach to study cold and heat stress responses in plants[J]. Frontiers in Plant Science, 2022, 13: 1075279. DOI:10.3389/fpls.2022.1075279 (  0) 0) |
| [38] |
TIAN Y, ZHENG H, ZHANG F, et al. PRC2 recruitment and H3K27me3 deposition at FLC require FCA binding of COOLAIR
[J]. Science Advances, 2019, 5(4): eaau7246. DOI:10.1126/sciadv.aau7246 (  0) 0) |
| [39] |
YANG H, BERRY S, OLSSON T S G, et al. Distinct phases of Polycomb silencing to hold epigenetic memory of cold in Arabidopsis
[J]. Science, 2017, 357(6356): 1142-1145. DOI:10.1126/science.aan1121 (  0) 0) |
 2023, Vol. 44
2023, Vol. 44


